Практикум 8
Задание1.Использование разных вариантов BLAST.
В качестве контига для работы я выбрал KQ971639.1 (Рис1) ,длинной 29226 bp.Это контиг принадлежащий орагнизму Tribolium castaneum.
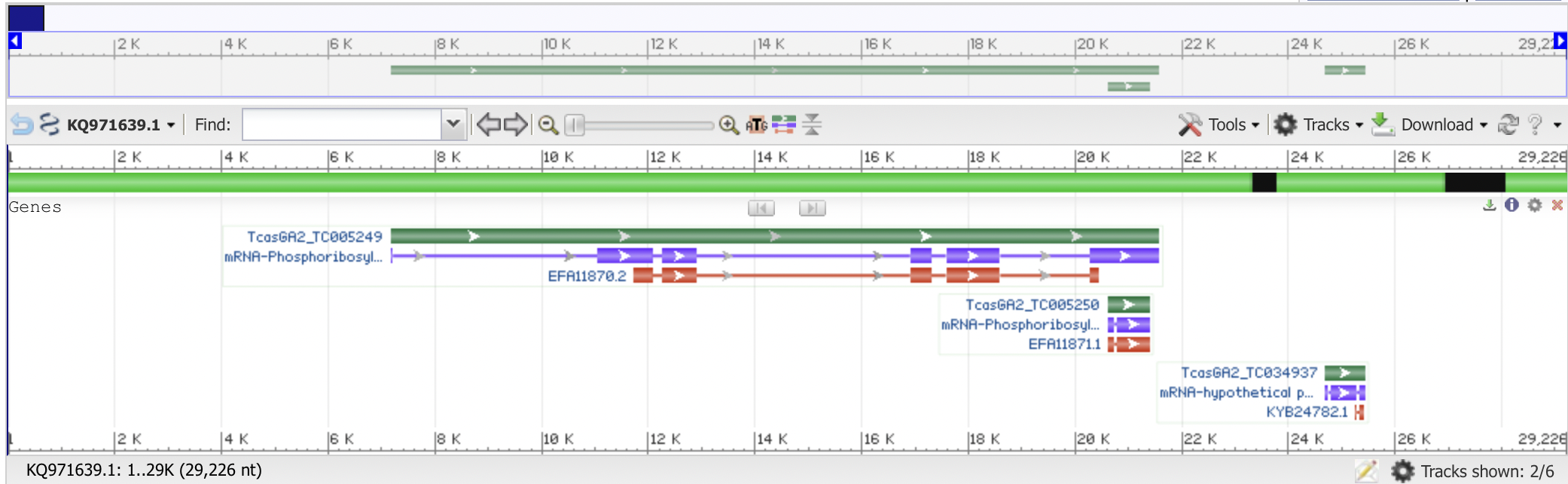
На графике выше можно увидеть, что в данном контиге 3 гена :
1)Gene: TcasGA2_TC005249 ; Location: 7,176..21,588; ;Length: 14,413 nt
2)Gene: TcasGA2_TC005250 ; Location: 20,606..21,401; Length: 796 nt;
3)Gene: TcasGA2_TC034937; Location: 24,696..25,442; Length: 747 nt;
В дальнейшем для работы я буду использовать фрагмент из этого контига, соответствующий 1 гену :
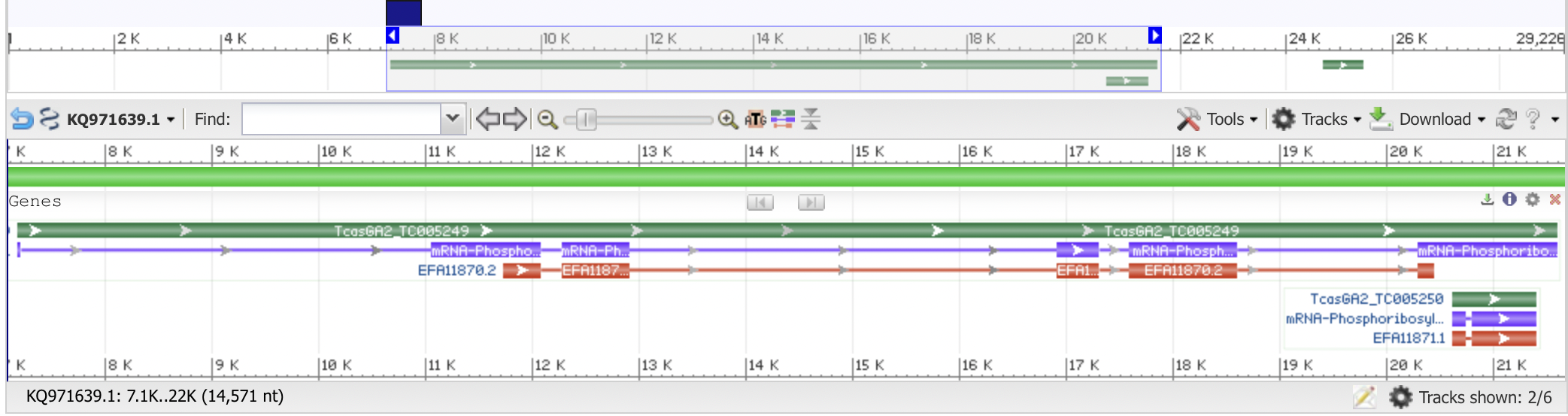
Выше (Рис 2) представлен приближенный график участка за который я берусь. Здесь зеленый - ген(в теории светло зеленый - нетраслируемая область, но у меня ее нет) ,фиолетовый- мРНК, красный - кодирующая последовательность.
Для BLAST поиска я исключил всех представителей класса insects (taxid:50557). Максимальное число находок- 5000, E-value- 0.05, длина слова- 11. В результате Blastn запроса я получил 14 находок, среди которых оказались части генома, принадлежащие пауку мета бурнети, моллюску пателла, аризонскому древесному скорпиону и др. .
Megablast запрос был выполнен с параметрами: Длинна слова -25 , E-value - 0.056 максимально число находок - 5000.В результате Megablast запроса я ничего не получил:( Наверно, это ожидаемо,учитывая , что я убрал всех ближайших родственников. Если провести megablast без ограничения, то будет 41 находка среди которых есть жуки из того же семейства, что и Tribolium castaneum.
В результате blastx запроса я получил 428 находок среди которых оказались белки принадлежщие ранее упомянутому скорпиону, молюску и др. .Запрос был выполнен при тех же параметрах, только длинна слова была изменена на 6.
При запуске tblastx выдало ошибку :(
Существуют разные алгоритмы blast, использующиеся для решения разных задач :
blastn используется для поиска гомологов не белок кодирующих последовательностей (из-за вырожденности генетического кода и алгоритмов Blast белок кодирующие не найдутся; по крайней мере это маловероятно до длины слова 4) .
Megablast оп сути является blastn, но имеет более жесткие параметры поиска. Он применяется для поиска очень похожих нуклеотидных последовательностей. Его можно использовать, например, при поиске расположения гена в полностью аннотированном геноме.
Blastx -алгоритм, принмающий нуклеотидную последовательность, транслирующий ее и ищущий гомологи в белковой базе данных. Может пригодиться при поиске кодируемых белков при наличии собранного транскритома.
tBlastx получает нуклеотидную последовательность, транслирует ее и и ищет схожие в нуклеотидной базе данных, транслированной в шести рамках считывания. Может, например, пригодиться, если мы знаем, что наша последовательность белок кодирующая и мы не смогли найти ее гомологов в белковой базе данных. Мы можем преположить, что эта белок кодирующая последовательность была найдена у какого-то другого организма, но при этом не была аннотирована. Тогда мы сможем найти гомологов нашей последовательности в транслированной нуклеотидной базе данных с помощью tblastx.
Задание2.Поиск в геноме эукариот генов основных рибосомальных РНК по далекому гомологу
Сначала я проиндексировал геном своего оганизма командой :
makeblastdb -in GCF_000146045.2_R64_genomic.fna -dbtype nucl
Далее был проведен локальной поиск BLAST :
blastn -task blastn -query 16s_eischerichia.fasta -db GCA_000002335.3_Tcas5.2_genomic.fna -evalue 0.000001 -out blast1.out -outfmt 7
blastn -task blastn -query 23s_eischerichia.fasta -db GCA_000002335.3_Tcas5.2_genomic.fna -evalue 0.000001 -out blast2.out -outfmt 7
Файлы 16s_eischerichia.fasta b 23s_eischerichia.fasta содержат в себе последовательности 16S и 23S рРНК, соответственно .16S и 23S рРНК входят в состав малой и большой субъединиц рибосомы, учавствуя в трансляции.
Вывод первого кода
Вывод второго кода
Был использован blastn, т.к входные данные - днк и я ищу не белок кодирующие последовательности (соответсвенно ищу по нуклеотидной базе данных).Зaпрос был выполнен при стандартных параметрах, только evalue был изменен на 0.00001. Было получено 12 и 5 находок соответственно. Я не нашел ни одного гомолога рРНК. В теории должно быть что-то найдено в митохондриальной ДНК, но BLAST и здесь ничего не нашел. Вручную мной были проверены все находки выданный BLAST и все они совпадают с участками аннотированных CDS белка. Вероятно, это означает, что геном выбранного эукариота плохо аннотирован/собран. Хотя он является референсным и в принципе по своим параметрам (длине и числе генов) соответсвует ожидаемым для жука.