В рамках практикума я буду работать с белком LNRN_BACSU.В Uniprot была получена последовательность выбранного белка и его структура
Задание 1. Знакомство с базой данных OPM
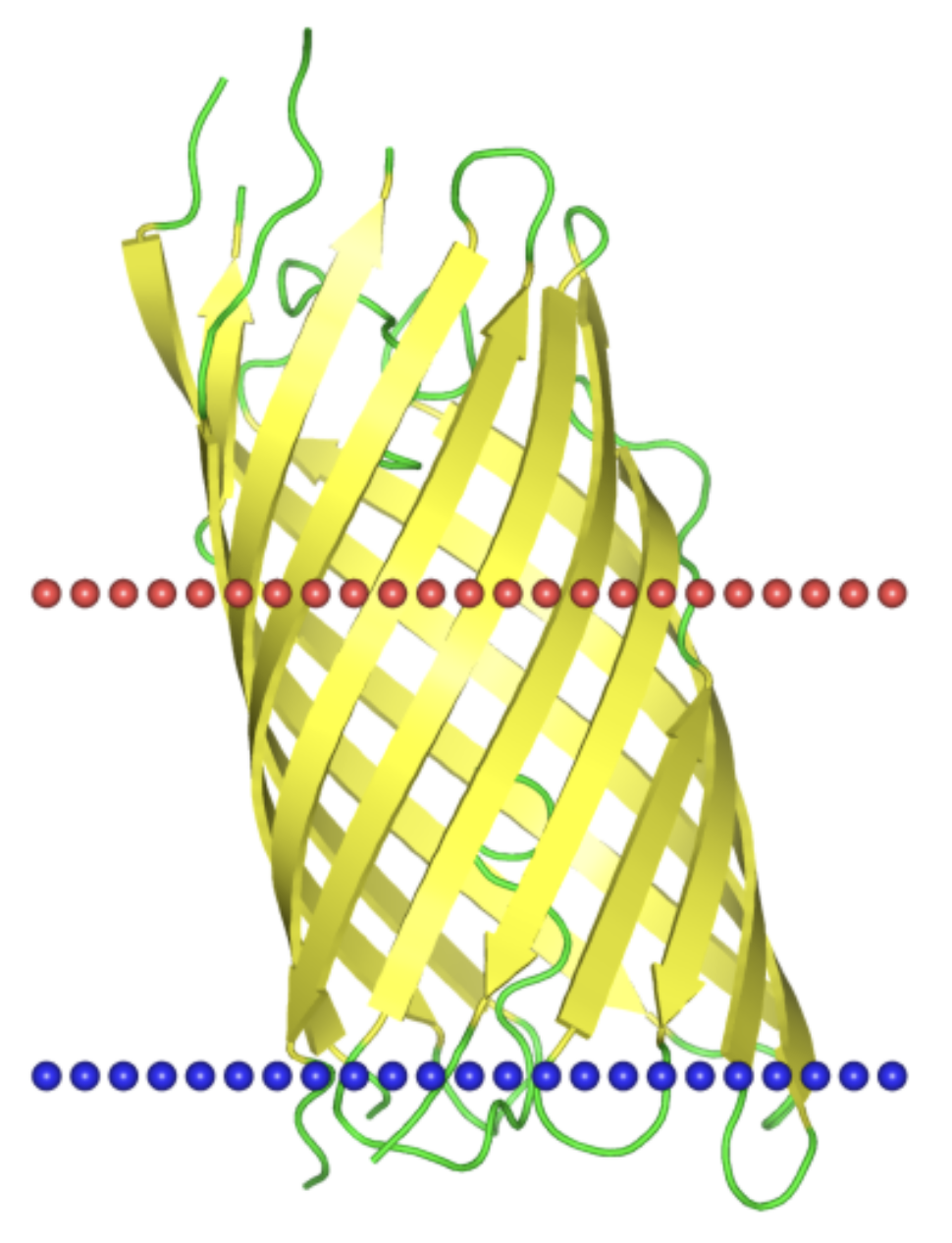
В базе OPM был выбран белок, являющийся внеклеточной сериновой протеазой Escherichia coli- Esp P. Функциональный анализ показал, что EspP способна расщеплять пепсин А и фактор свертывания крови V человека. Белок локализирован во внешней мембране бактерии, а погруженная в мембрану часть структуры представляет собой бета-бочонок.
| Идентфикатор Uniprot | ESPP_ECO57 |
|---|---|
| Толщина трансмембранной части белка: | 25.1 Å |
| Координаты трансмембранных участков: | 1(1039-1047),2(1062-1069),3(1078-1086),4(1103-1113),5(1117-1125),6(1149-1161),7(1165-1175),8(1204-1214),9(1220-1230),10(1259-1268),11(1272-1279),12(1291-1300) |
| Количество остатков а/к в одном тяже β-листа: | 9 |
| Число трансмембранных структур | 12 |
| Локализация | Внешняя мембрана грамм-отрицательной бактерии |
Задание 2.DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
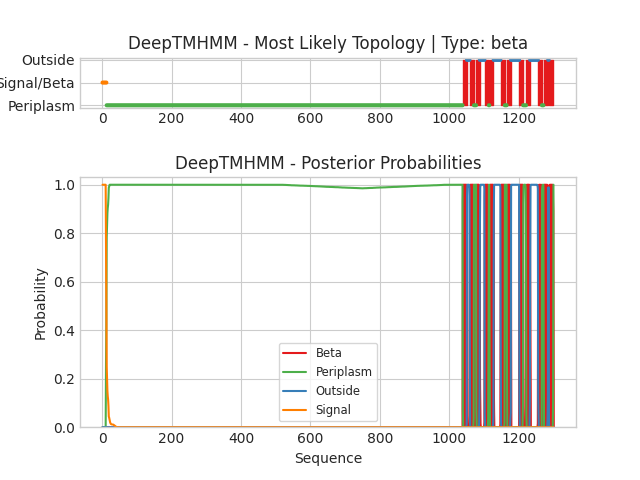
Для анализа программой DeepTMHMM были использованы 2 ранее упомянутых белков: LNRN_BACSU (Рис 1) и ESPP_ECO57 (Рис 2). На верхней картинке изображено расположение участков белка в мембране. Красным выделены трансмембранные участки, розовым - внутриклеточные, синим - внеклеточные, оранжевым отмечен сигнал локализации, зеленым - участок периплазмы . На нижнем рисунке для каждого остатка белка показаны вероятности вхождения в одну из этих категорий.Выходит, по оси X - номер аминокислотного остатка, по оси Y - вероятность принадлежности к категории.
Текстовая выдача для LNRN_BACSU и для ESPP_ECO57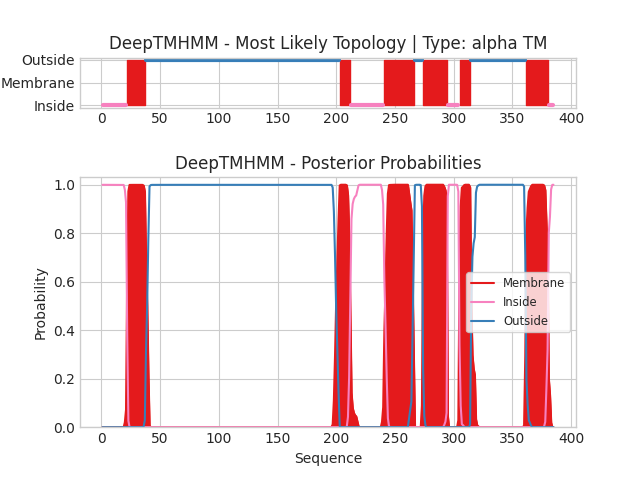
Мы видим, что все 6 трасмембарнных участков LNRN_BACSU удалось предсказать
Для ESPP_ECO57 было предсказано 12 трансмембранных участков. Значительная часть белка находится в периплазме.
Задание 3. PPM: Предсказание положения выданного белка в мембране
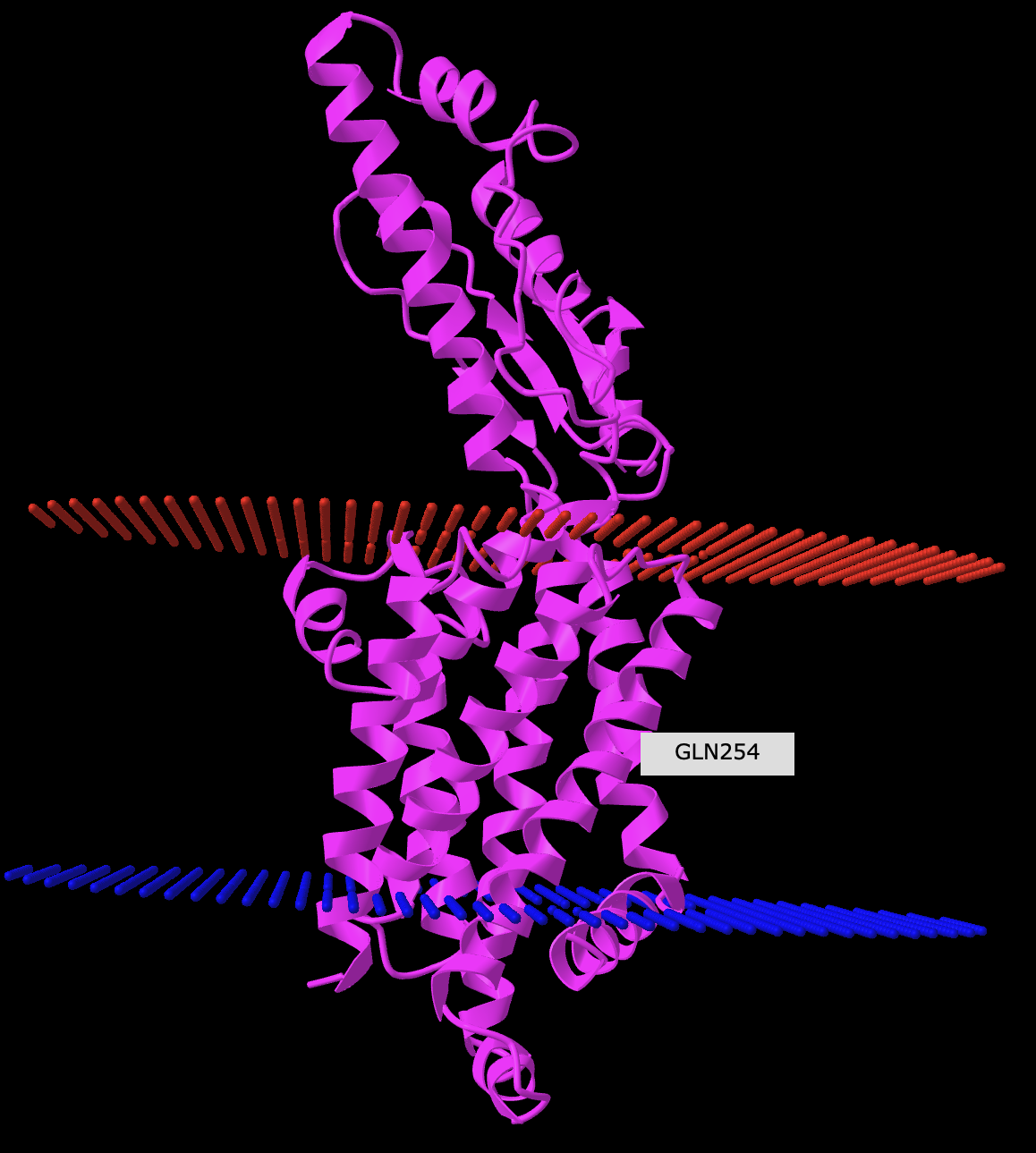
Используя сервис PPM было получено предсказание положения белка LNRN_BACSU в мембране.
| Type of membrane. | Gram-positive bacteria inner membrane |
|---|---|
| Allow curvature. | no |
| Topology (N-ter). | in |
Тип мембраны был выбран таковым, т.к. белок принадлежит грамположительной бактерии B. Subtilis
Задание 4. Сравнение алгоритмов предсказания трансмембранных спиралей.
Сравним результаты использованных программ и баз данных.
Результаты DeepTMHMM и PPM для белка LNRN_BACSU не совпали. DeepTMHMM предсказал наличие 6 мембранных участков (в Uniprot тоже 6 участков),а PPM предсказал 7, он добавил трансмембарнный участок 6( 328- 341). Это участок, который в легенде предсказания AlphaFold считается надежным.Остальные участки имеют схожик координаты, но отличаются в пределах нескольких аминокислот. В целом, структура предсказанная AlphaFold имеет довольну высокую оценку качества предсказания для большей части белка. Плохо предсказан лишь участок (169-191), который DeepTMHMM был предсказан, как участок за пределами мембраны, поэтому мне кажется, что достоверность модели AlphaFold не повлияло на работу PPM
Что касается ESPP_ECO57: DeepTMHMM предсказал для него 12 трансмембранных участков, что совпадает с информаицей в OPM. Причем участки он предсказал довольно точно, координаты отличаются на пару аминокислот.