Практикум 4
1. Составление списка гомологичных белков, включающих паралоги
Для поиска белков, гомологичных CLPX_ECOLI в протеомах бактерий, эти протеомы были собраны в один файл. На основе полученного файла была создана локальная база данных для запуска blastp. Blastp был запущен при параметрах Е-value - 0.001. В результате было получено 20 находок, которые можно увидеть здесь
2. Реконструкция и визуализация
Затем были использованы белковые последовательности находок blast для построения дерева. Использовалась программа FastME с параметрами:
"Gamma distributed rates across sites" — No
Starting tree — BIONJ
No refinement
100 бутстреп реплик
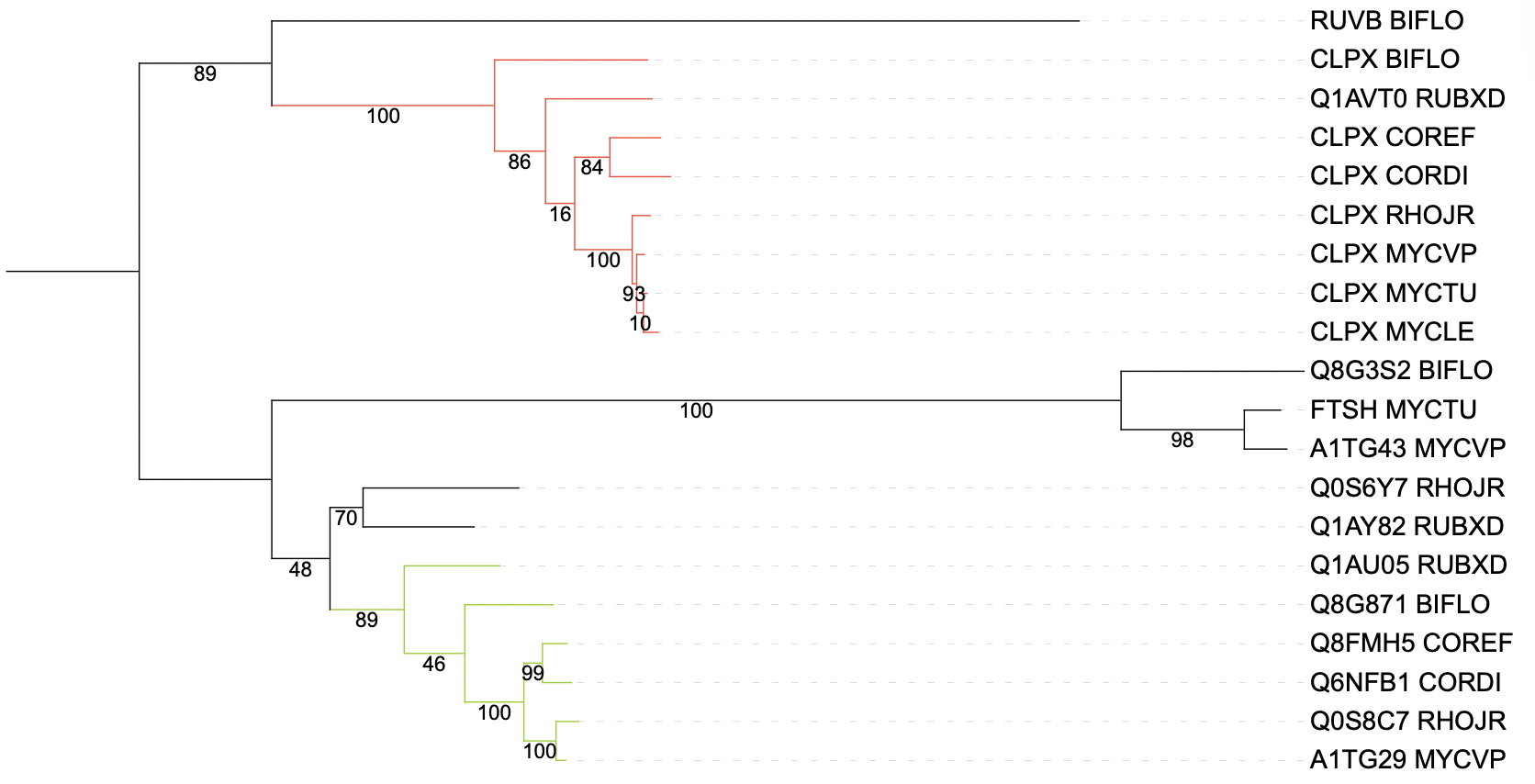
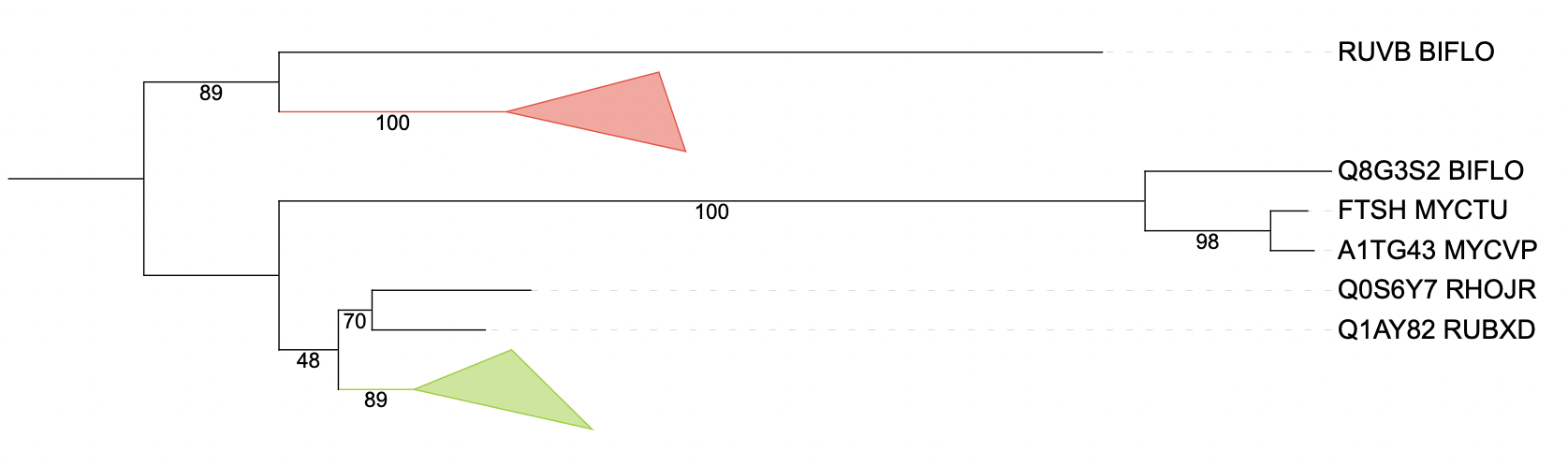
Полученное дерево было укоренено в среднюю точку.(Рис 1) На дереве можно выделить следующих ортологов: A1TG29 MYCVP и Q0S8C7 RHOJR, Q6NFB1 CORDI и Q8FMH5 COREF, Q0S6Y7 RHOJR и Q1AY82 RUBXD, A1TG43 MYCVP и FTSH MYCTU, CLPX MYCTU и CLPX MYCLE, CLPX COREF и CLPX CORDI; и следующих парологов: RUVB BIFLO и CLPX BIFLO,Q1AY82 RUBXD и Q1AU05 RUBXD, A1TG29 MYCVP и CLPX MYCVP6
На основании полученных данных можно выделить две ортологичные группы: ATP-зависимые протеазы Clp (красный), и ATP-зависимые цинковые металлопротеазы (салатовый).
Упомянутые группы были "схлопнуты".(Рис2)