Главная страница > Второй семестр > Сравнение фрагмента полного множественного выравнивания с соответствующим фрагментом "эталонного" выравнивания
Для выполнения задания выбран глутаминамидотрансферазный домен II класса (GATase_2, код доступа
PF00310).
В качестве эталонного выравнивания использовался участок выравнивания доменов, гомологичных данному и принадлежащих
следующим белкам (в скобках указаны номера начального и конечного аминокислотных остатков фрагментов последовательностей,
входящих в состав эталонного выравнивания):
Полные аминокислотные последовательности перечисленных выше белков приведены здесь.
| 1 | 0 | 2 | 0 | 3 | 0 | 4 | 0 | 5 | 0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | 1 | : | Y | A | S | D | N | A | I | L | A | H | E | R | L | S | I | V | D | V | N | A | - | - | G | A | Q | P | L | Y | N | Q | - | Q | K | T | H | V | L | A | V | N | G | E | I | Y | N | H | Q | A | L | R | A | E | Y | G | D | R | - | Y | Q | F | : | 5 | 6 | |||||||||||
| P | U | R | 1 | _ | H | A | E | I | N | 1 | : | R | L | Q | G | N | A | G | L | G | H | V | R | Y | P | T | A | G | S | S | S | V | S | E | A | Q | P | F | Y | V | N | S | P | Y | G | V | T | L | V | H | N | G | N | L | T | N | S | V | E | L | K | E | K | V | F | K | T | A | R | R | H | : | 6 | 0 | |||||||||||
| G | L | M | S | _ | B | A | C | S | U | 1 | : | N | V | E | A | K | A | G | I | G | H | T | R | W | A | T | H | G | E | P | S | Y | L | N | A | H | P | H | Q | S | A | - | L | G | R | F | T | L | V | H | N | G | V | I | E | N | Y | V | Q | L | K | Q | E | Y | L | - | Q | D | V | E | L | : | 5 | 8 | |||||||||||
| N | O | D | M | _ | R | H | I | L | V | 1 | : | P | L | S | G | T | V | G | I | A | H | T | R | W | A | T | H | G | A | P | T | E | C | N | A | H | P | H | F | T | D | - | - | - | G | V | A | V | V | H | N | G | I | I | E | N | F | S | K | L | K | D | A | L | A | E | V | G | T | K | F | : | 5 | 7 | |||||||||||
| G | F | A | 1 | _ | Y | E | A | S | T | 1 | : | T | F | V | S | H | C | G | I | A | H | T | R | W | A | T | H | G | R | P | E | Q | V | N | C | H | P | Q | R | S | D | P | E | D | Q | F | V | V | V | H | N | G | I | I | T | N | F | R | E | L | K | T | L | L | I | N | K | G | Y | K | F | : | 6 | 0 | |||||||||||
Рис. 1. Участок эталонного выравнивания последовательностей глутаминамидотрансферазного домена II класса следующих белков: ASNB_ECOLI, PUR1_HAEIN, GLMS_BACSU, NODM_RHILV, GFA1_YEAST (по данным Pfam). Красным выделены колонки, содержащие пять аминокислотных остатков, сходных по физико-химическим свойствам, зеленым — четыре сходных остатка, желтым — три сходных остатка.
| 1 | 0 | 2 | 0 | 3 | 0 | 4 | 0 | 5 | 0 | 6 | 0 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | : | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | C | S | I | F | G | V | F | D | I | K | T | D | A | V | E | L | R | K | K | A | L | E | L | S | R | L | M | R | H | R | ||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | C | G | I | V | G | I | V | S | Q | S | - | P | V | N | E | S | I | Y | A | A | L | T | L | L | Q | H | R | G | Q | D | ||||||
| G | L | M | S | _ | B | A | C | S | U | : | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | C | G | I | V | G | Y | I | G | Q | L | - | D | A | K | E | I | L | L | K | G | L | E | K | L | E | Y | R | G | Y | D | ||||||
| N | O | D | M | _ | R | H | I | L | V | : | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | C | G | I | V | G | I | V | G | H | K | - | P | V | S | E | R | L | I | E | A | L | G | R | L | E | Y | R | G | Y | D | ||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | T | F | V | S | H | C | G | I | A | H | T | R | W | A | T | H | G | R | P | E | Q | V | N | C | H | P | Q | R | S | D | P | E | D | Q | F | V | V | V | H | N | - | G | I | I | T | N | F | R | E | L | K | T | L | L | I | N | K | G | Y | K | ||||||
| 7 | 0 | 8 | 0 | 9 | 0 | 1 | 0 | 0 | 1 | 1 | 0 | 1 | 2 | 0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | : | G | P | D | W | S | G | I | Y | A | S | D | N | A | I | L | A | H | E | R | L | S | I | V | D | V | N | A | G | A | Q | P | L | Y | N | Q | Q | K | T | H | V | L | A | V | N | G | E | I | Y | N | H | Q | A | L | R | A | E | Y | G | D | R | ||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | A | A | G | I | V | T | V | D | D | E | N | R | F | R | L | R | K | A | N | G | L | V | S | D | V | F | H | Q | E | H | M | L | R | L | Q | G | N | A | G | L | G | H | V | R | Y | P | T | A | G | S | S | S | V | S | E | A | Q | P | F | Y | ||||||
| G | L | M | S | _ | B | A | C | S | U | : | S | A | G | I | A | V | A | N | E | Q | G | - | I | H | V | F | K | E | K | G | R | I | A | D | L | R | E | V | V | D | A | N | - | V | E | A | K | A | G | I | G | H | T | R | W | A | T | H | G | E | P | S | Y | L | N | A | H | P | H | Q | ||||||
| N | O | D | M | _ | R | H | I | L | V | : | S | S | G | V | A | T | I | F | E | G | E | - | L | H | R | R | R | A | E | G | K | L | G | N | L | K | T | R | L | K | E | A | P | L | S | G | T | V | G | I | A | H | T | R | W | A | T | H | G | A | P | T | E | C | N | A | H | P | H | F | ||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | F | E | S | D | T | D | T | E | C | - | - | - | I | A | K | L | Y | L | H | L | Y | N | T | N | L | Q | N | G | H | D | L | D | F | H | E | L | T | K | L | V | L | L | E | L | E | G | S | Y | G | L | L | C | K | S | C | H | Y | P | N | E | ||||||
| 1 | 3 | 0 | 1 | 4 | 0 | 1 | 5 | 0 | 1 | 6 | 0 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | : | Y | Q | F | Q | T | G | S | D | C | E | V | I | L | A | L | Y | Q | E | K | G | - | - | - | - | - | - | - | P | E | F | L | D | D | L | Q | G | M | F | A | F | A | |||||||||||||||||||||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | V | N | S | P | Y | G | V | T | L | V | H | N | G | N | L | T | N | S | - | - | - | - | - | - | - | - | - | V | E | L | K | E | K | V | F | K | T | A | R | R | H | |||||||||||||||||||||||||
| G | L | M | S | _ | B | A | C | S | U | : | S | A | L | G | R | F | T | L | V | H | N | G | V | I | E | N | Y | V | Q | L | K | Q | E | Y | L | Q | D | V | E | L | K | S | D | T | D | T | E | V | V | V | Q | |||||||||||||||||||||||||
| N | O | D | M | _ | R | H | I | L | V | : | T | D | G | V | A | V | V | H | N | G | I | I | E | N | F | S | K | L | K | D | A | L | A | E | V | G | - | T | K | F | Q | T | D | T | D | T | E | V | I | A | H | |||||||||||||||||||||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | V | I | A | T | R | K | G | S | P | L | L | I | G | V | K | S | E | K | K | L | K | V | D | F | V | D | - | V | E | F | P | E | E | N | A | G | Q | P | E | I | P | |||||||||||||||||||||||||
Рис. 2. Участок множественного выравнивания последовательностей следующих белков: ASNB_ECOLI, PUR1_HAEIN, GLMS_BACSU, NODM_RHILV, GFA1_YEAST (выравнивание проведено с помощью программы ClustalW). Синим выделены фрагменты последовательностей ASNB_ECOLI и GFA1_YEAST, входящие в состав эталонного выравнивания (эти фрагменты были неверно сопоставлены другим последовательностям). Зеленым выделены столбцы множественного выравнивания последовательностей PUR1_HAEIN, GLMS_BACSU и NODM_RHILV, совпадающие со столбцами эталонного выравнивания, красным выделены столбцы множественного выравнивания тех же последовательностей, не совпадающие со столбцами эталонного выравнивания (см. рис.1).
| 1 | 0 | 2 | 0 | 3 | 0 | 4 | 0 | 5 | 0 | 6 | 0 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | : | Y | A | S | D | N | - | A | I | L | A | H | E | R | L | S | I | V | D | V | N | A | G | A | Q | P | L | Y | N | Q | Q | K | T | H | - | - | - | - | - | - | V | L | A | V | N | G | E | I | Y | N | H | Q | A | L | R | A | E | Y | G | D | R | ||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | V | T | V | D | D | E | - | N | R | F | R | L | R | K | A | N | G | L | V | S | D | V | F | H | Q | E | H | M | L | R | L | Q | G | - | - | - | - | - | - | N | A | G | L | G | H | V | R | Y | P | T | A | G | S | S | S | V | S | E | A | Q | ||||||
| G | L | M | S | _ | B | A | C | S | U | : | A | V | A | N | E | - | - | Q | G | I | H | V | F | K | E | K | G | R | I | A | D | L | R | - | E | V | V | D | A | N | V | E | A | - | - | - | - | - | - | K | A | G | I | G | H | T | R | W | A | T | H | G | E | P | S | Y | L | N | A | H | ||||||
| N | O | D | M | _ | R | H | I | L | V | : | A | T | I | F | E | - | - | G | E | L | H | R | R | R | A | E | G | K | L | G | N | L | K | T | R | L | K | E | A | P | L | S | G | - | - | - | - | - | - | T | V | G | I | A | H | T | R | W | A | T | H | G | A | P | T | E | C | N | A | H | ||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | A | I | D | G | D | E | A | D | S | T | F | I | Y | K | Q | I | G | K | V | S | A | L | K | E | E | I | T | K | Q | N | P | N | R | D | V | T | F | V | S | H | C | G | I | A | H | T | R | W | A | T | H | G | R | P | E | Q | V | N | C | H | ||||||
| 7 | 0 | 8 | 0 | 9 | 0 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | : | Y | Q | F | Q | T | G | S | D | C | E | V | I | L | A | L | Y | Q | E | K | G | P | E | F | L | D | D | L | Q | G | M | F | A | F | A | L | Y | ||||||||||||||||||||||||||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | P | F | Y | V | N | S | P | Y | G | V | T | L | V | H | N | G | N | L | T | N | S | V | E | L | K | E | K | V | F | K | T | A | R | R | H | V | ||||||||||||||||||||||||||||||
| G | L | M | S | _ | B | A | C | S | U | : | P | H | Q | S | A | L | G | - | R | F | T | L | V | H | N | G | V | I | E | N | Y | V | Q | L | K | Q | E | Y | L | Q | D | - | - | V | E | L | ||||||||||||||||||||||||||||||
| N | O | D | M | _ | R | H | I | L | V | : | P | H | F | T | D | G | - | - | - | V | A | V | V | H | N | G | I | I | E | N | F | S | K | L | K | D | A | L | A | E | V | G | - | T | K | F | ||||||||||||||||||||||||||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | P | Q | R | S | D | P | E | D | Q | F | V | V | V | H | N | G | I | I | T | N | F | R | E | L | K | T | L | L | I | N | K | G | - | Y | K | F | ||||||||||||||||||||||||||||||
Рис. 3. Участок множественного выравнивания последовательностей, составленных из первых 180 аминокислотных остатков следующих белков: ASNB_ECOLI, PUR1_HAEIN, GLMS_BACSU, NODM_RHILV, GFA1_YEAST (выравнивание проведено с помощью программы ClustalW). Синим выделен фрагмент последовательности ASNB_ECOLI, входящей в состав эталонного выравнивания (этот фрагмент был неверно сопоставлен другим последовательностям). Зеленым выделены столбцы множественного выравнивания последовательностей PUR1_HAEIN, GLMS_BACSU, NODM_RHILV и GFA1_YEAST, совпадающие со столбцами эталонного выравнивания, красным выделены столбцы множественного выравнивания тех же последовательностей, не совпадающие со столбцами эталонного выравнивания (см. рис.1).
| 1 | 0 | 2 | 0 | 3 | 0 | 4 | 0 | 5 | 0 | 6 | 0 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| A | S | N | B | _ | E | C | O | L | I | : | Y | A | S | D | N | A | I | L | A | H | E | R | L | S | I | V | D | V | N | - | - | A | G | A | Q | P | L | Y | N | Q | Q | K | - | T | H | V | L | A | V | N | G | E | I | Y | N | H | Q | A | L | R | A | E | Y | G | D | R | - | - | Y | Q | ||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | R | L | Q | G | N | A | G | L | G | H | V | R | Y | P | T | A | G | S | S | S | V | S | E | A | Q | P | F | Y | V | N | S | P | Y | G | V | T | L | V | H | N | G | N | L | T | N | S | V | E | L | K | E | K | V | F | K | T | A | R | R | H | ||||||
| G | L | M | S | _ | B | A | C | S | U | : | N | V | E | A | K | A | G | I | G | H | T | R | W | A | T | H | G | E | P | S | Y | L | N | A | H | P | H | Q | S | A | L | G | - | R | F | T | L | V | H | N | G | V | I | E | N | Y | V | Q | L | K | Q | E | Y | L | Q | D | - | - | V | E | ||||||
| N | O | D | M | _ | R | H | I | L | V | : | P | L | S | G | T | V | G | I | A | H | T | R | W | A | T | H | G | A | P | T | E | C | N | A | H | P | H | F | T | D | G | - | - | - | V | A | V | V | H | N | G | I | I | E | N | F | S | K | L | K | D | A | L | A | E | V | G | - | T | K | ||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | T | F | V | S | H | C | G | I | A | H | T | R | W | A | T | H | G | R | P | E | Q | V | N | C | H | P | Q | R | S | D | P | E | D | Q | F | V | V | V | H | N | G | I | I | T | N | F | R | E | L | K | T | L | L | I | N | K | G | - | Y | K | ||||||
| A | S | N | B | _ | E | C | O | L | I | : | F | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| P | U | R | 1 | _ | H | A | E | I | N | : | V | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G | L | M | S | _ | B | A | C | S | U | : | L | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| N | O | D | M | _ | R | H | I | L | V | : | F | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G | F | A | 1 | _ | Y | E | A | S | T | : | F | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис. 4. Участок множественного выравнивания фрагментов последовательностей следующих белков: ASNB_ECOLI, PUR1_HAEIN, GLMS_BACSU, NODM_RHILV, GFA1_YEAST (выравнивание проведено с помощью программы ClustalW). Зеленым выделены столбцы, совпадающие со столбцами эталонного выравнивания, красным — не совпадающие со столбцами эталонного выравнивания (см. рис.1).
Множественное выравнивание полных последовательностей белков было осуществлено с помощью программы ClustalW. Результаты
выравнивания приведены на рис. 2. В связи с тем, что участки последовательностей ASNB_ECOLI
и GFA1_YEAST, входящие в состав эталонного выравнивания (рис. 1), были неверно сопоставлены
участкам других последовательностей, ни одна из
колонок множественного выравнивания не присутствует в эталонном выравнивании. Совпадающие колонки могут быть найдены только в пределах
трех оставшихся последовательностей: PUR1_HAEIN, GLMS_BACSU
и NODM_RHILV. Для этих последовательностей 29 из 60-ти столбцов эталонного выравнивания
совпадают со столбцами множественного выравнивания.
Отклонения от эталонного выравнивания можно объяснить тем, что
на вход программы ClustalW были поданы полные последовательности белков. Так как ClustalW предназначена для построения
глобальных выравниваний, короткие участки последовательностей, сходные в результате случайного совпадения аминокислотных
остатков, оказывают большое влияние на результат выравнивания и становятся причиной ошибок. Для проверки этой гипотезы было проведено
множественное выравнивание укороченных последовательностей белков, составленных из первых 180 аминокислотных остатков
(участки последовательностей, принадлежащие эталонному выравниванию, входят в состав оставшихся фрагментов). Результаты выравнивания
приведены на рис. 3.
Из пяти последовательностей участок только одной (ASNB_ECOLI)
был неверно сопоставлен остальным последовательностям. Для других последовательостей 46 из 60-ти колонок эталонного выравнивания
совпали с колонками множественного выравнивания. Расхождения в основном связаны с различиями в расстановке гэпов.
Таким образом, после удаления негомологичных участков выравниваемых последовательностей результат множественного выравнивания приблизился к эталонному
выравниванию. Следовательно, именно протяженные негомологичные участки последовательностей препятствовали построению правильного
выравнивания гомологичных участков.
Повысить качество результата множественного выравнивания можно путем удаления тех участков выравниваемых последовательностей,
про которые заранее известно то, что они негомологичны. Такие участки можно выявить, например, путем парного локального выравнивания.
Данная методика была проверена на белках эталонного выравнивания. Для выявления гомологичных участков было проведено локальное выравнивание
последовательности ASNB_ECOLI с каждой из остальных последовательностей. Выравнивание осуществлялось с помощью
программы MatcherP (штраф за открытие гэпа составлял 14, штраф за удлиннение гэпа — 4, использовалась матрица замен BLOSUM62).
Среди различных вариантов локальных выравниваний были выбраны те, вес которых наибольший и длина
приблизительно соответствует длине домена белка (не менее 40-50 аминокислотных остатков). Добиться необходимой длины
можно с помощью использования различных матриц серии BLOSUM. Например, если длины локальных выравниваний при использовании BLOSUM62 слишком малы,
следует использовать матрицы с меньшими номерами, которые позволяют выявлять менее сходные и более протяженные участки последовательностей
(см. сайт Европейского института биоинформатики).
Ниже приведены номера начальных и конечных аминокислотных остатков
гомологичных участков последовательностей, выявленных путем локального выравнивания (в скобках дана ссылка на результат локального выравнивания):
Для проведения множественного выравнивания были взяты гомологичные фрагменты последовательностей белков, удлиненные приблизительно
на 30 аминокислотных остатков в обе стороны. Удлинение проведено во избежание неполного вхождения последовательностей эталонного
выравнивания в состав последовательностей, предназначенных для проведения множественного выравнивания
(выравнивемые последовательности приведены здесь).
Результат множественного выравнивания представлен на рис. 4.
47 из 60 колонок эталонного выравнивания совпали с колонками множественного выравнивания. При этом ни один из участков
пяти последовательностей не был ошибочно сопоставлен другим последовательностям. Расхождения связаны с различиями в расстановке гэпов.
Высокое сходство результатов множественного выравнивания с эталонным говорит о корректности применения методики.
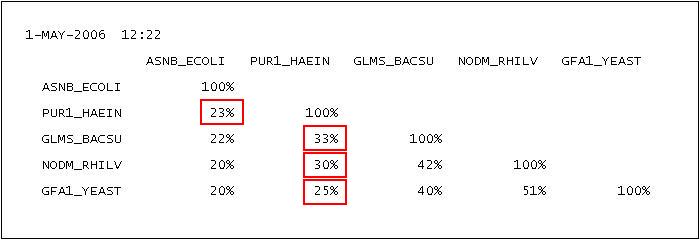
С помощью программы GENEDOC были получены матрицы попарной идентичности для эталонного выравнивания и для участка
множественного выравнивания (с предварительным выявлением гомологичных участков), соответствующего эталонному выравниванию (рис. 5, 6).
Матрицы содержат информацию о количестве идентичных позиций для каждой пары последовательностей. Например, наиболее сходными участками
последовательностей эталонного выравнивания являются участки последовательностей NODM_RHILV и
GFA1_YEAST (процент идентичных позиций составляет 51%), а наименее сходными участками
— участки последовательностей ASNB_ECOLI и
GFA1_YEAST (процент идентичных позиций составляет 20%).
В связи с тем, что для эталонного выравнивания (как для биологически правильного) количество совпадающих позиций
последовательностей близко к максимальному, качество множественного выравнивания можно оценивать путем сравнения матриц попарной
идентичности. Чем ближе проценты идентичности матрицы, составленной для участков последовательностей множественного выравнивания, к процентам идентичности
матрицы, составленной для эталонного выравнивания, тем ближе результаты множественного выравнивания к эталонному. Такой способ оценки справедлив
для тех случаев, когда (1) вес эталонного выравнивания действительно близок к максимальному, и (2) не существует выравниваний,
существенно отличных от эталонного, но обладающих сходным весом. Так как вероятность невыполнения этих условий достаточно мала,
было проведено сравнение матриц попарной идентичности, приведенных на рис. 5 и 6. Единственным отличием матрицы, составленной по
результатам множественного выравнивания, от матрицы, составленной по результатам эталонного, является то, что сходство последовательности
PUR1_HAEIN с другими последовательностями на несколько процентов ниже. Это говорит о том, что в
целом результаты множественного выравнивания соответствуют результатам эталонного, однако имеются небольшие расхождения, связанные
с сопоставлением участка последовательности PUR1_HAEIN участкам других последовательностей.

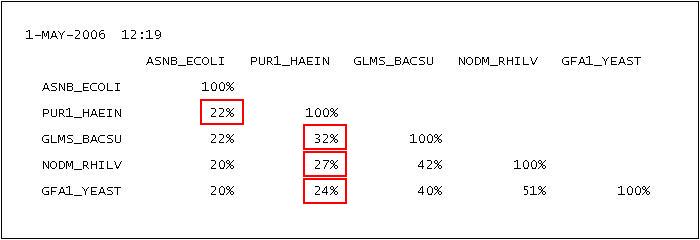
Рис. 6. Матрица попарной идентичности, составленная для участка множественного выравниванивания (с предварительным выявлением гомологичных участков), соответствующего эталонному выравниванию. Значения, отличающиеся от значений матрицы попарной идентичности эталонного выравнивания (рис. 5), выделены красным.
© Куравский Михаил Львович, 2006