Обзор генома и протеома бактерии Paenibacillus odorifer
Михайлова Екатерина
Резюме
В данной работе содержится анализ генома и протеома бактерии Paenibacillus odorifer. Посчитаны GC-skew и частоты стоп-кодонов, построена гистограмма длин белков, показано распределение генов по цепям ДНК.
Ключевые слова: геном, протеом, бактерия, Paenibacillus odorifer
1. Введение
Paenibacillus odorifer – грамположительная, спорообразующая бактерия, была выделена из лука-порея и других продуктов питания [1]. Показано, что данная бактерия способствует порче молока [2,3], ее споры стабильны к термической обработке, а адаптации, связанные с холодовым шоком [6], позволяют расти при температурах охлаждения на протяжении всего хранения молока, вызывая протеолиз и липолиз [2-5]. Также P. odorifer способна к нитрат и нитрит редукции [7]. Целью работы является изучение генома и протеома данной бактерии
2. Методы
Информация о геноме и протеоме получена из базы данных NCBI [8].
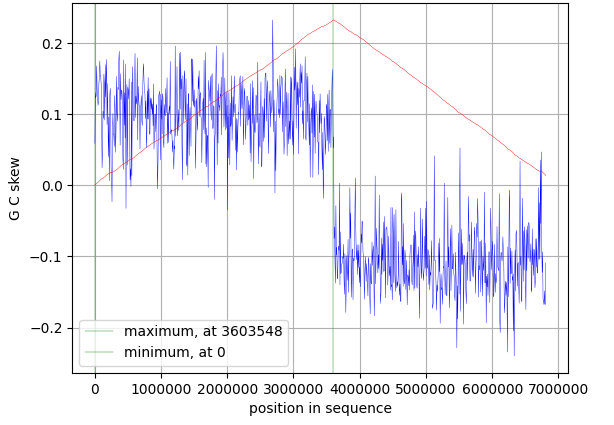
Для построения графика 1 была использована программа GenSkew[9]. Данная программа строит график интегрального значения (cumulative GC-skew) в определенном диапазоне(window), по позициям идут с некоторым шагом(step). Также на основе графика выводятся значения максимума и минимума. Window и step программа выбирает автоматически, на основе предложенного генома.
Код, с помощью которого получены данные из таблицы 1, был написан на языке Python, вместе с пояснениями его можно найти по ссылке в разделе “Сопроводительные материалы”.
Для составления рисунка 1, таблицы 2 и 3 была использована программа Google sheets, и ее функции: СРЗНАЧ, СТАНДОТКЛОНП, МЕДИАНА, МИН, МАКС – для получения статистических данных протеома; СЧЁТЕСЛИМН, BINOM.DIST – для нахождения количества генов на цепях и оценки случайности. (ссылки на таблицы приведены в разделе “Сопроводительные материалы”.
3. Результаты и обсуждение
3.1 Стандартные данные о геноме
Геном Paenibacillus odorifer состоит из одной кольцевой хромосомы и имеет длину 6,8 Mb, что является средним значением для рода Paenibacillus [10]. GC-состав данной бактерии равен 44,1 % [11], это немного меньше GC-состава бактерии Paenibacillus sp. JDR-2 (50,3 %) из того же рода [12]. Не очень высокий GC-состав согласуется с тем, что бактерия не является термофильной.
3.2 GC-skew
GC-skew представляет собой соотношение между нуклеотидами G (гуанин) и C (цитозин) и вычисляется по формуле:
𝐺𝐶 𝑠𝑘𝑒𝑤 = (𝑛(𝐺) + 𝑛(𝐶))/(𝑛(𝐺) − 𝑛(𝐶))
где n(G), n(C) – число нуклеотидов G и С. С его помощью можно найти точку начала и терминации репликации, для этого вычисляется cumulative GC-skew, который представляет собой сумму всех GC-skew, посчитанных ранее. Известно, что минимуму GC-skew соответствует точка начала репликации (oriC), а максимуму – точка терминации репликации (ter).
С помощью программы[9] построен график cumulative GC-skew вдоль генома Paenibacillus odorifer, window и step равны 6812 п.н.
Минимум cumulative GC-skew равен 0, а максимум – 3603548. Значит предположительные oriC и ter соответственно равны 0 и 3603548.
3.3 Частоты стоп-кодонов в кодирующих последовательностях
Частоты стоп-кодонов приведены в таблице 1
Данные подтверждают гипотезы, приведенные в статье[13]: высокая частота кодона TAA может быть связана тем, что TAA может узнаваться и фактором терминации RF1 и фактором терминации RF2, в то время как TGA и TAG – только одним
Также бактерия предпочтительнее использует TAA при низком GC-составе, что как раз характерно для Paenibacillus odorifer.
| Стоп-кодон | TAG | TAA | TGA |
| Количество | 1292 | 3355 | 1260 |
3.4 Гистограмма длин белков
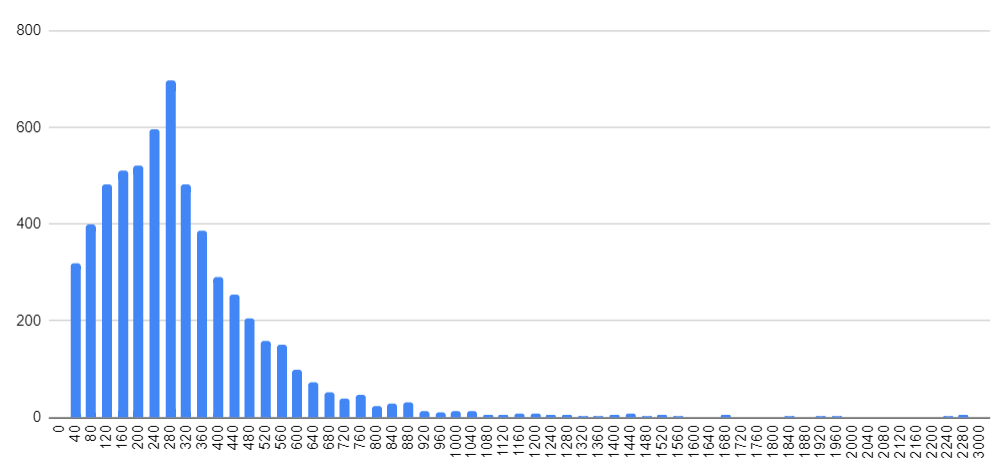
На рисунке 1 представлена гистограмма длин белков с карманом 40, согласно этим данным наибольшее количество белков имеет длину 280-300 аминокислотных остатков.
Также проведены статистические расчеты, данные представлены в таблице 2. Средняя длина равна 330, самый короткий белок имеет длину 25 аминокислотных остатков, а самый длинный – 2655.
| Показатель | Значение |
| Среднее | 330 |
| Стандартное отклонение | 233 |
| Медиана | 289 |
| Минимальное значение | 25 |
| Максимальное значение | 2655 |
3.5 Распределение генов по цепям
В таблице 3 приведено распределение генов по двум цепям ДНК, а также приведена степень случайности.
Когда степень случайности низкая, в данном случае у генов, кодирующих белки, и у псевдогенов (гены, которые когда-то кодировали белок, но утратили эту возможность) можно сказать, что такое распределение по цепям возникло под влиянием каких-то факторов и возможно имеет важное для бактерии значение.
В случае когда степень случайности велика, как у генов транспортных РНК, можно сделать противоположный вывод.
Степень случайности распределения генов рибосомальных РНК оценить затруднительно, нельзя сказать точно, имеет ли для бактерии значение, расположен ген на “+” или “-” цепи.
| Название | На “+” цепи | На “-” цепи | Степень случайности |
| gene protein_coding | 2841 | 3018 | 0,022 |
| pseudogene | 37 | 22 | 0,067 |
| rRNA | 18 | 12 | 0,362 |
| tRNA | 40 | 47 | 0,520 |
4. Сопроводительные материалы
5. Благодарность
Благодарю преподавательский состав за помощь в освоении новых методов, которые помогли мне написать этот мини-обзор.
6. Список источников
- Berge, O., Guinebretiere, M.H., Achouak, W., Normand, P. and Heulin, T. (2002)Paenibacillus graminis sp. nov. and Paenibacillus odorifer sp. nov., isolated from plant roots, soil and food. Int J Syst Evol Microbiol 52, 607– 616.
- Beno SM, Cheng RA, Orsi RH, Duncan DR, Guo X, Kovac J, Carroll LM, Martin NH, Wiedmann M. 2020. Paenibacillus odorifer, the predominant Paenibacillus species isolated from milk in the United States, demonstrates genetic and phenotypic conservation of psychrotolerance but clade-associated differences in nitrogen metabolic pathways. mSphere 5:e00739-19
- Ivy RA, Ranieri ML, Martin NH, Bakker HC, Xavier BM, Wiedmann M, Boor KJ. 2012. Identification and characterization of psychrotolerant sporeformers associated with fluid milk production and processing Appl EnvironMicrobiol 78:1853–1864
- Doll EV, Scherer S, Wenning M. 2017. Spoilage of microfiltered and pasteurized extended shelf life milk is mainly induced by psychrotolerant spore-forming bacteria that often originate from recontamination. Front Microbiol 8:135.
- Ranieri M, Huck J, Sonnen M, Barbano D, Boor K. 2009. High temperature, short time pasteurization temperatures inversely affect bacterial numbers during refrigerated storage of pasteurized fluid milk. J Dairy Sci 92:4823–4832
- Phadtare S. 2004. Recent developments in bacterial cold-shock response. Curr Issues Mol Biol 6:125–136.
- Buxton R. 2011. Nitrate and nitrite reduction test protocols. American Society for Microbiology, Washington, DC. Accessed 1 October 2019.
- База данных геномов и протеомов NCBI
- Программа GenSkew
- Данные о GC-составе бактерий рода Paenibacillus
- Данные о GC-составе Paenibacillus odorifer
- Lightfield J, Fram NR, Ely B (2011) Across Bacterial Phyla, Distantly-Related Genomes with Similar Genomic GC Content Have Similar Patterns of Amino Acid Usage. PLoS ONE 6(3): e17677. doi:10.1371/journal.pone.0017677
- Alexander T Ho, Laurence D Hurst, Variation in Release Factor Abundance Is Not Needed to Explain Trends in Bacterial Stop Codon Usage, Molecular Biology and Evolution,2021;,msab326,https://doi.org/10.1093/molbev/msab326