Нуклеотидный BLAST
Выбор контига
В сборке генома Nematostella vectensis не было контигов длиной менее 10 тысяч пар оснований содержащих при этом CDS, поэтому был выбран контиг
NW_026019269.1 длиной 21264 п.н., и взят его фрагмент. Координаты фрагмента 1-3000, последовательность фрагмента.
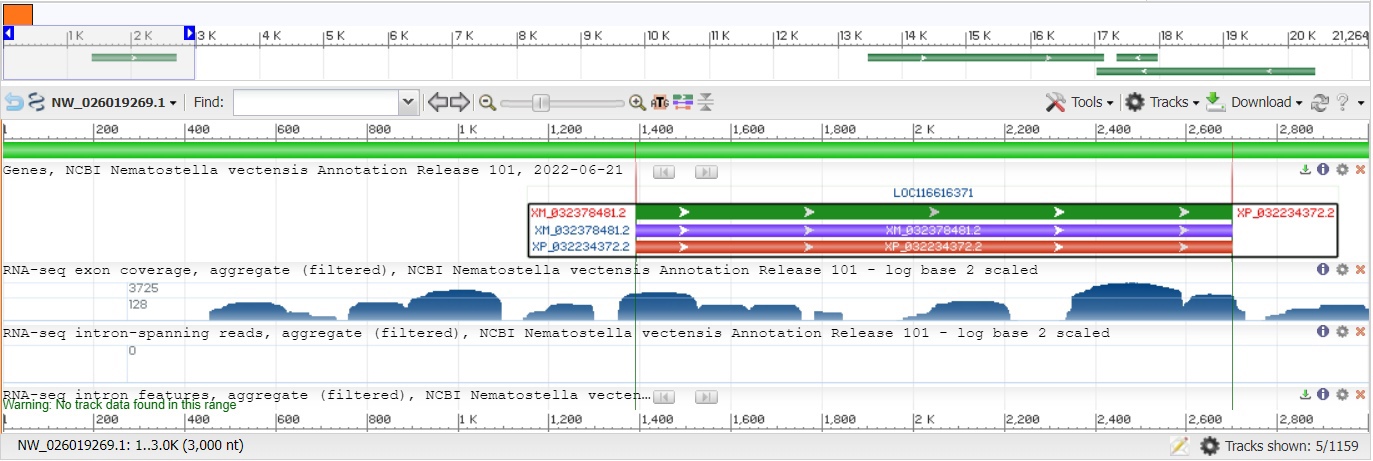
На рис.1 видно, что данный фрагмент содержит один ген и соответсвующие ему мРНК и CDS.
Запуск BLAST
Использовался BLAST на сайте NCBI. Для исключения был выбран таксон Cnidaria(taxid: 6073).1. Megablast и blastn
Так как при стандартных параметрах находок не было, megablast запускался при длине слова 16 и e-value 0.5. Был выдан только один результат. При запуске blastn со стандартными параметрами находок было 7, минимальный e-value среди находок 0.003.
Во всех этих случаях выровнялись небольшие кусочки (около 40 нуклеотидов) в конце CDS, включающие поли(А)-хвост. Видимо это не какой-то домен белка, а просто совпавший концевой участок.
Megablast нужен для поиска практически идентичных последовательностей (может быть использован при картирования генома).
Blastn - для поиска достаточно похожих последовательностей (поиск гомологов среди близкородственных видов), для некодирующих белок последовательностей (поиск гомологов среди тРНК или рРНК).
2. Blastx и tblastx
Blastx был запущен со стандартными параметрами, но количество выдаваемых выравниваний было увеличено до 5000. Программа выдала 3457 находок, в основном это гипотетические или неохарактеризованные белки. На этот раз выровнялись уже более длинные участки (около 400 аминокислот), можно предположить, что эти участки являются доменом белка.
К сожалению, при запуске tblastx, выходила ошибка о том, что программе не хватает вычислительных ресурсов для обработки запроса, несмотря на попытки менять параметры.
Blastx - транслирует нуклеотидную последовательность и ищет в белковой базе (подходит для поиска дальних гомологов, но необходимо чтобы белки были предсказаны).
Tblastx - транслирует нуклеотидную последовательность и ищет в транслированной базе (также подходит для поиска дальних гомологов, белки необязательно предсказаны).
Поиск генов основных рибосомальных РНК по генам E. coli
Для работы локального BLAST геном Nematostella vectensis был проиндексирован с помощью команды:
makeblastdb -in GCF_932526225.1.fna -dbtype nuclПоиск проводился по генам 16S рРНК (входит в состав малой субъединицы рибосомы, участвует в инициации трансляции), и 23S рРНК (входит в состав большой субъединицы, участвует в пептидилтрансферазной реакции).
Так как нам даны последовательности рРНК для которых нет соотвествующих белков, использовался blastn. Находок было много, поэтому E-value был
изменен со стандартного 10 на 0.05, и использована опция -outfm для более удобного просмотра.
Команды:
blastn -task blastn -query rRNA_ecoli_16S.txt -db GCF_932526225.1.fna -out 16S_out.txt -outfmt 7 -evalue 0.05 blastn -task blastn -query rRNA_ecoli_23S.txt -db GCF_932526225.1.fna -out 23S_out.txt -outfmt 7 -evalue 0.05
Для 16S рРНК было найдено 95 гомологов, для 23S рРНК - 435. Эукариотическими гомологами 16S и 23S рРНК являются 18S и 28S рРНК, их мы ожидаем увидеть среди находок.
Гомологичные 16S рРНК последовательности входили в состав участков, аннотированных как "рРНК малой субъединицы рибосомы". А гомологичные 23S рРНК последовательности принадлежали фрагментам генома с аннотацией "рРНК большой субъединицы рибосомы". Это подтверждает наше предположение.