Трансмембранные белки
OPM
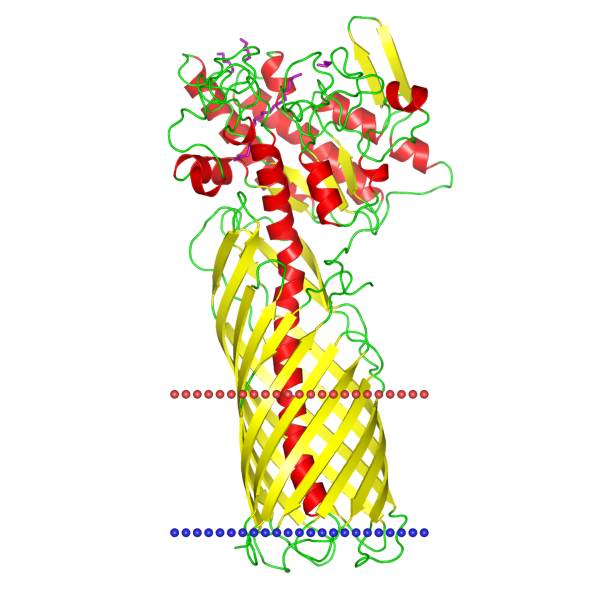
Для анализа из базы OPM был выбран белок с β-листами в трансмембранной части - Esterase EstA (эстераза EstA). Данный белок участвует в синтезе рамнолипидов и образовании биопленки у Pseudomonas aeruginosa. Идентификатор PDB - 3KVN, идентификатор UniProt - ESTA_PSEAE.
| Название белка | Esterase EstA |
| Толщина гидрофобной части белка в мембране | 24.4 Å |
| Координаты трансмембранных участков | 1(347-356),2(374-382),3(388-397),4(414-424),5(428-437),6(466-475),7(484-494),8(523-532),9(538-547),10(581-589),11(595-604),12(613-620) |
| Среднее количество остатков в одном β-тяже белка | 9 |
| Локализация | Внешняя мембрана |
.
DeepTMHMM
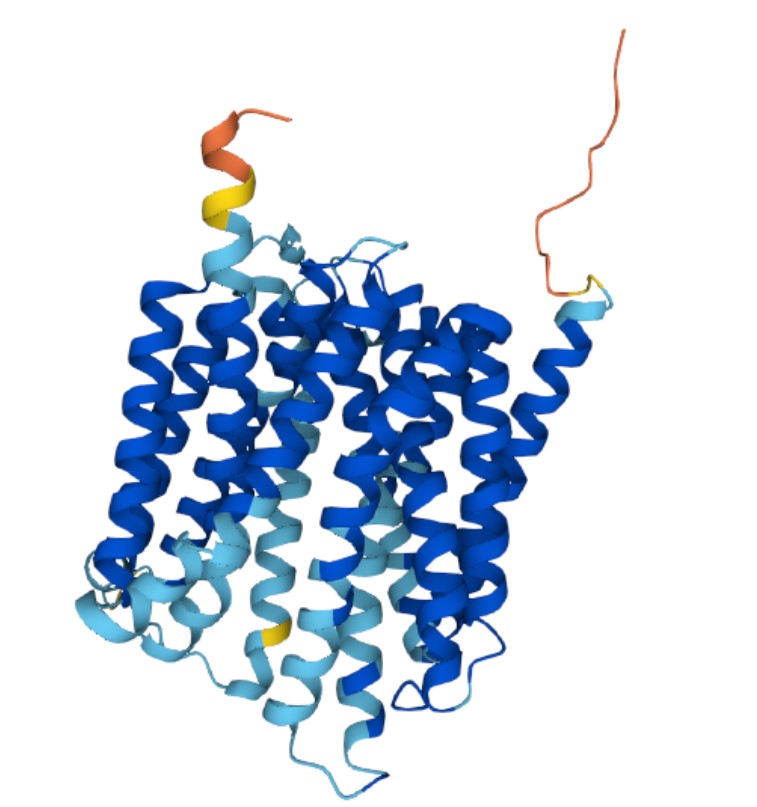
Также для практикума нам был выдан α-спиральный белок. Так как предложенный идентификатор (Y1401_MYCTU) устарел (запись была разделена на две), я решила взять белок из резерва - MAMH_MAGGM. Он вероятнее всего участвует в транcпорте железа и необходим для биоминерализации магнитосомы. Его последовательность и предсказанная с помощью AlphaFold трехмерная структура.
Для MAMH_MAGGM и β-листового белка с помощью сервиса DeepTMHMM были предсказаны трансмембранные элементы:
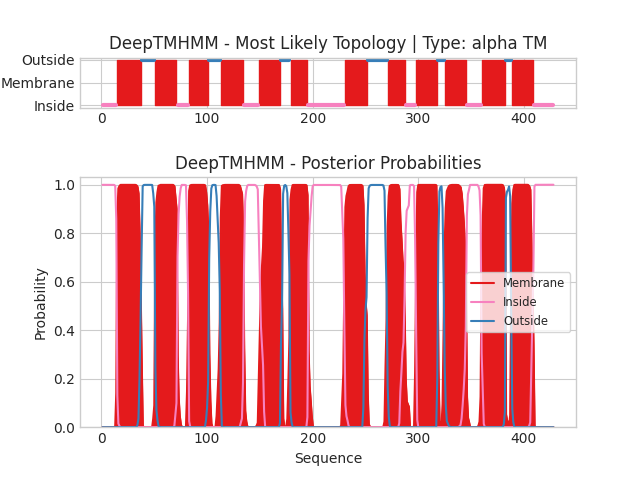
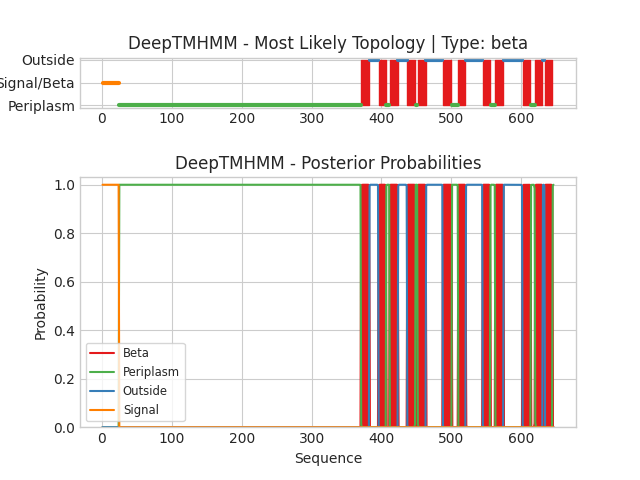
На графике по оси абсцисс отложен номер аминокислотного остатка, по оси ординат - вероятность определенной локализации остатка (нижний график), и предсказанная топология (верхний график).
Текстовая выдача для α-спирального белка и β-листового белка.
PPM
Также предсказание для α-спирального белка MAMH_MAGGM было получено с помощью сервиса PPM 3.0. Он был запущен со следующими параметрами:
- Type of membrane: vacuole membrane (согласно литературе [1] данный белок расположен внутри мембраны магнитосомы)
- Allow curvature: no
- Topology (N-ter): in (согласно предсказанию DeepTMHMM)
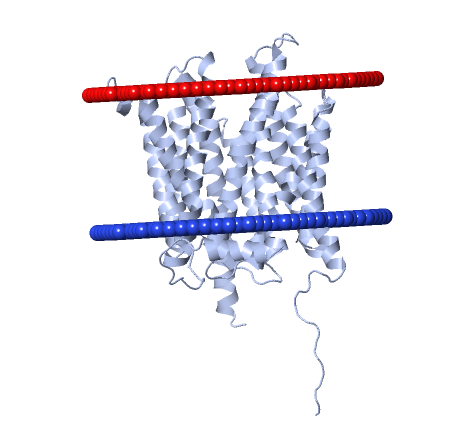
| Название белка | Magnetosome protein MamH |
| Толщина гидрофобной части белка в мембране | 31.0 ± 0.8 Å |
| Координаты трансмембранных участков | 1(17- 38), 2(50- 72), 3(83- 106), 4(109- 134), 5(151- 176), 6(177- 195), 7(228- 251), 8(266- 287), 9(299- 316),10(323- 344),11(360- 383),12(387- 407) |
| Среднее количество остатков в одном β-тяже белка | 22 |
| Локализация | мембрана магнитосомы |
.
Сравнение алгоритмов предсказания
Для β-листового белка предсказание трансмембранных участков сервером DeepTMHMM не очень совпало с информацией из базы OPM. Он не предсказал первый мембранный участок, и предсказал дополнительный участок в конце. Координаты остальных также часто не совпадают.
Для α-спирального белка предсказание DeepTMHMM практически совпадает с предсказанием PPM, координаты отличаются на пару нуклеотидов.
В AlphaFold плохое качество предсказания имеют N и C концы белка и ещё два небольших участка. В остальном качество высокое (Рис. 5). Я думаю достоверность модели учитывается при предсказании на сервере PPM.