Практикум 4. Визуализация деревьев
Составление списка гомологичных белков
Для работы blastp протеомы выбранных в предыдущих практикумах бактерий были объединены в один файл и проиндексированы.
makeblastdb -in all_proteomes.fa -dbtype protДалее были найдены гомологи белка CLPX_ECOLI:
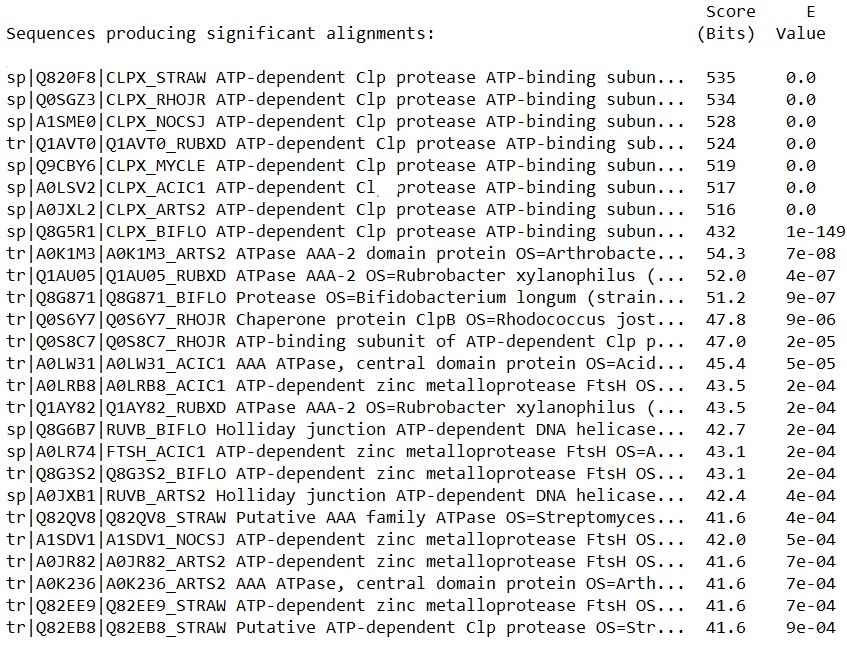
blastp -query clpx_ecoli.fa -db all_proteomes.fa -evalue 0.001 -out clpx_out.txtБыло получено 26 находок (Табл. 1), среди которых АТФ-зависимые Clp протеазы и цинковые протеазы, а также другие белки обладающие АТФазной активностью.
Реконструкция и визуализация
С использованием сервиса NGPhylogeny.fr были выравнены последовательности гомологов (алгоритм MAFFT) и реконструировано дерево (программа FastME c параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, 100 бутстреп реплик). Полученное дерево в Newick формате.
Также было получено изображение в ITOL (Рис. 1):
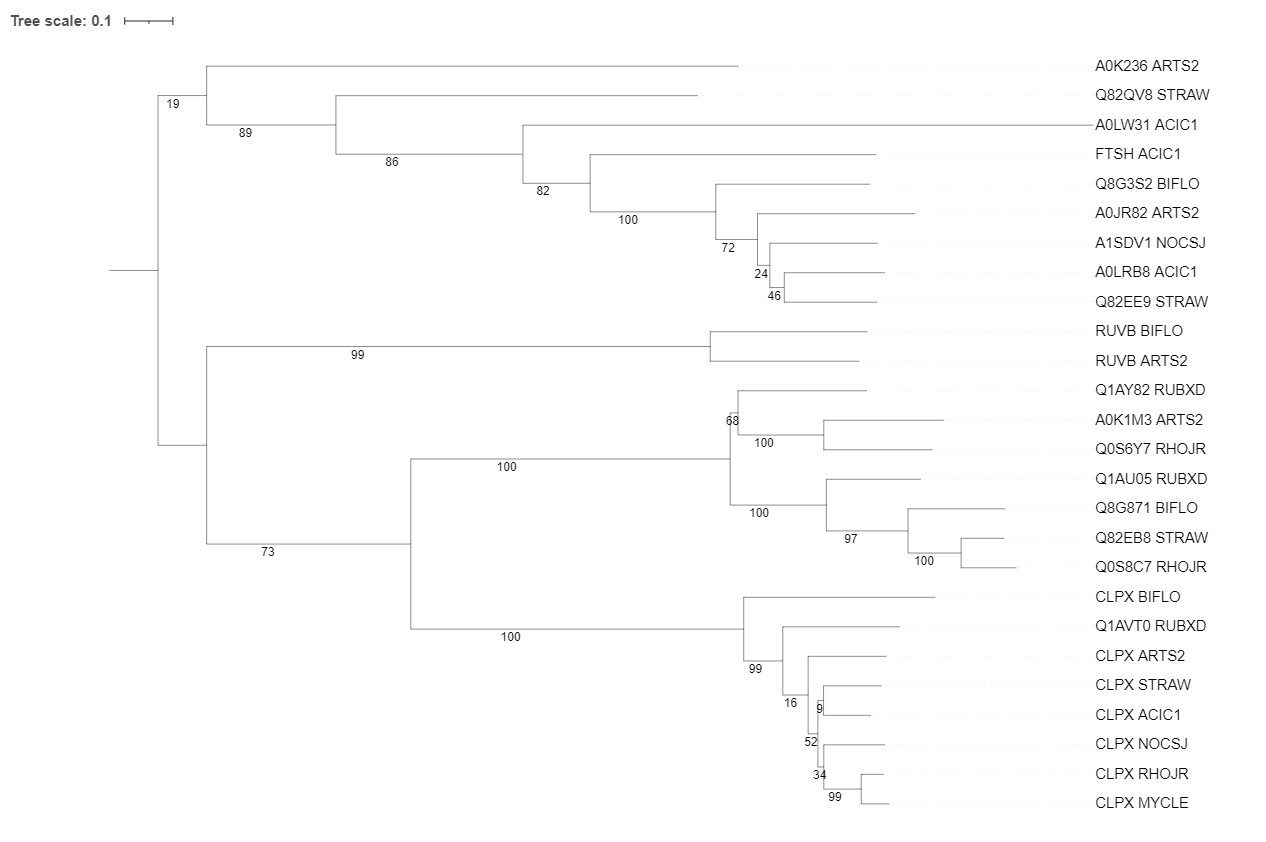
На дереве можно выделить несколько пар отрологов и паралогов (Табл. 2):
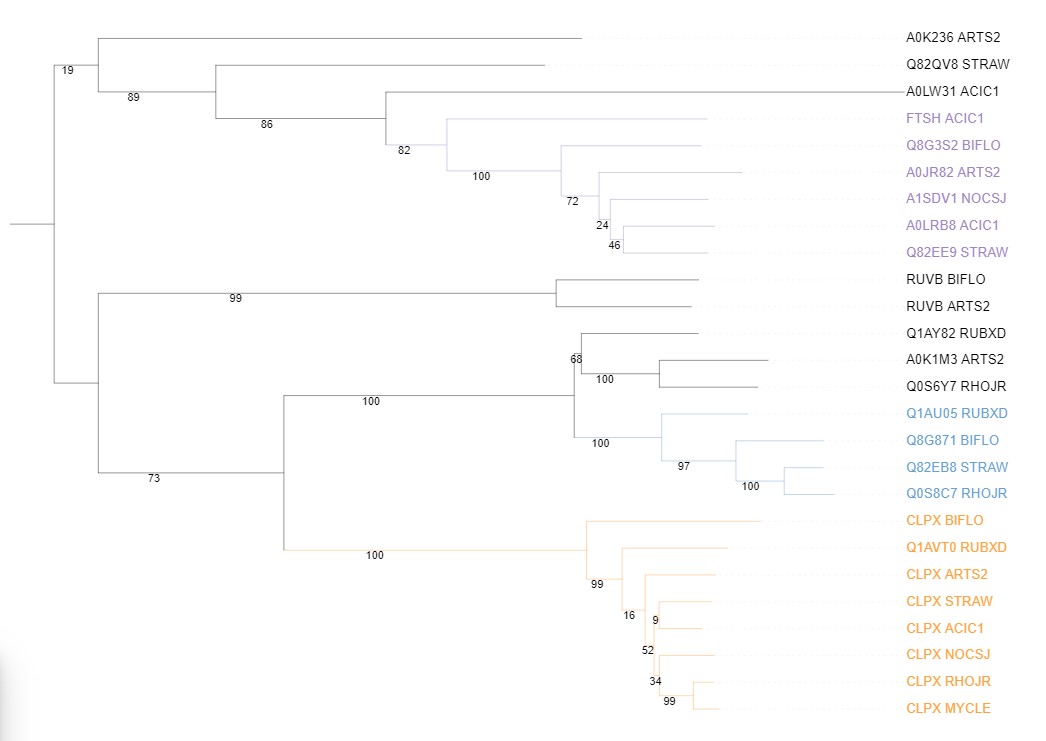
| Ортологи | Паралоги |
| RUVB BIFLO и RUVB ARTS2 | Q8G871 BIFLO и CLPX BIFLO |
| CLPX RHOJR и CLPX MYCLE | Q82EB8 STRAW и Q82QV8 STRAW |
| A0K1M3 ARTS2 и Q0S6Y7 RHOJR | FTSH ACIC1 и A0LRB8_ACIC1 |
Далее ортологичные группы были покрашены:
Затем ортологичные группы были схлопнуты:
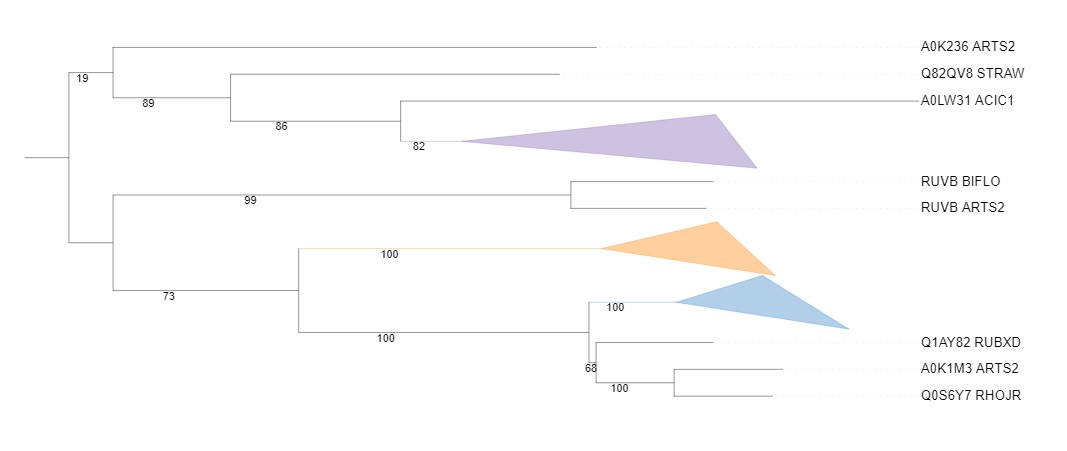
Фиолетовая клада содержит АТФ-зависимые цинковые протеазы, в ней отсутсвуют белки из бактерий MYCLE, RHOJR, RUBXD. В целом клада соответсвует филогенетическому дереву.
В кладу синего цвета входят функционально разные белки из бактерий RUBXD, BIFLO, STRAW, RHOJR. Веток, совпадающих с ветками дерева филогении бактерий нет.
Оранжевая клада содержит Clp протеазы, в ее состав вошли все бактерии. Как и для дерева филогении характерны ветки {RHOJR, MYCLE} и {STRAW, ACIC1}.