Практикум 7. Сигналы и мотивы - 2
Подготовка данных
Для выполнения практикума был выбрана референсная сборка GCF_000758725.1 генома Paenibacillus odorifer. С помощью сервиса Operon-mapper был получен список оперонов. Далее с использованием скрипта Георгия Муравьева был составлен список координат промоторов (промотором считалась область 100 нуклеотидов перед началом оперона) и получены три файла: для обучения, тестирования и негативного контроля.
Запуск MEME
Для запуска использовался локальный MEME:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50В результате было найдено 3 мотива:

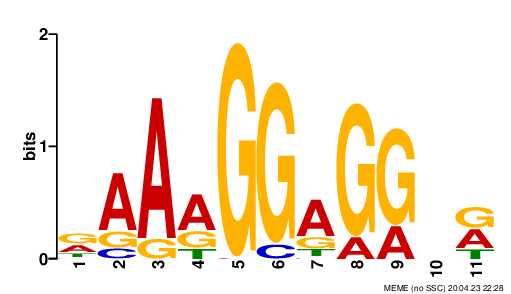
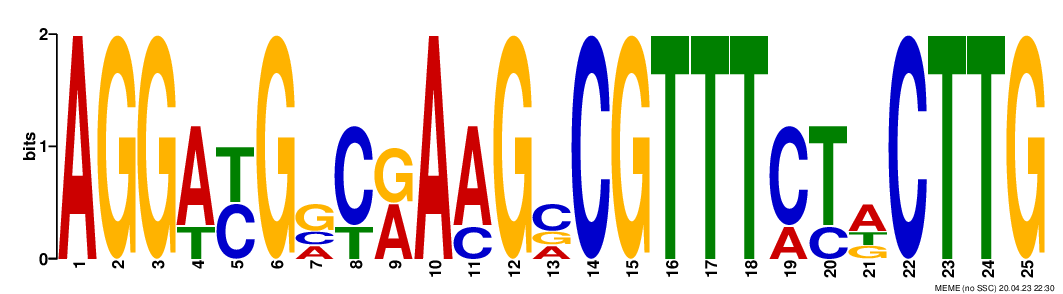
Для дальнейшей работы был выбран 1-й мотив, так как он похож на последовательность Шайна - Дальгарно и имеет минимальный e-value.
Поиск сигнала в материале для тестирования с помощью FIMO
С помощью FIMO мотив был найден в положительном и отрицательном контроле:
fimo --norc -motif HWTTCCTCCHYWW -thresh 0.001 meme.txt promotors.fasta
fimo --norc -motif HWTTCCTCCHYWW -thresh 0.001 meme.txt negative.fastaДля положительного было получено 2276 находок (37,6%), для отрицательного - 81 (15,7%). Видно, что в промоторной области данный мотив встречается чаще.