1. Консервативный мотив в выравнивании последовательностей гомологичных белков

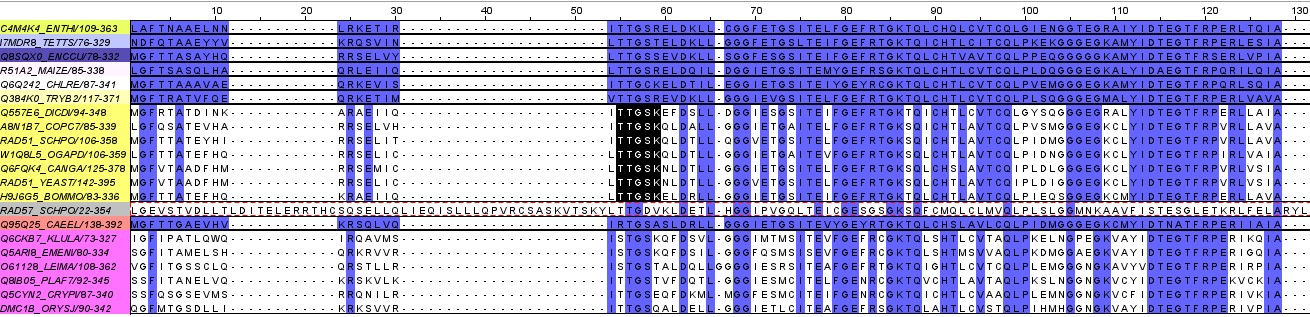
Seed содержит 21 последовательность. В выравнивании много консервативных участков, после покраски above identity threshold 100% для дальнейшей работы был выбран мотив GE.R[TSC]GK[ST]Q (Рис.1).
В выравнивании этот мотив встречается в 20 из 21 последовательности. С помощью сервиса MyHits был произведен поиск мотивов по базе данных SwissProt, было получено 25 находок. Все из них относятся к семейству гомологичных рекомбиназ, это говорит о том, что данный мотив действительно специфичен.
2. Мотив, специфичный для клады филогенетического дерева
С помощью метода NJ в JalView было построено филогенетическое дерево:
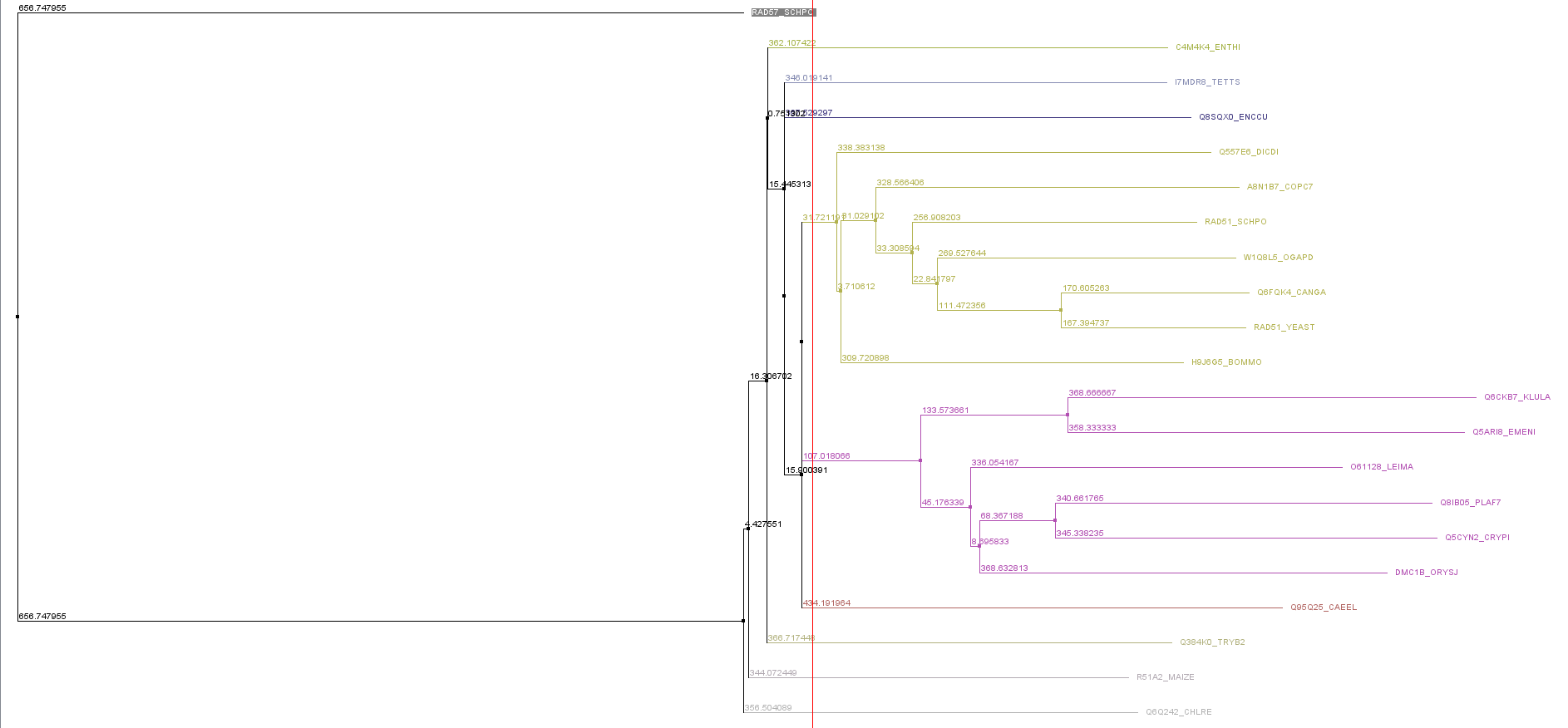
Была выбрана клада, показанная желтым цветом, она содержит 7 белков. Далее был найден характерный только для неё мотив: TTGSK.
3. PSI-BLAST
Для работы с PSI-BLAST был выбран белок с идентификатором Q7VDL2, принадлежащий бактерии Prochlorococcus marinus. Он является ингибитором клеточного деления.
Необходимое семейство было найдено после второй итерации. Добавленный после 4 итерации белок является лишним, так как разница между его e-value и e-value "лучшей" неправильной находки небольшая.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 146 | Q9AG20.1 | 0.005 | A8GFG7.1 | 0.005 |
| 2 | 188 | B6JKX0.1 | 7e-8 | - | - |
| 3 | 188 | Q9ZM51.1 | 2e-12 | A7H8E6.1 | 0.014 |
| 4 | 189 | A8MHK8.1 | 0.001 | A7H8E6.1 | 0.013 |