Мини-обзор генома и протеома бактерии Leptospira santarosai serovar Shermani str. LT 821
Суркова Милана Александровна
Факультет биоинженерии и биоинформатики, МГУ им. М.В. Ломоносова
Москва, Россия.
Аннотация
В мини-обзоре рассматривается бактерия Leptospira santarosai serovar Shermani str. LT 821, которая является патогенной спирохетой, вызывающей инфекционное заболевание лептоспироз. В результате обработки данных были получены результаты: гистограмма длин белков, информация о старт-кодонах в кодирующих последовательностях, диаграмма распределения кодонов для аминокислоты лейцин.
Введение
Leptospira santarosai serovar Shermani str. LT 821 принадлежит к домену Bacteria, типу Spirochaetes, классу Spirochaetia, порядку Leptospirales, семейству Leptospiraceae и роду Leptospira. Этот штамм представляет собой патогенную спирохету [1], вызывающую лептоспироз — зоонозное инфекционное заболевание, характеризующееся высокой температурой, лихорадкой, желтухой, кровотечением, почечной недостаточностью [2]. Геном бактерии состоит из двух кольцевых хромосом. Общий размер генома Leptospira santarosai serovar Shermani str. LT 821 составляет 3 983 611 пар оснований. В геноме идентифицировано 4 080 белок-кодирующих генов и 43 гена РНК [1].
Материалы и методы
С сервера NCBI были загружены: полная последовательность генома, последовательности всех белок-кодирующих участков, таблица локальных особенностей генома [3].
В Google Sheets была добавлена таблица с последовательностями всех белок-кодирующих участков. Из нее на другой лист была скопирована колонка с длинами генов. Затем была вычислена длина белка, закодированного в гене, с помощью формулы (дл.гена/3)-1 (см. листы cds и prot_lengths в сопроводительном файле 1). С использованием функций MIN и MAX были вычислена минимальная и максимальная длина белка. Была выбрана длина каждого кармана равная 50. В соседних колонках были взяты границы карманов с помощью Еi-1&"-"&Ei и были найдены числа длин белков, попадающих в карманы благодаря функции СЧЕТЕСЛИ(B:B,">="&E(i-1), B:B, "<"& Ei. На основании полученных данных была построена гистограмма длин белков с границами карманов по оси X и количеством подходящих белков по оси Y (см. лист prot_len_hist в сопроводительном файле 1).
С помощью функций Bash (grep -A 1 '^>' *cds*, grep -A 1 'pseudo=true' *cds*, grep -v '^>', grep -v '^-', cut -c1-3, sort, uniq -c) была извлечена информация о количестве всех старт-кодонов и количестве старт-кодонов псевдогенов (см. листы cds_all и cds_pseudo в сопроводительном файле 2). Далее была составлена таблица, в которой с помощью функций ЕСЛИОШИБКА(ВПР) были перенесены соответствующие кодонам значения и было посчитано количество “нормальных” старт-кодонов (см. лист all_startcodons в сопроводительном файле 2).
В сервисе Google Colab на языке программирования Python был написан код для подсчета частоты встречаемости кодонов для аминокислоты лейцин и была построена диаграмма с соответствующими кодонами, кодирующими лейцин, по оси X и частотой их встречаемости по оси Y (см. сопроводительный файл 3).
Результаты и Обсуждение
Длины белков, закодированных в геноме бактерии Leptospira santarosai serovar Shermani str. LT 821
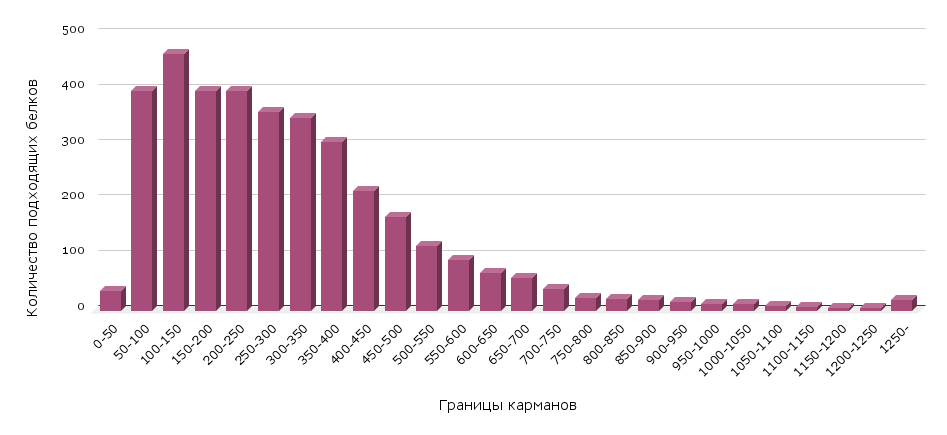
По полученным данным (см. Рисунок 1) можно сделать вывод, что наиболее частые длины белков находятся в диапазонах от 100 до 300 аминокислот. Пик приходится на интервал 100-150 аминокислот. После значения длины, равного 50, происходит резкий скачок. После диапазона 300-350 аминокислот происходит постепенное снижение числа белков с увеличением их длины. Белки с длиной более 1000 аминокислот встречаются редко. Результаты являются нормальными для прокариотического организма. Большинство белков у бактерий имеют длину 100-300 аминокислот, что соответствует основным функциональным белкам. Короткие белки могут быть регуляторными элементами или сигнальными пептидами. Длинные белки встречаются редко, потому что они выполняют структурные и ферментативные функции, требующие несколько функциональных доменов.
Старт-кодоны в кодирующих последовательностях
Из таблицы старт-кодонов в кодирующих последовательностях (см. лист all_startcodons в сопроводительном файле 2) видно, что наибольшее число раз во всех кодирующих последовательностях, а именно 2897, встречается кодон ATG, кодирующий аминокислоту метионин. В “нормальных” генах - в 2843 случаях, в псевдогенах - в 54. Следовательно, ATG является основным старт-кодоном. Этот кодон является самым распространенным для инициации трансляции у эукариот и прокариот. Альтернативным старт-кодоном является GTG, встречающийся 193 раза. ATA, ATT, ATC - редкие альтернативные кодоны, так как появляются гораздо реже (16 - 21 раз). Встречаются кодоны, появляющиеся исключительно в псевдогенах (AAT, AAA, AAG, GAC и т. д.). Это значит, что такие старт-кодоны могли появиться вследствие мутаций или в результате ошибок при секвенировании. Такие кодоны, как AGG, AGA, AGC, AAC появляются в 0 % случаев, поэтому они не могут быть старт-кодонами.
Анализ кодонов для аминокислоты лейцин
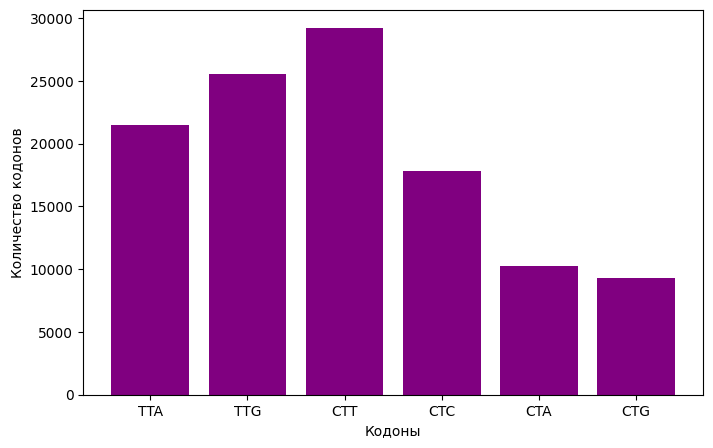
На основе полученных данных (см. Рисунок 2) можно сделать вывод, что CTT является наиболее частым кодоном, встречающимся в геноме Leptospira santarosai serovar Shermani str. LT 821 (встречается 29216 раз). Также довольно часто встречаются кодоны TTA (21475 раз) и TTG (25557 раз). Кодоны CTA и CTG встречаются реже (10272 и 9340 раз). Это может означать, что наиболее оптимизированными для экспрессии белков кодонами являются CTT, TTG, TTA и что кодоны CTA, CTG имеют меньшую функциональную значимость. Такая адаптация у бактерии к использованию определенных кодонов для аминокислоты лейцин может повышать эффективность синтеза белков.
Благодарности
Выражаю благодарность Русинову Ивану Сергеевичу за научное руководство в написании мини-обзора.
Сопроводительные материалы
1.Таблицы с данными для построения гистограммы и сама гистограмма
3. Код Python для подсчета количества кодонов аминокислоты лейцин и для диаграммы распределения кодонов
Список литературы
1. GENOME: Leptospira santarosai. Kyoto Encyclopedia of Genes and Genomes ( KEGG ). Chou LF, Chen TW, Ko YC, Pan MJ, Tian YC, Chiu CH, Tang P, Hung CC, Yang CW. 2015.
2. Li-Fang Chou, Ting-Wen Chen, Yi-Ching Ko, Ming-Jeng Pan, Ya-Chung Tian, Cheng-Hsun Chiu, Petrus Tang, Cheng-Chieh Hung and Chih-Wei Yang. Potential impact on kidney infection: a whole-genome analysis of Leptospira santarosai serovar Shermani. Emerging Microbes & Infections. November 2014. PMID: 26038504. PMCID: PMC4274889. DOI: 10.1038/emi.2014.78.
3. National Center for Biotechnology Information ( NCBI ).