Практикум 8 и 10. UniProt
Милейко Пётр Павлович
Введение
Информация о белке
Информация об организме
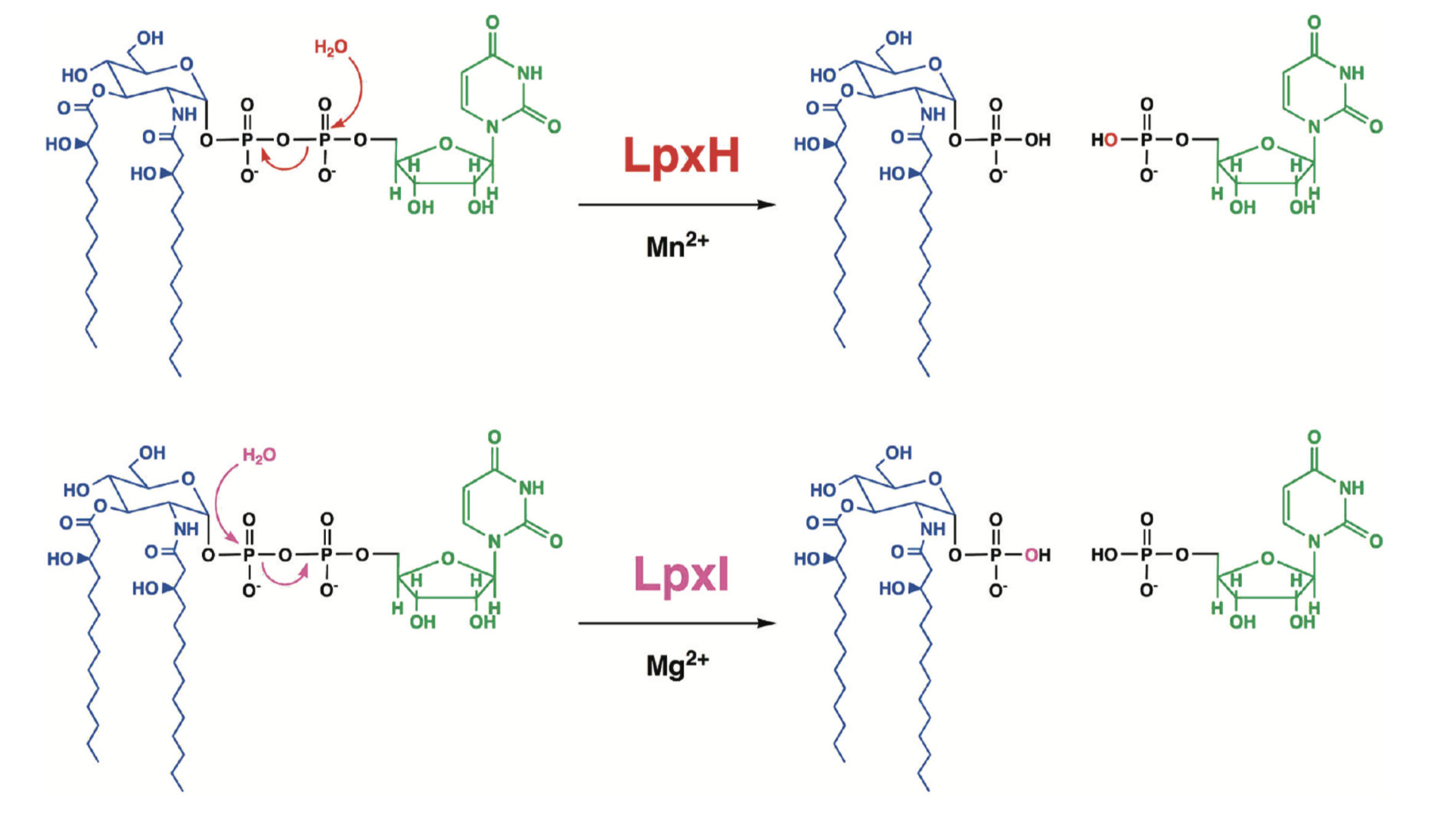
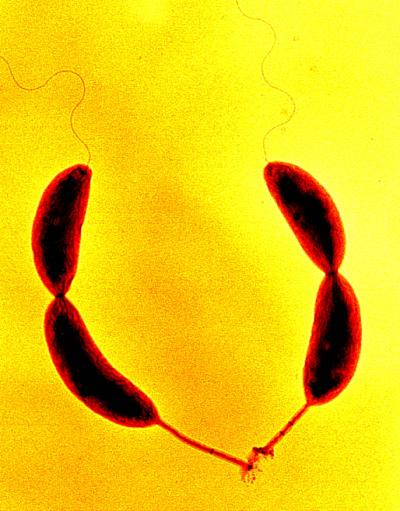
Caulobacter crescentus, или caulobacter vibrioides грамотрицательная олиготрофная бактерия, обитающая в пресноводных реках и озерах. На примере Caulobacter crescentus изучают ассиметрическое деление и клеточную регуляцию жизненного цикла прокариотических клеток из за ряда преимуществ данной бактерии при лабораторных исследованиях, одно из таких преимуществ это способностьб бактерии жить в средах с малым количеством питательных веществ. Именно из данной бактерии была впервые получена UDP-2,3-диацилглюкозамин гидролаза. Данный белок закодирован в гене LpxI.
Общая информация о белке из UniProt
| Параметр | Результат |
|---|---|
| RecName | UDP-2,3-diacylglucosamine pyrophosphatase |
| ID | LPXI_CAUVC |
| AC | Q9A716 |
| EC | 3.6.1.54 |
| PDB IDs | 4GGM, 4J6E |
| EMBL ID | AE005673 |
| Mass | 29634 MW |
| Length | 280 АА |
Сеансы поиска в UniProt
| Номер запроса | Запрос | Результат |
|---|---|---|
| 1 | name:"udp-2 3-diacylglucosamine pyrophosphatase" NOT organism:metagenome taxonomy:"Bacteria [2] | 1761 (Swiss-Prot+TrEMBL) |
| 2 | name:"udp-2 3-diacylglucosamine pyrophosphatase" NOT organism:metagenome NOT taxonomy:"Bacteria [2]" | 5 (TrEMBL) |
| 3 | name:"udp-2 3-diacylglucosamine pyrophosphatase" NOT organism:metagenome NOT taxonomy:bacteria NOT taxonomy:"Archaea (9ARCH) [2157] 0 | 0 |
| 4 | name:"udp-2 3-diacylglucosamine pyrophosphatase" NOT organism:metagenome taxonomy:"Proteobacteria [1224] | 1216 (Swiss-Prot+TrEMBL) |
| 5 | name:"udp-2 3-diacylglucosamine pyrophosphatase" NOT organism:metagenome gene:lpxi | 22 (Swiss-Prot+TrEMBL) |
| 6 | name:"udp-2 3-diacylglucosamine pyrophosphatase" NOT organism:metagenome gene:lpxi NOT taxonomy:"Proteobacteria [1224]" | 2 (TrEMBL) |
Вывод: С помощью расширенного поиска по UniProt удалось узнать что UDP-2,3-диацилглюкозамин гидролаза присутствует только у бактерий и архей. При этом большая часть этих организмов является протеобактериями. Также удалось выяснить что ген LpXi, в котором закодирована информация о белке, характерен не только для Caulobacter crescentus, но и для некоторых других бактерий.
Формат записи
- FT
Строки Feature Table дают краткую информацию об области или участке, представляющие интерес в последовательности белка. Первое слово в строке обозначает тип информации. Далее идут номера аминокислотных остатков. Под номерами может находится краткая заметка, описывающая чем интересен данный участок белка. Под заметкой находится ссылка на источник. Так интересным мне показалась данная информация:

Изучив литературу, я нашел фрагмент в котором рассказывалось, о том что ученые меняли некоторые АО на аланин и следили за изменением каталитической активностью фермента, при этом полное ее исчезновения произошло после замены ASP225 на аланин, что и подтверждается строками FT.
Анализ гомологичных последовательностей с помощью UniRef
- UniRef100_Q9A716
- UniRef90_Q9A716
- UniRef50_Q9A716
В данном кластере присутствуют 3 записи о белке UDP-2,3-diacylglucosamine pyrophosphatase LpxI у 2-х различных штаммов Caulobacter vibrioides. "Representative" является изучаемая последовательность. От сюда мы можем сделать вывод, что данная последовательность является уникальной для данного вида.
В данном кластере находятся 7 записей. Изучаемая запись белока также как и в UniRef100 является репрезентативной последовательностью. Также данные варианты последовательности белока присутствуют у 3-х различных штаммов Caulobacter vibrioides.
В этом кластере находятся 179 записей. Изучаемая последовательность также является репрезентативной. Из записей можно узнать, что данные последовательности белка принадлежат определенным 5-ти родам семейств Caulobacteraceae, Hyphomonadaceae, Rhizobiaceae. Все данные семейства относятся к классу альфа-протеобактерии. Следовательно мы можем сделать вывод, что данные схожие последовательности белков представлены в малом количестве.
Сравнение протеомов
Как говорилось выше Caulobacter crescentus пресноводных водоемах, поэтому было бы интересно сравнить ее протеом с протеомом близкородственной бактерии Asticcacaulis excentricus, которая обитает в морской среде, ведь значительное отличие в протеоме возможно будет связана с белками, которые обеспечивают адаптацию к соленой воде. Как видно из таблицы 3, я проверил бактерию на количество транслоказ, затем на количество транслоказ, отвечающих за перенос через мембрану катионов, анионов и хелатов. К сожалению результаты были почти идентичными (также стоит отметить, что общее количество белков мало отличается, что говорит о близкородственности бактерий). Поэтому я решил посмотреть количество ферментов каждого класса. В таблице 4 представлены результаты поиска. Как мы видим, все классы белков Caulobacter crescentus количественно мало отличаются от белков Asticcacaulis excentricus, кроме количества гидролаз. Для дальнейшего исследования я использовал Excel. Я скачал все названия гидролаз Caulobacter crescentus и Asticcacaulis excentricus и с помощью опции "условное форматирование -> правила выделения ячеек" сравнил состав гидролаз бактерий. В итоге получилось что 42 фермента Asticcacaulis excentricus не совпадают с ферментами Caulobacter crescentus. Caulobacter crescentus имеет 26 ферментов отличных от гидролаз другой бактерии. Данные ферменты никак не связаны с приспособлением к морской среде. Соответсвенно связь ферментов и среды обитания не очевидна.
| Организм | Proteome ID | Количество белков | Количество белков в базе Swiss-Pro | Количество трансмембранных белков | Количество ферментов | Транслоказа | EC 7.2 | EC 7.3 |
|---|---|---|---|---|---|---|---|---|
| Caulobacter vibrioides (strain ATCC 19089 / CB15) | UP000001816 | 3,720 | 573 (15,4%) | 664 | 686 | 31 | 1 | 3 |
| Asticcacaulis excentricus (strain ATCC 15261 / DSM 4724 / VKM B-1370 / CB 48) | UP000001492 | 3,743 | 4 (0.1%) | 721 | 750 | 31 | 1 | 2 |
| Организм | Оксидоредуктазы | Трансферазы | Гидролазы | Лиазы | Изомеразы | Лигазы |
|---|---|---|---|---|---|---|
| Caulobacter vibrioides (strain ATCC 19089 / CB15) | 100 | 264 | 137 | 66 | 46 | 61 |
| Asticcacaulis excentricus (strain ATCC 15261 / DSM 4724 / VKM B-1370 / CB 48) | 104 | 259 | 186 | 76 | 42 | 66 |
Список литературы
- Louis E. Metzger IV and Christian R. H. Raetz. An Alternative Route for UDP-Diacylglucosamine Hydrolysis in Bacterial Lipid A Biosynthesis. // Biochemistry, Vol. 49, No. 31, 2010, p. 67156726. https://pubs.acs.org/doi/10.1021/bi1008744
- Louis E Metzger IV, John K Lee, Janet S Finer-Moore1, Christian R H Raetz & Robert M Stroud. LpxI structures reveal how a lipid A precursor is synthesized. // Nature Structural & Molecular Biology Vol. 19, p. 11321138(2012) https://www.nature.com/articles/nsmb.2393
- Информация о белке UniProt
- Velocity Hughes, Chao Jiang, Yves Brun. Current Biology. Vol. 22, Issue 13, p1155-1266 ссылка на статью