Для определения функции нуклеотидной последовательности полученной при расшифровки хроматограммы в практикуме 6, и для нахождения таксономического положения организма, геному которого она принадлежала, я использовал blastn, так как мы не знаем кодирует ли эта последовательность какой-нибудь белок. Для поиска был выбран алгоритм megablast со стандартными параметрами, чтобы сразу найти близкие гомологи. С выдачей можно ознакомиться по ссылке. Все 100 находок указывают на то, что данная нуклеотидная последовательность скорее всего кодирует субъединицу I цитохром с-оксидазу (COI). Первые 13 лучших находок (имеющих процент идентичности >90%) принадлежат Polycirrus, что говорит нам о том, что моя последовательность вероятнее всего принадлежит данном роду. Данная последовательность наиболее близка к последовательностям Polycirrus medusa и нескольких неклассифицированных организмов. Для более точного определения вида изучаемая последовательность была выравнена с помощью алгоритма muscle в JalView на последовательности находок. Скачать выравнивание можно по ссылке. Множественное выравнивание привело меня к выводу о том, что скорее всего исследуемая последовательность принадлежит Polycirrus medusa. Особенно это заметно на участке на рис.1. На позициях 261, 268, 273, 297 нуклеотиды исследуемой последовательности совпадают с нуклеотидами только Polycirrus medusa, отсюда можно сделать вывод о том, что последовательность скорее всего была получена из генома Polycirrus medusa.

Для нахождения гена был выбран контиг (ссылка на NCBI) длиною 21,763 нуклеотидов серого волка Canis lupus. Файл с последовательностью контига доступен по ссылке. Так как надо найти белок по нуклеотидной последовательности, был использован алгоритм blastx. Белок искался по базе RefSeq, настройки были выставлены по умолчанию кроме word-size – 6, а также из поиска был исключен поиск по белкам Canis lupus. С результатами выдачи можно ознакомиться по ссылке. Как видно из выдачи, контиг скорее всего содержит ген кодирующий белок WD repeat-containing protein 37, так как процент индентичности в обоих случаях выше 85 процентов, а значение e-value очень маленькое.
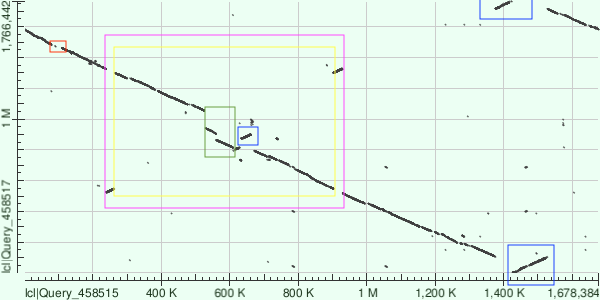
Для данного задания была выбрана пара бактерий, относящихся к одному роду: Campylobacter coli и Campylobacter jejuni. Сборки полных геномов (хромосомы и плазмиды) были найдены на NCBI Genome, но для построения карты локального сходства были взяты последовательности хромосом (NZ_CP046317 и NZ_LN831025). Для того что бы получить карту локального сходства был использован blastn с алгоритмом megablast с параметрами по умолчанию (рис.2) Как видно из карты локального сходства (рис. 2ß) обе хромосомы сильно похожи, на это указывают и показатели идентичности (85,78) и покрытия (83%), которые выдал мне blastn. Тем не менее на карте видны крупные хромосомные перестройки: