Ссылки на скачивание исходных .ab1 файлов: прямая цепочка и обратная цепочка
Для получения консенсуса были осуществлены следующие действия:
- Сначала стоит определить нечитаемые участки хроматограмм.
- У прямой последовательности: 5'-1..10-3' и 5'-657..700-3'
- У обратной последовательности: 3'-1..33-5'; 3'-680..721-5'
- С помощью программы Ugene были получены последовательности обратной и прямой цепочек.
- Последовательность обратной цепи была перевернута и для нее же была найдена комплементарная последовательность (с помощью Ugene).
- Теперь с помощью Jalview были выравнены две последовательности (прямая и comlement+reverse) и в результате сборки был получен контиг, который мы будем использовать в качестве референсной последовательности: ссылка на выравнивание
Выравняем хроматограммы обратной и прямой цепочек на контиг (контиг берем в качестве референсной последовательности) в Ugene и получим консенсусную последовательность, которую надо отредактировать в виду наличия проблемных нуклеотидов на хроматограммах цепочек. (ссылка на выравнивание прямой, обратной последовательностей и референса)
- Ugene удалила нечитаемые участки прямой и обратной последовательности, но учитывать я буду участки которые определил в пункте 1. (если рассматривать координты которые были закреплены за нуклеотидами в Ugene, то я вырезал позиции 1-42 и ):
- У прямой: 5'-1..10 -3'; 5'-692..700-3'
- У обратной: 3'-1..33-5'; 3'-715..721-5'
- Отредактировав проблемные нуклеотиды в последовательностях и удалив нечитаемые участки, я получил итоговую консенсусную последовательность. (ссылка на консенсус и ссылка на исправленные прямую, обратную последовательности и референс без удаления концов)
Анализ проблемных нуклеотидов.
В данном примере программа правильно определила сложный участок. На хроматограмме прямой цепи видно пятно краски. То что на позиции 110 и 113 присутствуют именно тимин и гуанин подтверждает и хроматограмма обратной последовательности.
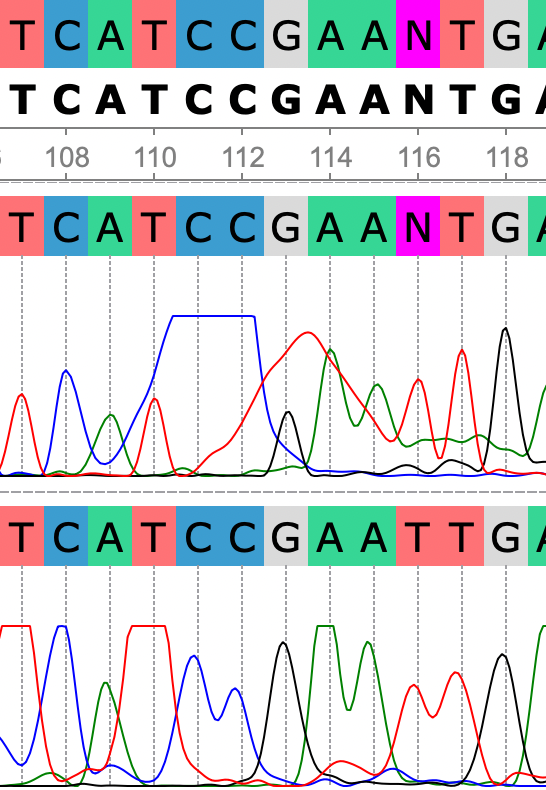
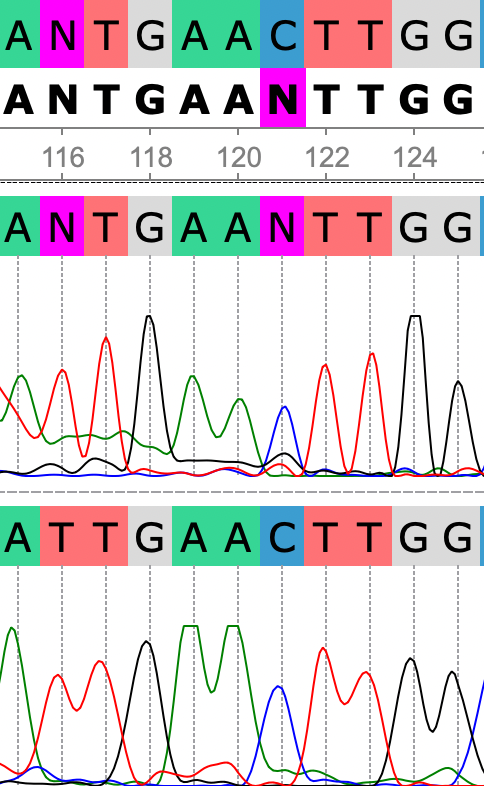
Из-за большого количества шумов программа не смогла определить нуклеотиды в 116 и 121 позициях. Но так как сигналы тимина и цитозина довольно таки выражены, и средняя высота шумов не превышает и половины пиков, можно сделать вывод, что в 116-тимин а в 121-цитозин. Также данное удтверждение подтверждают выраженные пики тимина игуанина на этих позициях на хроматограмме обратной последовательности.
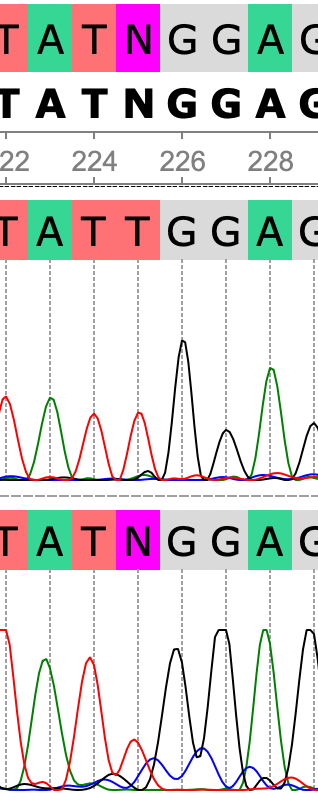
В данном случае на рис. 3 можно было бы предположить что в 225 позиции присутствует полиморфизм, так как высота пика цитозина превышает половину высоты пика тимина. Но если рассматривать область 224-228, то видно, что средняя высота шумов примерно равна данном 'цитозиновому пику'. При этом стоит отметить выраженный сигнал тимина на хроматограмме прямой цепи. Из всего вышесказанного можно сделать вывод, что на проблемной позиции находится тимин. Меняем N на T.
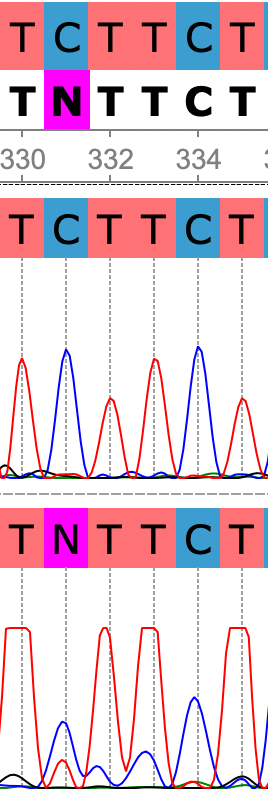
В данном случае (рис.4) в 331 позиции на хроматограмме обратной цепи также можно подумать, что мы имеем дело с полиморфизмом. Но в близких к 331 позициях также присутствует шум, также как и в предыдущем примере на прямой цепи нет шумов и присутствует выраженный сигнал тимина: делаем вывод, что в данной позиции находится тимин, меняем N на T.
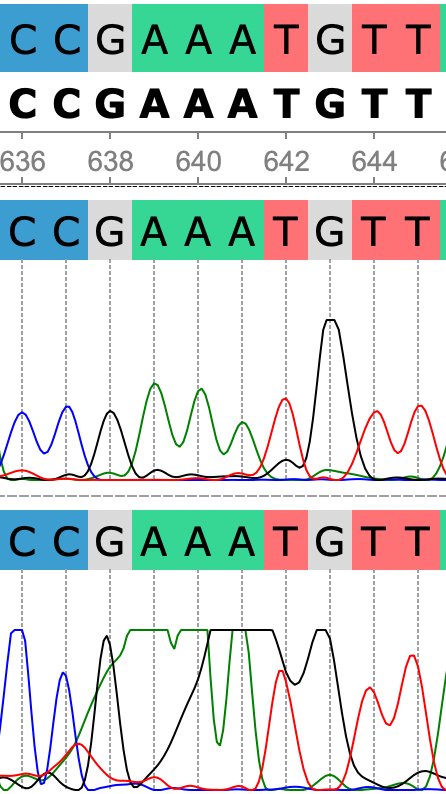
На рис. 5 продемонстрирован еще один пример пятна краски по причине ошибки фореза. К счастью программа правильно разобралась с определением нуклетидов на 638-642 позициях.
Характеристика хроматограммы
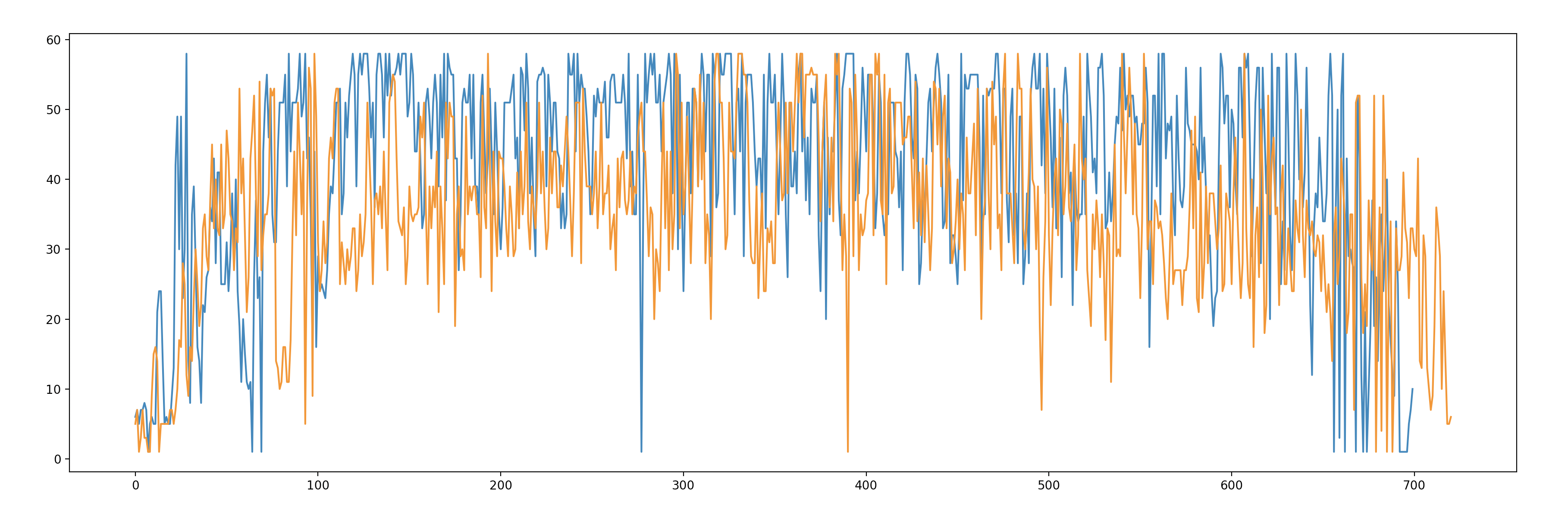
В целом хроматограммы довольно качественные. Нечитаемые участки (как и ожидалось) находятся в начале и в конце хроматограмм. В основном проблемные участки возникают из-за повышения уровня шума, но таких участков мало. Также имеются пятна краски из за ошибок при форезе, но их всего две. Качество прочтения отображено на рис.6.
Нечитаемый фрагмент хроматограммы
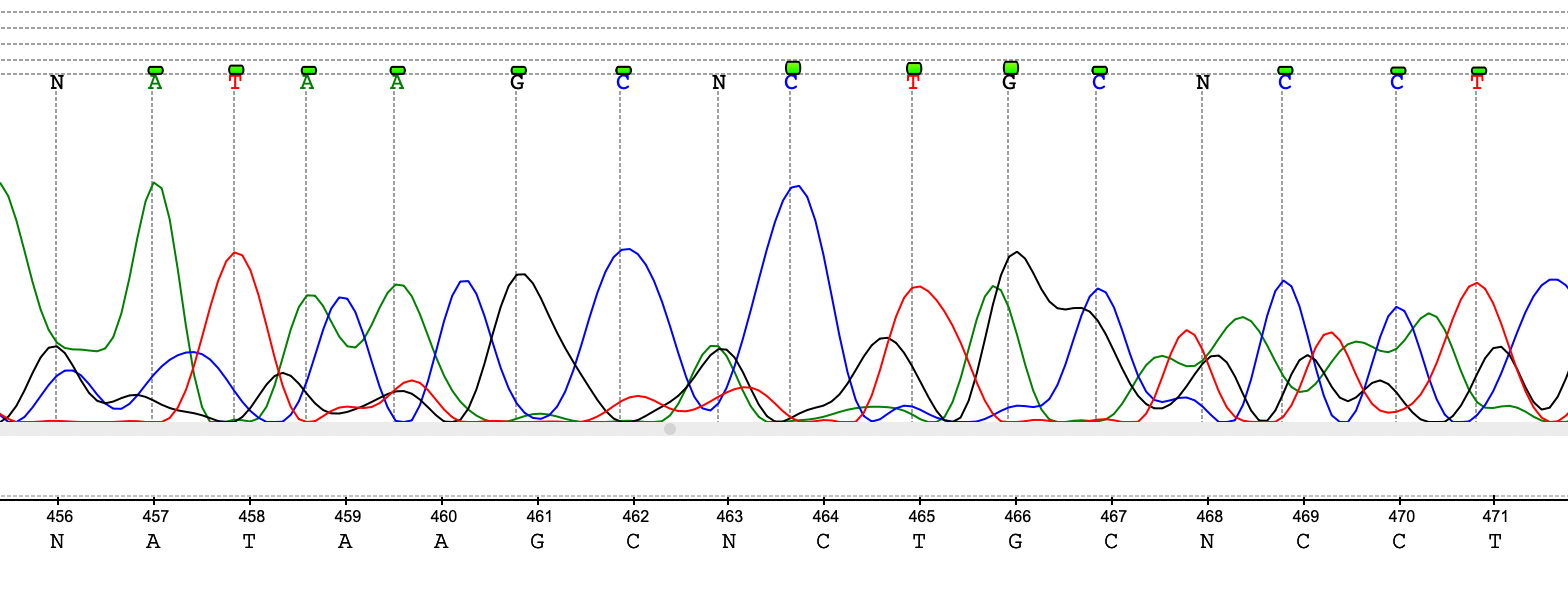
Для примера был взят файл kamp3_18SIII_F_F03_WSBS-Seq-1-08-15.ab1, был рассмотрен участок (хотя на самом деле плохо на протяжении всей хроматограммы) 456-471. Как видно из рис. 7 в данной хромотограмме каждому нуклеотиду соотвествует несколько пиков при этом расстояние между соседними пиками все время разное. Скорее всего при секвенировани в одном препарате присутствовало несколько фрагментов ДНК.