Знакомство с базой данных OPM.
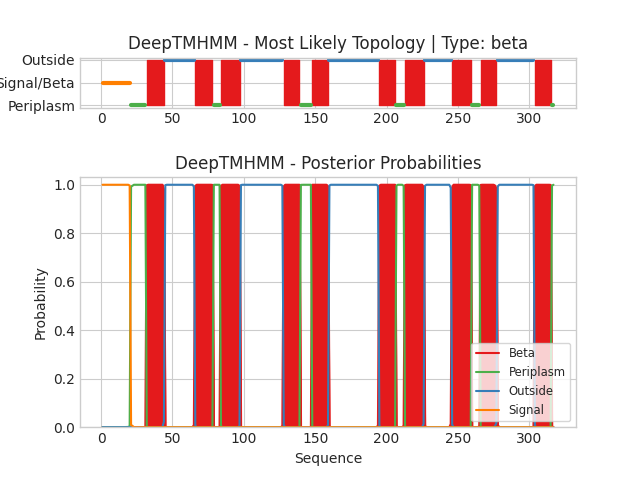
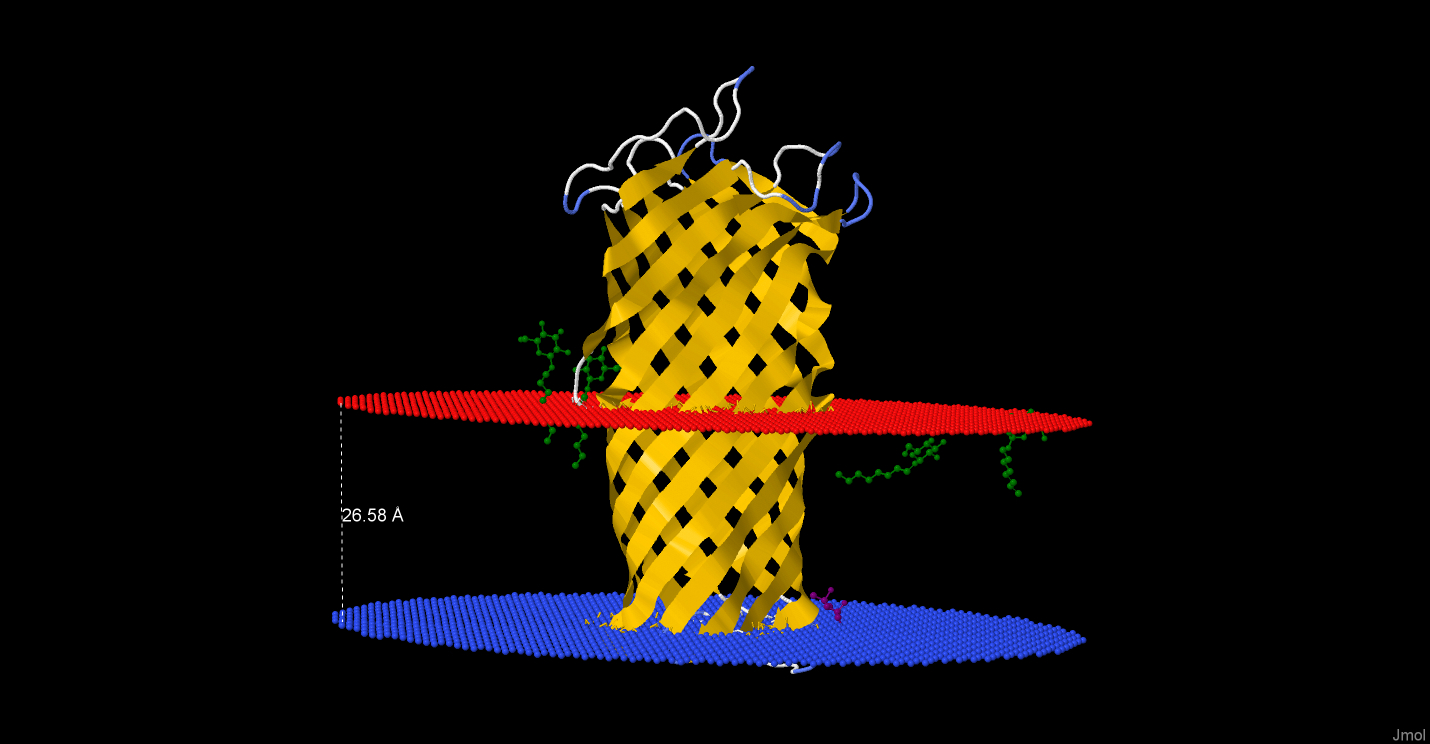
В базе данных OPM был выбран трансмембранный белок Outer membrane protease OmpT - протеаза внешней мембраны OmpT, принадлежащая Escherichia coli (PDB ID: 1i78 , Uniprot ID: OMPT_ECOLI). Часть белка, погруженная в мембрану, представляет собой β-бочонок (рис. 1). Красным цветом на рисунке 1 показана положительно заряженная часть мембраны (обращена наружу), синим цветом показана отрицательно заряженная часть мембраны (обращена в сторону периплазматического пространства). Данный белок разрезает полипептиды между положительными аминокислотными остатками (Arg, Lys, His). Некоторые характеристики белка из OPM представлены в таблице 1.
| Параметр | Значение |
|---|---|
| Толщина гидрофобной части белка в мембране | 26.5 Å |
| Координаты трансмембранных участков | 1(11-20),2(50-61),3(64-72),4(111-121),5(126-136),6(178-187),7(192-201),8(230-240),9(245-254),10(287-296) |
| Средняя длина трансмембранного сегмента | 10.4 |
| Местонахождение | Наружная мембрана грамотрицательной бактерии |