Код доступа проекта по секвенированию бактерии Buchnera aphidicola str. Tuc7: SRR4240360. Чтения были скачаны в папку /mnt/scratch/NGS/missshults12 на kodomo за счёт команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz
Сначала был создан файл со всеми адаптерами с помощью команды:
cat /mnt/scratch/NGS/adapters/*.fa adapters.fa
Далее была запущена программа trimmomatic для удаления возможных остатков адаптеров в чтениях:
TrimmomaticSE -phred33 SRR4240360.fastq.gz trimSRR4240360.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7
Результат работы команды:
Input Reads: 8254632 Surviving: 8212774 (99.49%) Dropped: 41858 (0.51%)
Видим, что было удалено 41858 чтений из 8254632 (файл весит 193 Мбайта, в то время как исходный - 194 Мбайта).
Далее с помощью той же программы trimmomatic с правых концов чтений были удалены нуклеотиды с качеством ниже 20 и оставлены чтения, длина которых не меньше 32 нуклеотидов:
TrimmomaticSE -phred33 trimSRR4240360.fastq.gz finalSRR4240360.fastq.gz TRAILING:20 MINLEN:32
Результат работы команды:
Input Reads: 8212774 Surviving: 7915474 (96.38%) Dropped: 297300 (3.62%)
Было удалено 297300 чтения из 8212774.
В итоге, файл стал весить 184 Мбайта (исходный файл весил 194 Мбайта).
С помощью программы velveth были созданы k-меры длины k=31:
velveth . 31 -short -fastq finalSRR4240360.fastq.gz
Cборка генома программой velvetg на основе k-меров с помощью команды:
velveth .
В результате сборки генома получили: N50 = 43070.
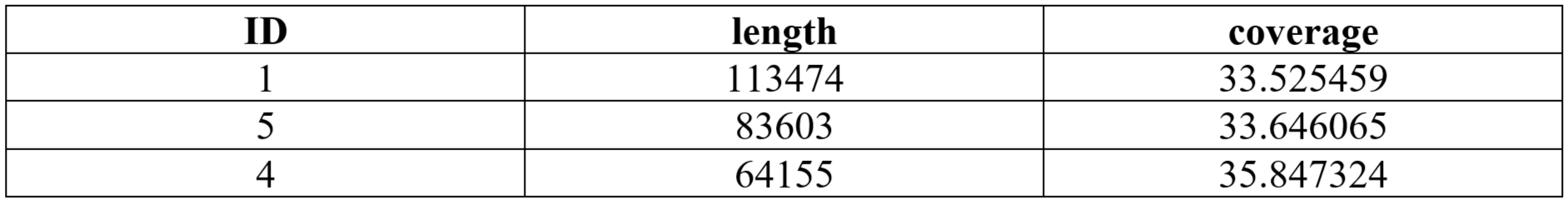
В файле contigs.fa, полученном в результате сборки генома, ищем контиги с наибольшими длинными:
grep '^>' contigs.fa | cut -f2,4,6 -d '_' | sort -k2 -t '_' -V -r | less

Найти данные контиги также можно в файле stats.txt с помощью Google-таблиц.
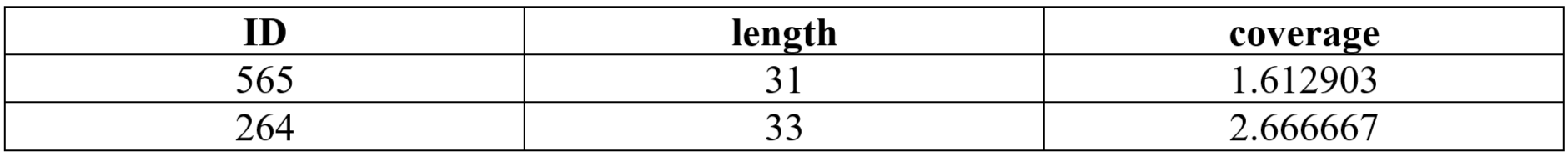
В файле contigs.fa ищем контиги с аномально большим покрытием:
grep '^>' contigs.fa | cut -f2,4,6 -d '_' | sort -k3 -t '_' -V -r | less

В файле contigs.fa также ищем контиги с аномально малым покрытием:
grep '^>' contigs.fa | cut -f2,4,6 -d '_' | sort -k3 -t '_' -V | less
Самые длинные контиги были выделены в отдельные файлы, после чего программой megablast каждый из трёх контигов был сравнён с хромосомой Buchnera aphidicola (GenBank/EMBL AC - CP009253).
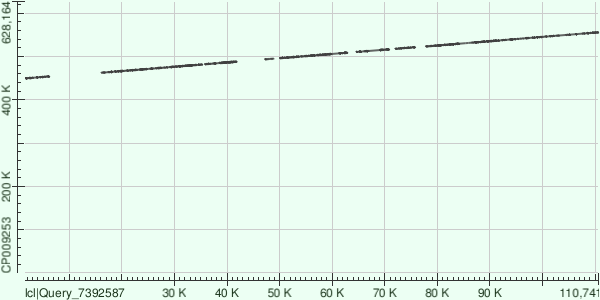
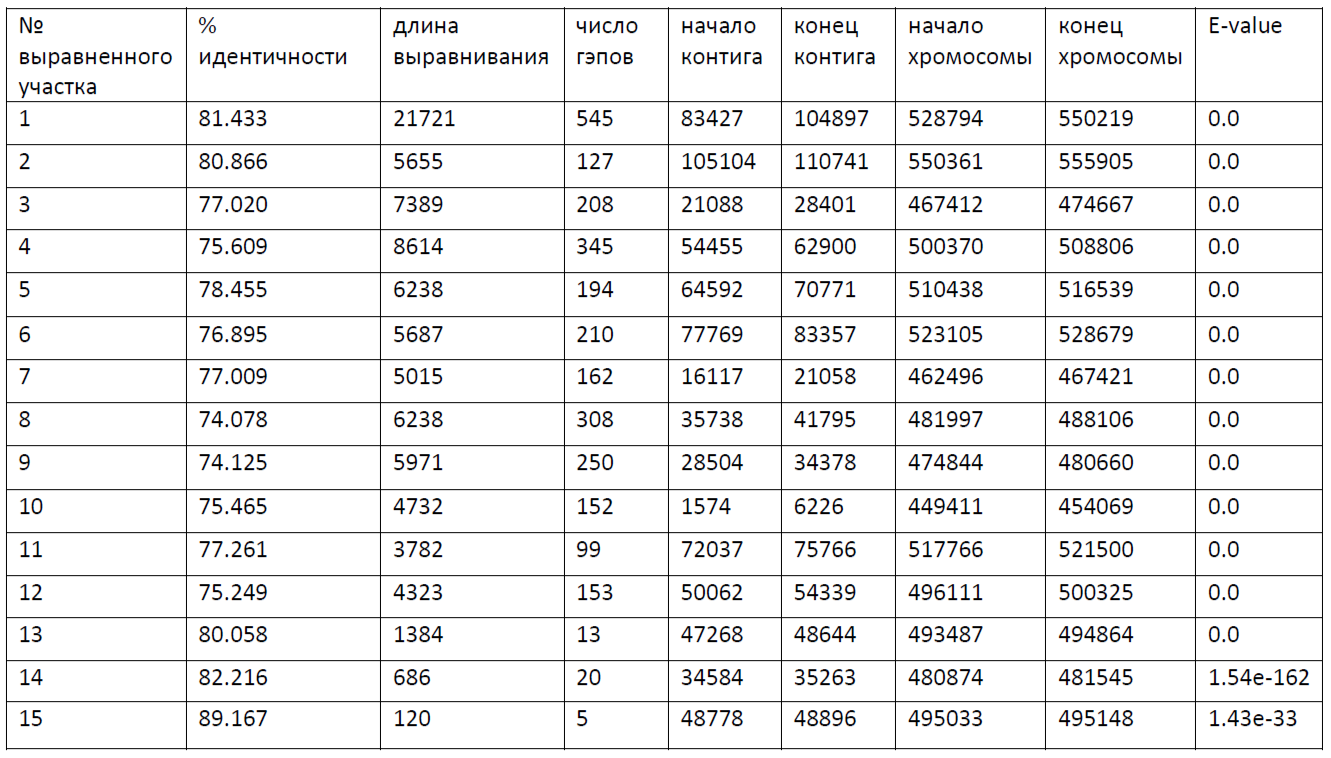
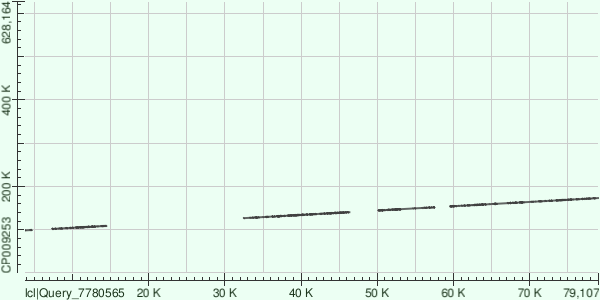
Начало участка хромосомы, соответствующего контигу - 495033; конец участка хромосомы, соответствующего контигу - 550219. Контиг ложится на хромосому 15 разными участками.
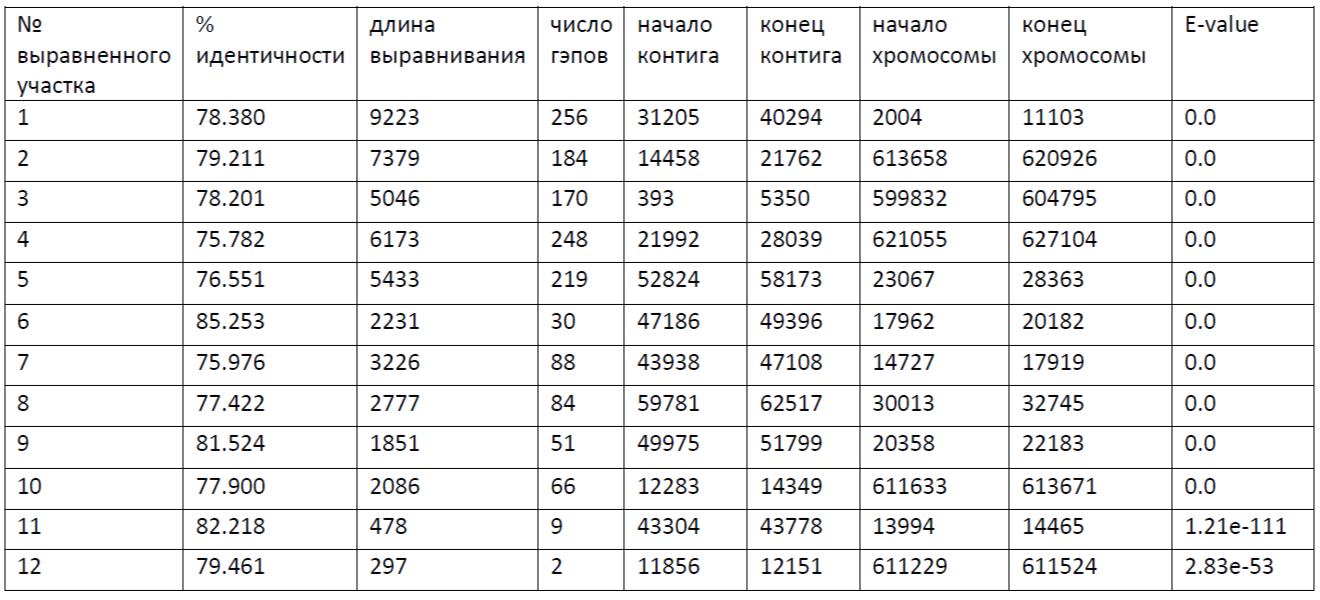
Краткие результаты по картированию приведены в таблице:
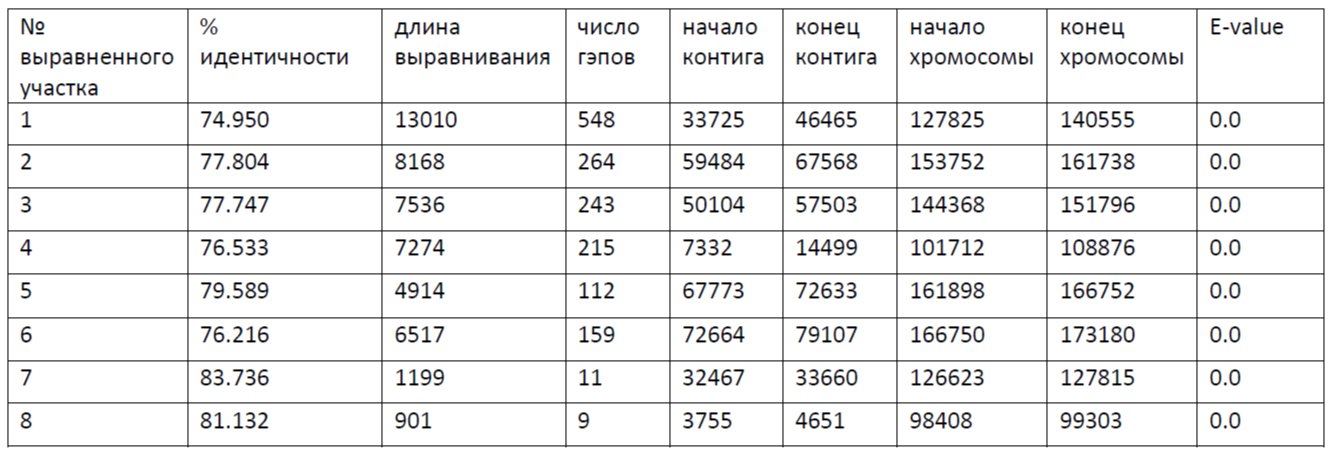
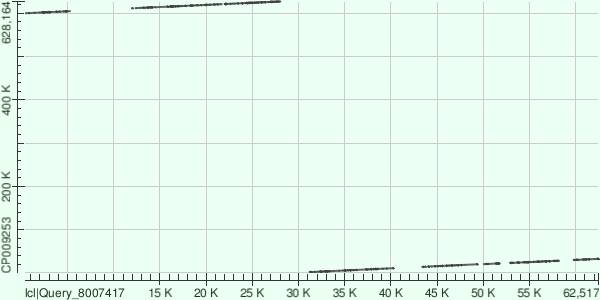
Начало участка хромосомы, соответствующего контигу - 98408; конец участка хромосомы, соответствующего контигу - 140555. Контиг ложится на хромосому 8 разными участками.
Краткие результаты по картированию приведены в таблице:
Начало участка хромосомы, соответствующего контигу - 2004; конец участка хромосомы, соответствующего контигу - 620926. Контиг ложится на хромосому 12 разными участками.
Краткие результаты по картированию приведены в таблице: