Для работы в данном практикуме был взят домен Hyaluronan / mRNA binding family (AC в базе данных Pfam PF04774). К этому домену относятся гиалуронан-связывающий белок HABP4 и мРНК-связывающий белок PAI-1. Было замечено, что HABP4 связывает гиалуронан, но неизвестно, является ли это его основной функцией in vivo. Также было замечено, что он связывает РНК, но с меньшим сродством, чем гиалуронан. Белок, связывающий мРНК, PAI-1, специфически связывается с мРНК ингибитора активатора плазминогена 1-го типа (PAI-1) и, как считается, участвует в регуляции стабильности мРНК.
В выравнивании seed у домена 67 последовательности. Идентичных последовательностей в выравнивании не оказалось, но было 3 очень похожих последовательности, которые затем были удалены. В итоге, осталось 64 последовательности.
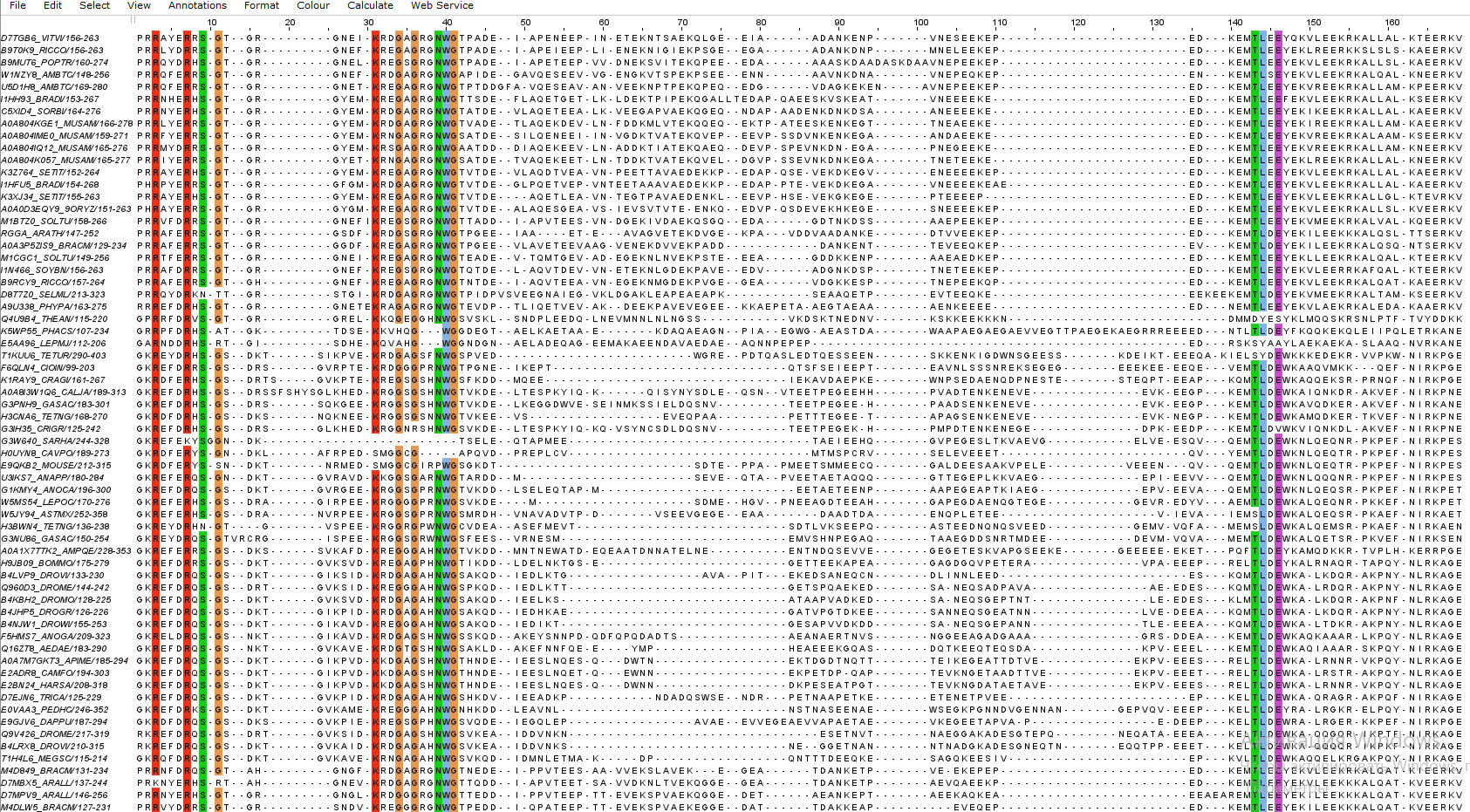
Далее применила окраску Clustal и выставила параметр Above identity threshold, равный 100%, но в результате этого ничего окрасилось, поэтому начала снижать Above identity threshold. На 95% появились окрашенные колонки, но они находились довольно далеко друг от друга, поэтому продолжила снижать параметр и остановилась на 90%.

Выделилось 3 мотива, информационное содержание IC каждого достаточно хорошее. Я решила остановиться на втором мотиве (на второй группе выделенных колонок), так как он достаточно длинный, что повышает вероятность того, что он вряд ли будет найден в случайных (то есть не относящихся к данному домену) последовательностях. Соотвественно смогла составить следующий паттерн мотива [KS][RKMQ].[GAH].[GR].{2}[NP]WG, по которому программа находит мотив в 60 последовательностях. Мотив находится только на месте второй группы выделенных колонок (смотри Рис. 1.), на других позициях выравнивания мотив не обнаруживается, то есть мотив достаточно специфичен.
Для Prosite паттерн выбранного мотива выглядит следующим образом [KS]-[RKMQ]-x-[GAH]-x-[GR]-x(2)-[NP]-W-G. В Prosite была найдена 31 последовательность, где есть участок, соотвествующий этому паттерну (выдачу можно посмотреть здесь).
Полученные последовательности вырoвняла с помощью встроенного алгоритма MAFFT в Jalview (смотри выравнивание здесь). С помощью паттерна проверила, насколько хорошо выровнялся мотив, и в 3 последоватедьностях он не выровнялся (при этом в выровненных последовательностях между аминокислотами этого мотива стоит очень много гэпов), поэтому решила удалить эти 3 невыровненные последовательности, и снова провела выравнивание, в результате мотив идеально выровнялся.
P. S. Прежде, чем удалять эти последовательности, посмотрела в UniProt, что это за белки, и ни один из них никак не относился к гиалуронанобразующим или мРНК-связывающим белкам. Но стоит отметить, что среди оставшихся после удаления 28 белках помимо гиалуронанобразующих или мРНК-связывающих белков был ещё 3 никак к ним не относящиеся, тогда можно сказать, что я выбрала не достаточно специфичный мотив.
По выраниваниям seed было построено дерево с помощью NJ в Jalview.
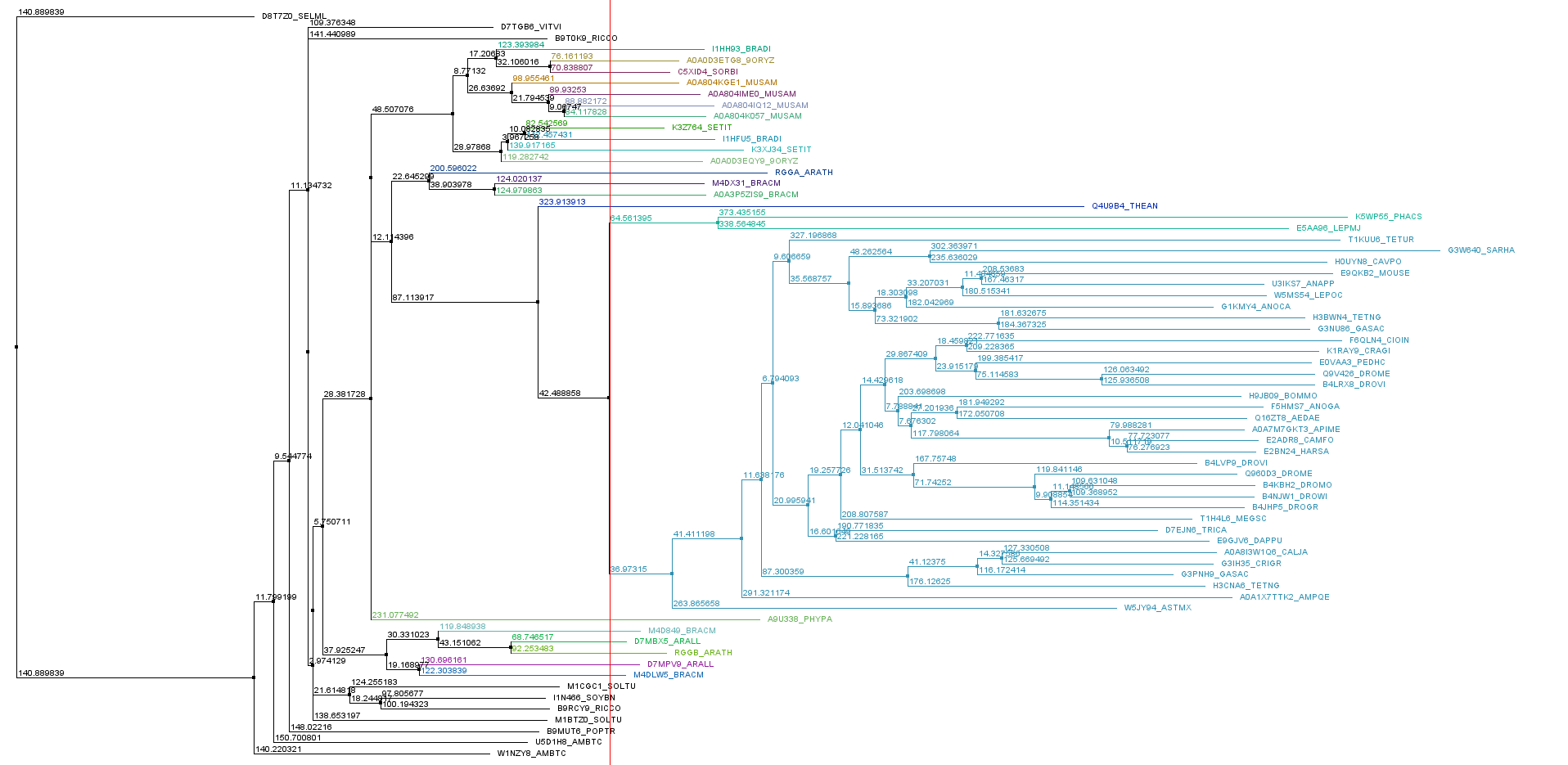
Решила проанализировать кладу, выделенную голубым цветом на дереве (смотри на Рис. 2. голубую кладу после красной вертикальной линии).
Опять применила окраску Clustal и выставила параметр Above identity threshold, равный 100%, у меня выделилось сразу 2 колонки, но этого мало для составления паттерна какого-то мотива, поэтому постепенно снижала значение параметра, и у меня выделился мотив, паттерн которого следующий [TS][LY]D[EV][WY][KR]. Провела по этому паттерну поиск мотива в других кладах, мотив нашёл только в выбранной кладе, при этом нашёлся во всех последовательностях данной клады, так что можно сделать вывод, что мотив специфичен конкретно для выбранной клады.
Был выбран белок P0AD49 - ингибитор A, ассоциированный с рибосомой. Это ингибитор из Escherichia coli. Во время стационарной фазы предотвращает образование рибосомы 70S, возможно, для того, чтобы регулировать эффективность трансляции во время перехода между экспоненциальной и стационарной фазами. Предполагается, что он ингибирует элонгацию трансляции, блокируя А-сайт.
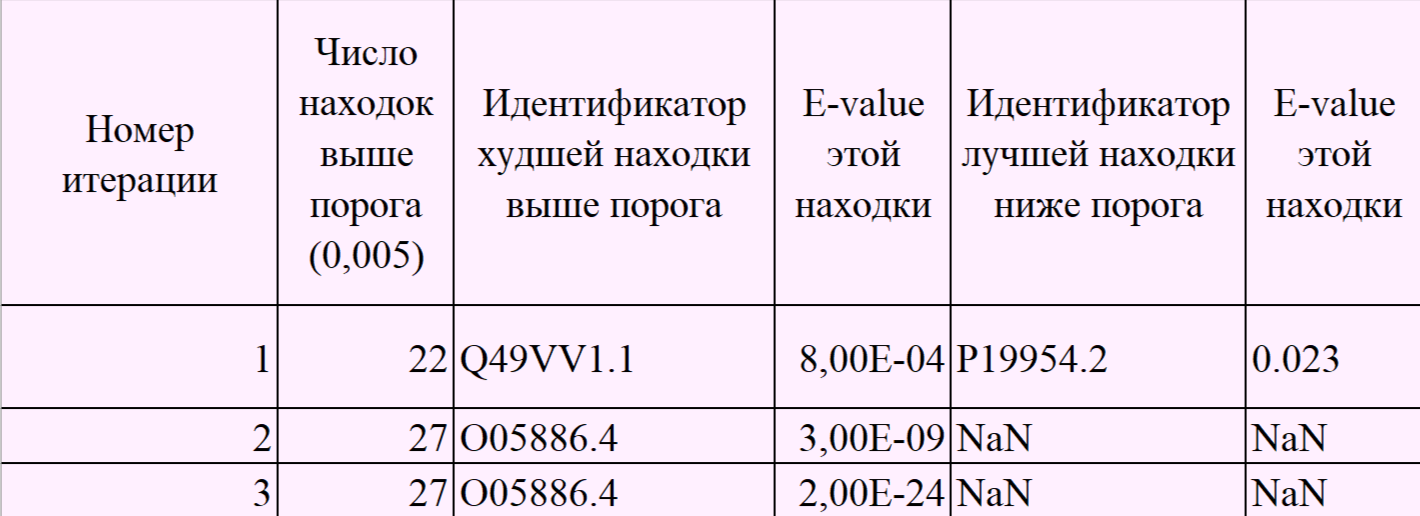
Из таблицы видно, что число находок, удовлетворяющих проговому E-value (0.005), перестало меняться на 3 итерации. Все полученные белки относятся к семейству ribosome hibernation-promoting factor, HPF/YfiA family. Наверное, можно сказать, что семейство "хорошее", так как максимальное число находок был найдено уже на 2 итерации.
Для этого задания я взяла домен PF00003. Взяла все 114 аннотированных последовательностей и запустила для них следующую команду:
meme pr4104.fasta -o meme_results -minw 4 -maxw 10 -nmotifs 4
То есть ищем 4 мотива, минимальная длина которых может быть 4 аминокислотных остатка, а максимальная - 10 остатков.
Выдачу можно посмотреть здесь.
В итоге, было найдено 4 мотива, каждый из которых имеет очень низкий E-value.

Теперь проверим, сколько раз встречается мотив c самым низким E-value (PENFNEAKFL) во взятых последовательностях:
fimo -motif PENFNEAKFI -thresh 0.001 ./meme_results/meme.txt pr4104.fasta
Выдачу можно посмотреть здесь.
Мотив встречается 154 раза в 114 последовательностях... Мотив немного перепредставлен. Мотив довольно длинный, поэтому можно предположить, что в одной архитектуре могут встречаться два одинаковых домена.
Оценка представленности сайта GATC проведена для генома бактерии Pseudomonas syringae. Для этого был необходим геном рассматриваемой бактерии и набор 24 сайтов.
cbcalc -s sites.txt -M -o res.tsv GCF_018394375.1_ASM1839437v1_genomic.fna
В результате получила файл, по которому построила гистограмму.
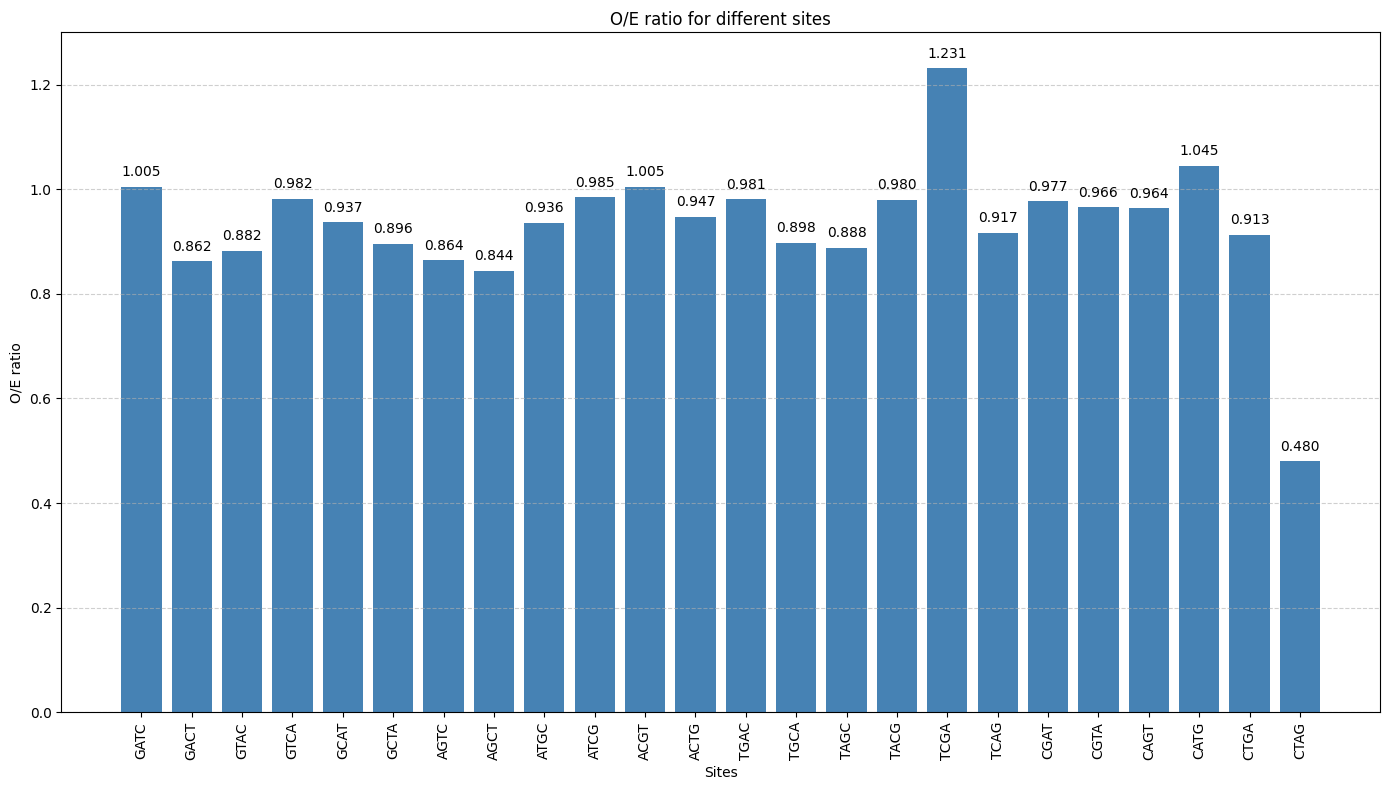
Cайт метилирования GATC имеет представленность чуть больше 1 так же, как и сайт ACGT. Видимо, оба этих сайта важены для метилирования, но существуют и два других сайты с большей представленностью (ТСGA и CATG). Наверное, они играют большую роль в метилировании (особненно сайт ТСGA) для Pseudomonas syringae.