Для данного практикума выбрала семейство 4Fe-4S binding domain (AC в базе данных Pfam PF00037) и по доменной архитектуре его подсемейство Domain of unknown function (DUF3470) (AC в базе данных Pfam PF11953).
Cемейство включает белки, содержащие домены, которые связываются с железо-серными кластерами. К ним относятся бактериальные ферредоксины, различные дегидрогеназы и различные редуктазы. Структура домена представляет собой альфа-антипараллельный бета-сэндвич.
Подсемейство было сформировано на домене, который пока не охарактеризован, но встречается у бактерий. Сам домен состоит из 50 аминокислот и содержит один полностью консервативный остаток N, который может иметь функциональное значение.
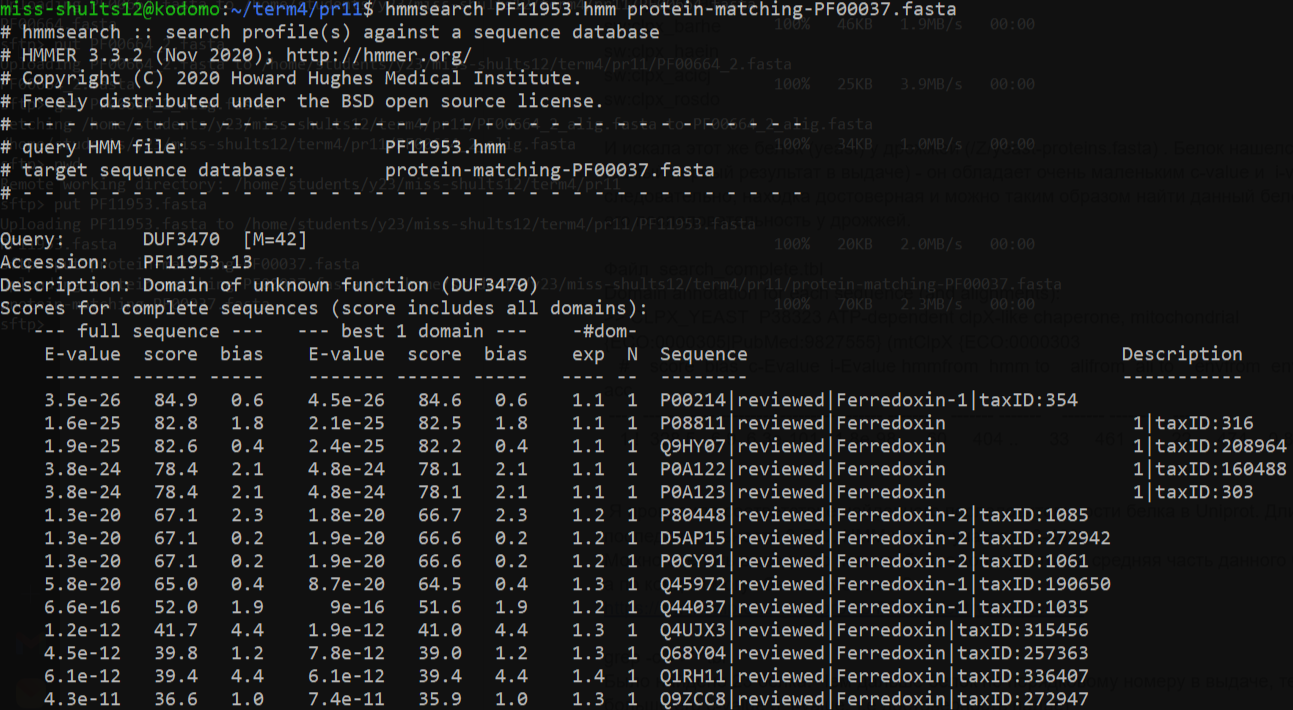
Для построения профиль HMM по последовательностям доменов (то есть не полных белков) выбранного подсемейства скачала выравнивание seed (содержит 145 последовательностей, смотри файл PF11953.fasta) и по этому выравниванию построила профиль (смотри файл PF11953.hmm). По построенному профилю с помощью hmmsearch искала белки, принадлежащие данному подсемейству, уже среди белков семейства, при этом проводила поиск только среди аннотированных последовательностей (это 249 полных последовательностей, смотри файл protein-matching-PF00037.fasta; всех последовательностей, в том числе и не аннотированных, 107 тысяч, но боюсь, что наш сервир просто не выдержит такого количества последовательностей, поэтому взяла только аннотированные). Все необходимые файлы лежат в папке ~/term4/pr11.

Думаю, что оптимальный порог на вес находки, который лучше всего выделяет подсемейство на фоне семейства, это score, равный 65.
При этом случайных (то есть не содержащий домен подсемейства) находок не оказалось, все находки принадлежат подсемейству DUF3470.
