Для выполнения практикума были выбраны протеомы 7 бактерий отдела Pseudomonadota:
Протеомы выбранных бактерий были объединены в один файл с помощью следующей команды:
cat AROAE.fasta BORPE.fasta BURMA.fasta HAEIN.fasta NEIMA.fasta PASMU.fasta ROSDO.fasta > ~/term4/practice4/db.fasta
Файлы с протеомами были взяты из директории /P/y22/term4/Proteomes.
Затем была создана локальная база данных для дальнейшего запуска BLASTP:
makeblastdb -in db.fasta -dbtype prot
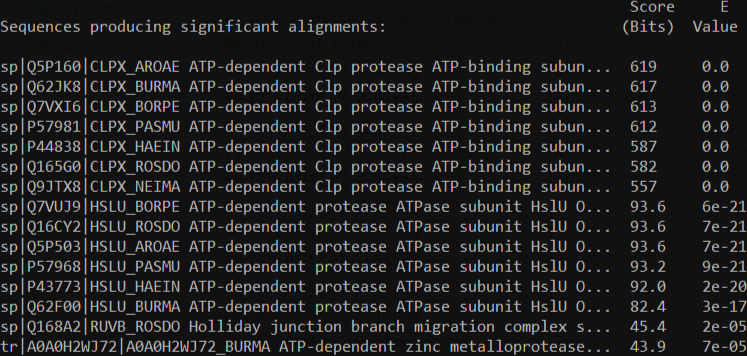
Далее на основе созданной базы данных был произведён поиск гомологов белка CLPX_ECOLI с порогом Е-value, равным 0,0001:
blastp -task blastp -query CLPX_ECOLI.fasta -db db.fasta -out blast_result.out -evalue 0.0001

Дерево было реконструировано с помощью программы FastME по модели p-distance.
дерево найденных гомологов в формате Newick
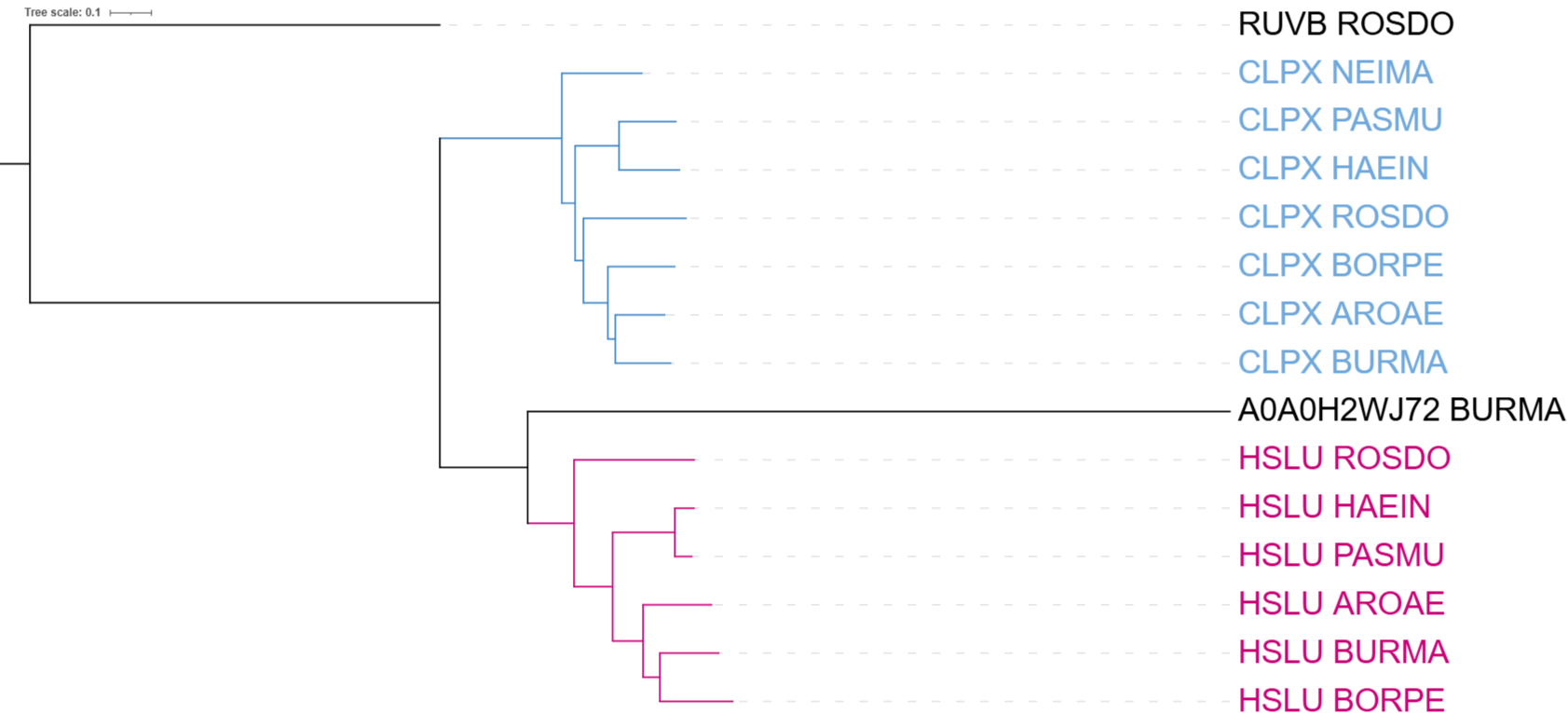
Ортологи: CLPX_PASMU и CLPX_HAEIN; CLPX_BORPE и CLPX_BURMA; HSLU_PASMU и HSLU_HAEIN; HSLU_AROAE и HSLU_BORPE.
Паралоги: CLPX_HAEIN и HSLU_HAEIN; CLPX_ROSDO и HSLU_ROSDO; CLPX_BURMA и HSLU_BURMA.
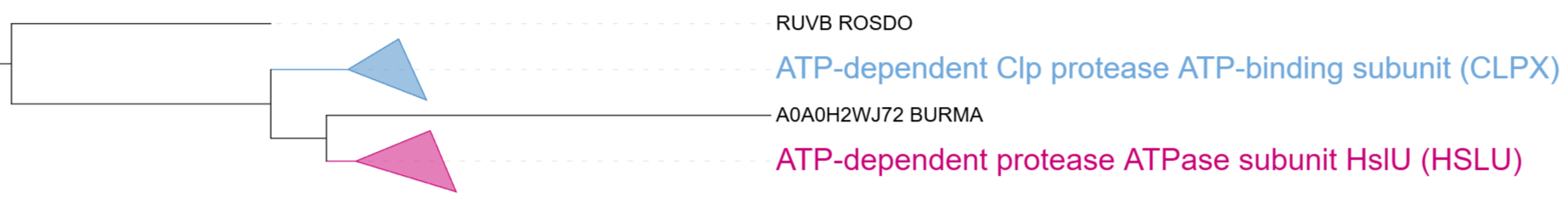
В ортологической группе (CLPX), выделенной голубым цветом, присутствует последовательность каждой из 7 выбранных бактерий, а в ортологической группе (HSLU), отмеченной розовым, присутствует только 6 последовательностей (нет последовательности NEIMA).
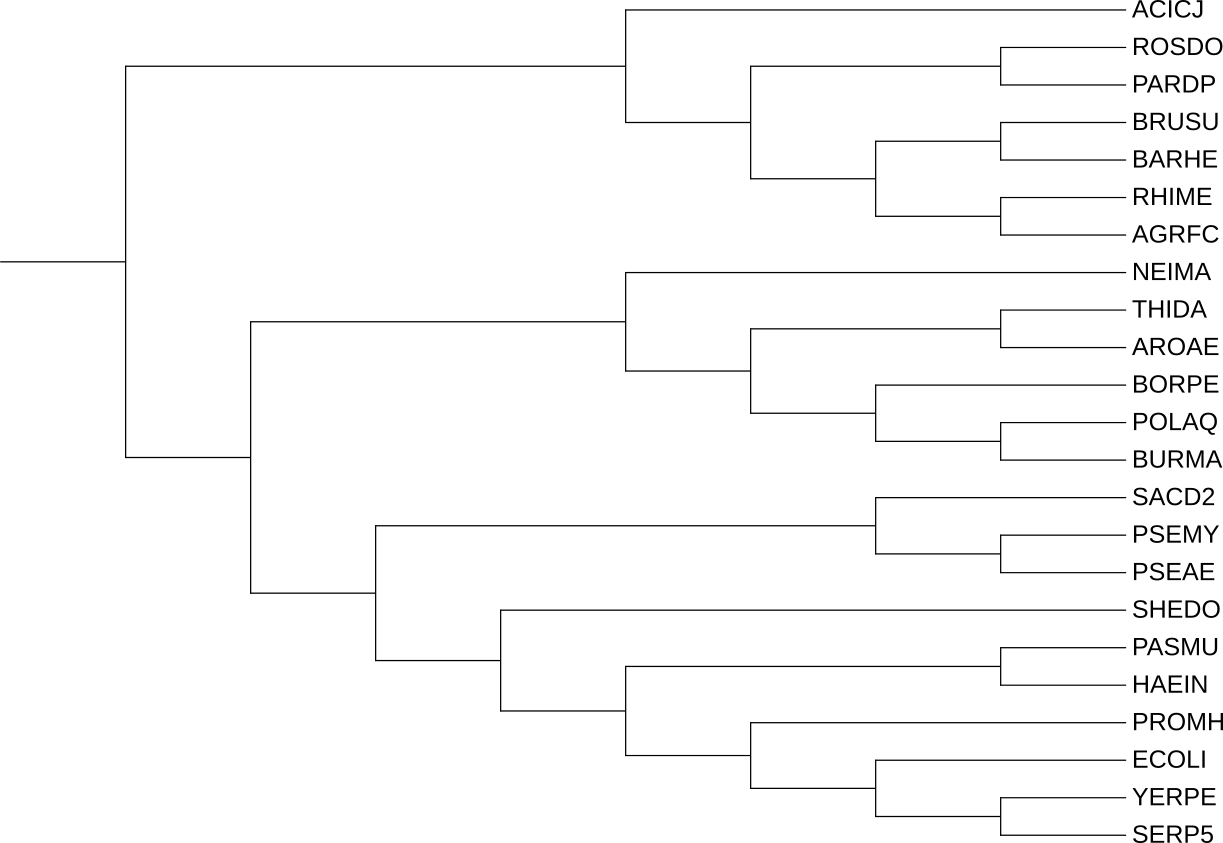
Если сравнивать реконструкции ортологических групп с филогенией бактерий (смотри Рис. 3. ниже), то клада ортологов HSLU соотвествует филогении бактерий, а в кладе CLPX последовательность CLPX_ROSDO, а не CLPX_NEIMA, должна являться внешней группой по отношению ко всей рассматриваемой кладе, то есть в данной кладе ортологов CLPX_NEIMA и CLPX_ROSDO надо поменять местами. Ещё, судя по филогении бактерий, клада (CLPX_BORPE, (CLPX_AROAE, CLPX_BURMA)) дожна выглядеть следующим образом (CLPX_AROAE, (CLPX_BORPE, CLPX_BURMA)).