
Отобранные бактерии
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Clostridium botulinum | CLOB1 |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Finegoldia magna | FINM22 |
| Geobacillus kaustophilus | GEOKA |
| Listeria monocytogenes | LISMO |
((FINM,(CLOTE,CLOB1)),(ENTFA,(((BACAN, BACSU),GEOKA),LISMO))); - скобочная формула.

Нетривиальные ветви:
Эти бактерии делятся на два больших таксона (две внешние ветви): Bacilli (ENTFA, BACAN, BACSU, GEOKA, LISMO) и
Clostridia (FINM2, CLOTE, CLOB1 - все принадлежат порядку Clostridiales, но
FINM2 выделяется в подпорядок Clostridiales incertae sedis ветвью, которая отделяет его от остальных Clostridiales). ENTFA, в отличие от остальных
Bacilli, относится к порядку Lactobacilliales, а не Bacilliales, и выделяется отдельной ветвью. LISMO, в отличие от
остальных Bacilliales, относится к таксону Listeriaceae, а не Bacilliaceae (так же отдельная ветвь). Bacillus anthracis
и subtillis относятся к одному роду, но разным группам: cereus и subtillis (каждая выделяется своей ветвью)
соответственно.
Выравнивание последовательностей было построено программой mafft.
Fprotpars выдала одно дерево.Оно совсем не такое, как ожидалось. Отсутствуют ветви, выделяющие от остальных:
Зато появились новые ветви, выделяющие:

Скобочная формула:((((RL2_FINM2,(RL2_CLOTE,RL2_CLOB1)),RL2_GEOKA),((RL2_ENTFA,RL2_LISMO), RL2_BACSU)),RL2_BACAN)

Таблица расстояний fprotdist. В качестве примера отклонения от ультраметричности рассмотрим расстояния CLOB1-CLOTE, CLOB1-FINM2 и CLOTE-FINM2. CLOB1- и CLOTE-FINM2, действительно больше, чем CLOB1-CLOTE, но они не равны между собой, а различаются на 0,457034 - 0,429604= 0,02743.
Расстояния от ENTFA и LISMO до BACSU больше, чем ENTFA-LISMO, но они различаются на одну десятую.
Для исследования отклонений от аддитивности вычислим следующие суммы:
Последние две суммы больше первой, но они различаются на пять сотых.
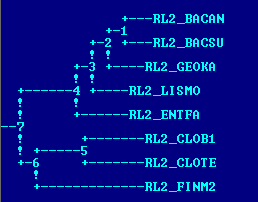
По алгоритму UPGMA получается правильное дерево. Скобочная формула:
(((((RL2_BACAN:0.06833,RL2_BACSU:0.06833):0.00526,RL2_GEOKA:0.07360):0.00803,
RL2_LISMO:0.08163):0.03655,RL2_ENTFA:0.11818):0.10887,((RL2_CLOB1:0.12434,
RL2_CLOTE:0.12434):0.09732,RL2_FINM2:0.22166):0.00539)

Если в дереве fprotpars GEOKA выделялась вместе с CLOB1, CLOTE и FINM2, то в дереве Neighbor Joining она выделяется с LISMO и ENTFA, а с (LISMO, BACSU,BACAN, ENTFA), как в правильном дереве GEOKA объединить нельзя.
BACSU объединяется с BACAN, как и в верном дереве. Отсутствует ветвь BACSU vs (ENTFA, LISMO), которая есть в дереве fprotpars.
В Neighbor Joining и fprotpars, в отличие от правильного, объединяются LISMO и ENTFA.
Скобочная формула Neighbour Joining:
(RL2_BACSU:0.06446,(RL2_GEOKA:0.07542,((RL2_LISMO:0.04903,
RL2_ENTFA:0.12587):0.02725,((RL2_CLOB1:0.11798,RL2_CLOTE:0.13070):0.09146,
RL2_FINM2:0.22752):0.13131):0.02236):0.00344,RL2_BACAN:0.07221)