| Бактерия | EMBL-запись | Координаты |
| GEOKA | EMBL; BA000043; BAD75499.1; -; Genomic_DNA | 10421..11973 |
| BACAN | EMBL:AE016879; AAP24167.1; -; Genomic_DNA. EMBL; AE017334; AAT29193.1; -; Genomic_DNA. EMBL; AE017225; AAT52450.1; -; Genomic_DNA. | 29129..30635 |
| BACSU | EMBL; U43929; AAC45959.1; -; Genomic_DNA. EMBL; D50302; BAA08834.1; -; Genomic_DNA. EMBL; AL009126; CAB11895.2; -; Genomic_DNA. | 30279..31832 |
| ENTFA | EMBL; AE016830; AAO80078.1; -; Genomic_DNA. | 248466..249987 |
| CLOB1 | EMBL; CP000726; ABS35648.1; -; Genomic_DNA | 9282..10783 |
| CLOTE | EMBL; AE015927; AAO37054.1; ALT_INIT; Genomic_DNA | complement(8715..10223) |
| FINM2 | EMBL; AP008971; BAG07576.1; -; Genomic_DNA. | 197837..199361 |
| LISMO | EMBL; AL591980; CAD00707.1; -; Genomic_DNA. | 96266..97811 |
В правильном дереве 5 нетривиальных ветвей. При различных реконструкциях часть из них может быть потеряна.
| Дерево | Сколько ветвей совпадают с правильным? | Какие отсутствуют |
| fprotpars | 2 ветви | 1. (BACAN, BACSU) 2. (GEOKA, BACAN, BACSU, LISMO) 3. (GEOKA, BACAN, BACSU) |
| fneighbor | 4 | 1. {FINM2,CLOTE, CLOB1, ENTFA} vs {LISMO, GEOKA, BACAN,BACSU} |
| upgma | правильное дерево | |
| retree | 3 | 1. {FINM2,CLOTE, CLOB1, ENTFA} vs {LISMO, GEOKA, BACAN,BACSU} 2. {FINM2,CLOTE, CLOB1, ENTFA,LISMO} vs {GEOKA, BACAN,BACSU} |
| выделение внешней группы | 4 | 1. {FINM2,CLOTE, CLOB1, ENTFA} vs {LISMO, GEOKA, BACAN,BACSU} |
| fdnapars | 1 | 1. Выделявшая LISMO, BACAN, BACSU и GEOKA. 2. Выделявшая BACAN, BACSU и GEOKA. 3. Выделявшая CLOTE, CLOB1, FINM |
Чтобы найти эти ссылки на записи EMBL, потребовалось получить соответствующие записи Swissprot командой entret sw:xxx, где ххх - Swissprot ID какого-нибудь белка этих бактерий.
Команда для получения выравнивания: muscle -in 16s.fasta -out 16s_aligned.fasta, где bacteria.fasta - файл с последовательностями, а 16s_aligned.fasta - файл с выравниванием.

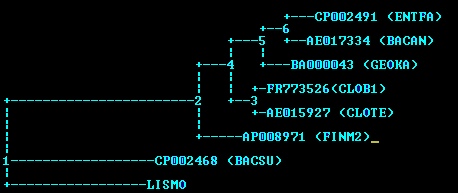
Скобочная формула: (((((CP002491:0.06056,AE017334:0.05135):0.03058,BA000043:0.05433):0.07739, (FR773526:0.03602,AE015927:0.04161):0.05432):0.05470,AP008971:0.09895):0.40577, CP002468:0.31591,LISMO:0.29765);
Это дерево построено программой fdnapars и отличается от правильного тем, что BACAN и BACSU не выделяются как родственные организмы, наоборот они очень далеки друг от друга.
Появились ветви, выделяющие BACAN и ENTFA; и BACAN, ENTFA и GEOKA.
Зато больше нет ветви, выделявшей LISMO, BACAN, BACSU и GEOKA.
В целом, деревья, построенные по белкам, ближе к верному. Cреди ошибок,допущенных в этом дереве, есть типичные и нетипичны: больше нигде нельзя найти ветви, выделяющие BACAN и ENTFA; и BACAN, ENTFA и GEOKA. Пропущенная ветвь, выделявшая LISMO, BACAN, BACSU и GEOKA, отсутствует и в большинстве белковых деревьев (см. таблицу).
Если судить по количеству "пропавших" ветвей, этот метод хуже всех методов реконструкции по белкам, за исключением fprotpars.
По сравнению с деревьями fprotpars, joining, retree в новом увеличилось расстояние от LISMO и BACAN до их ближайших соседей.
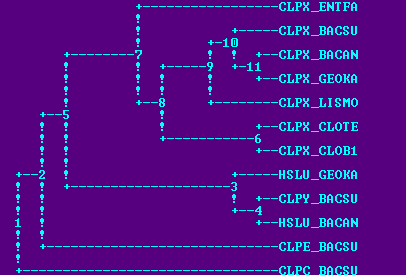
Паралоги CLPX_BACSU: CLPY_BACSU, CLPE_BACSU, CLPC_BACSU.
CLPX_BACAN и -GEOKA; CLPX_CLOTE и -CLOB1, CLPY_BACSU и HSLU_BACAN - ортологи.