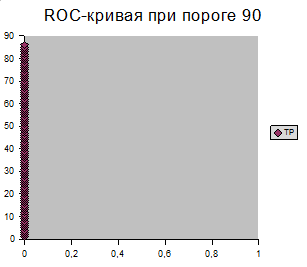
При значении порога 90 селективность наиболее близка к селективности паттерна, т.е. к 1.
При значении порога 90 селективность наиболее близка к селективности паттерна, т.е. к 1.
Для составления профиля был взят фрагмент выравнивания hit_all.msf, не соответствующий участку, по котрому был построен паттерн (см. файл). В выходном файле программы prophecy - матрица частот аминокислотных остатков. По столбцам - счет аминокислот,
по строкам - позиции в выравнивании. Указан размер профиля (length), максимальный счет и строка консенсуса.
Файл был получен следующим образом:
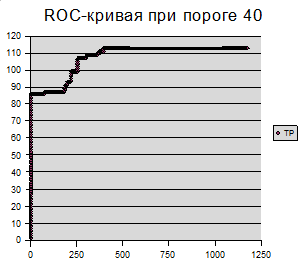
При пороге 30 находится 45754 находки.
Если повысить порог до 40, то всего 1292, >=50 - 630 находок, >=60 - 565.
Сравнивать списки будем с помощью функции ВПР. Будем искать наши находки в списке верных белков. Находки, которые совпадут - true positves, а остальные - false positives. Файлы с матрицей и списком находок лежат в директории: H:\Term4\Practice11.
 При значении порога 90 селективность наиболее близка к селективности паттерна, т.е. к 1.
При значении порога 90 селективность наиболее близка к селективности паттерна, т.е. к 1.