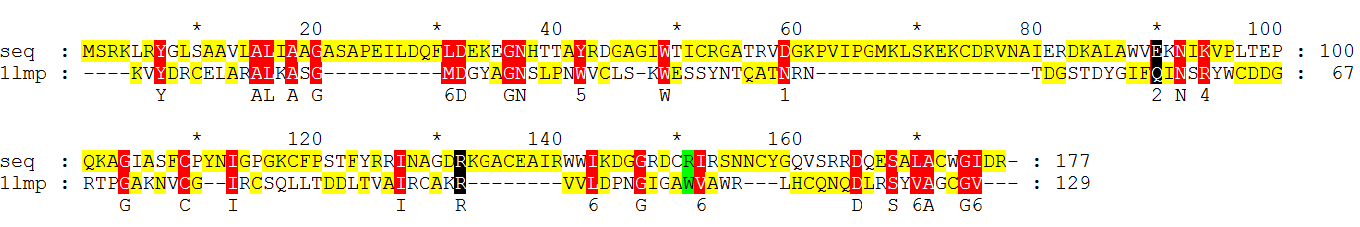
Для начала, с помощью программы Clustal постороили выравнивание лизоцима форели с лизоцимом фага энтеробактерии VT2-Sa. Нужно было сразу указать формат выдачи pir.
Затем требовалось найти, атомы каких остатков образуют водородные связи с лигандом в лизоциме форели - азоты и кислороды на пасстоянии до 3,5 А.
Теперь, с помощью выравнивания надо выяснить, какие остатки образуют эти связи в белке фага (отмечены зеленым на картинке)

Вносим необходимые изменения в файл 1lmp.pdb и получаем файл 1lmp_now.ent .
Можно запускать скрипт . Этот скрипт дает пять моделей исследуемого белка. Если сравнить в PyMol эти структуры с лизоцимом форели, видны некоторые недостатки выравнивания.
Например, длинная петля в лизоциме фага и несовпадение С и N концов, вообще лизоцим фага намного длиннее.

Совмещение 1lmp с 1ой моделью.

Совмещение 1lmp с 2ой моделью.
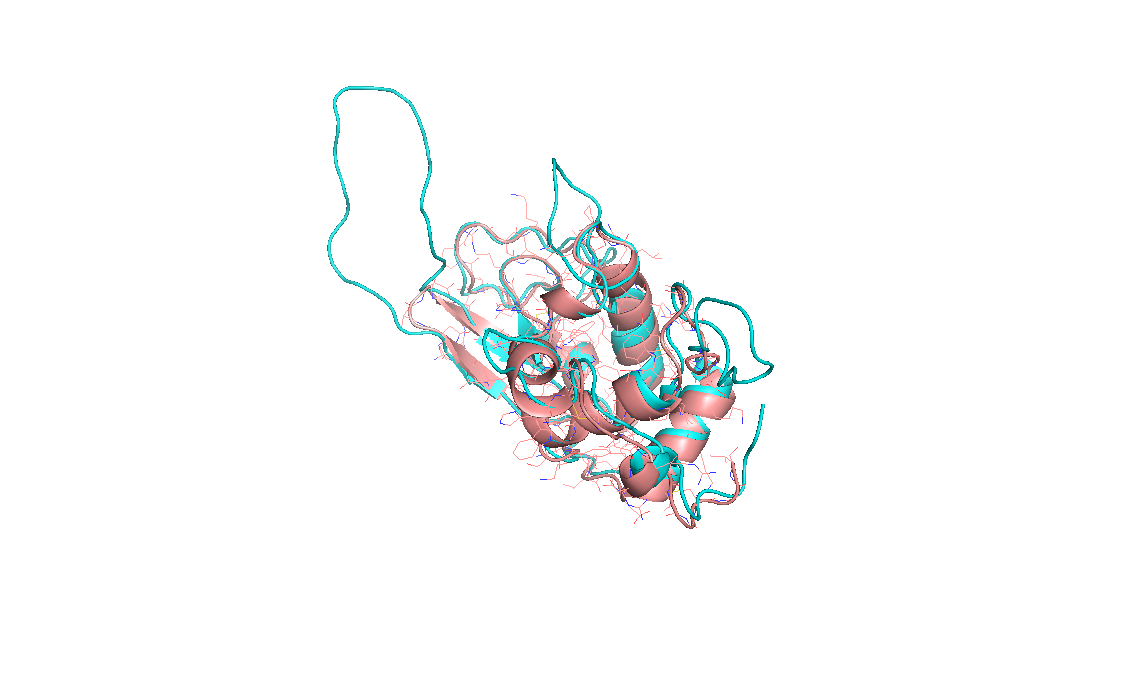
Совмещение 1lmp с 3й моделью.
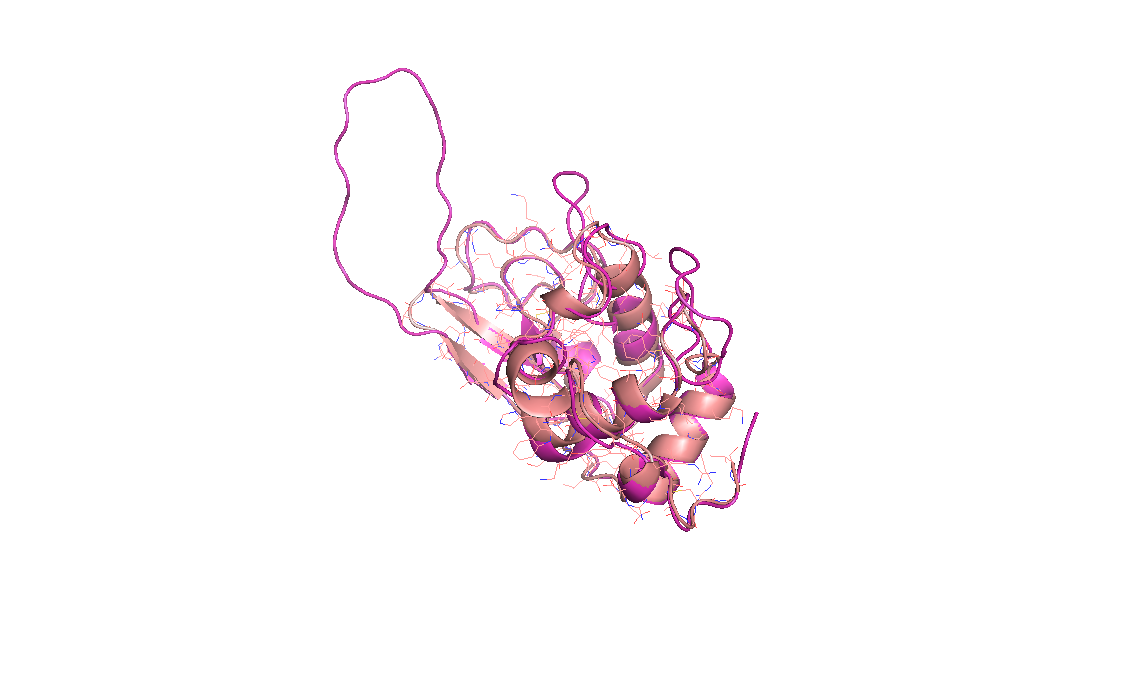
Совмещение 1lmp с 4ой моделью.

Совмещение 1lmp с 5ой моделью.
Сами модели визуально практически не отличаются, при наложении почти совпадают.