open3dalign.sh
import type=SDF file=compounds_best_conformer.sdf
align object_list=1
save file=aligned.sdf
Повезло! PyMol читает файл в данной кодировке. Получено изображение:

Для начала, для использования всех необходимых программ добавляем путь export PATH=$PATH:/home/preps/grishin/open3dtools/bin.
Затем строим выравнивание с лучшими конформациями.
open3dalign.sh
import type=SDF file=compounds_best_conformer.sdf
align object_list=1
save file=aligned.sdf
Повезло! PyMol читает файл в данной кодировке. Получено изображение:

Теперь можно приступать к построению модели. Команды:
open3dqsar.sh
import type=sdf file=aligned_ok.sdf
import type=dependent file=activity.txt
box
set object_list=60-85 attribute=TEST
set object_list=86-88 attribute=EXCLUDED
calc_field type=VDW force_field=MMFF94 probe_type=CR
cutoff type=max level=5.0 field_list=1
cutoff type=min level=-5.0 field_list=1
zero type=all level=0.05
sdcut level=0.1
nlevel
remove_x_vars type=nlevel
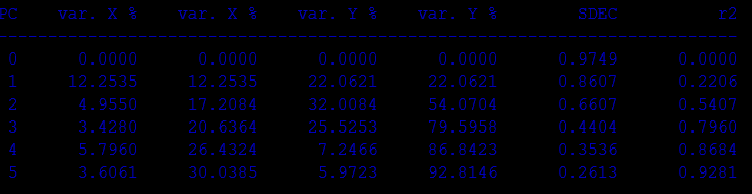
pls
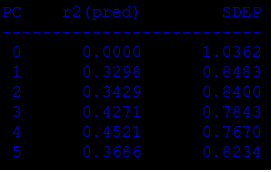
И получаем следующие коэффициенты:
cv type=loo runs=20
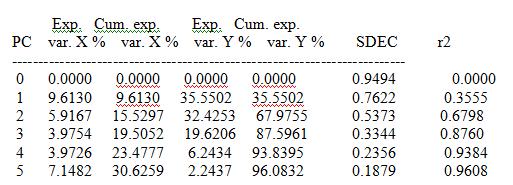
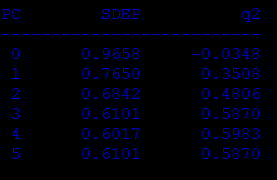
И получаем следующие коэффициенты:
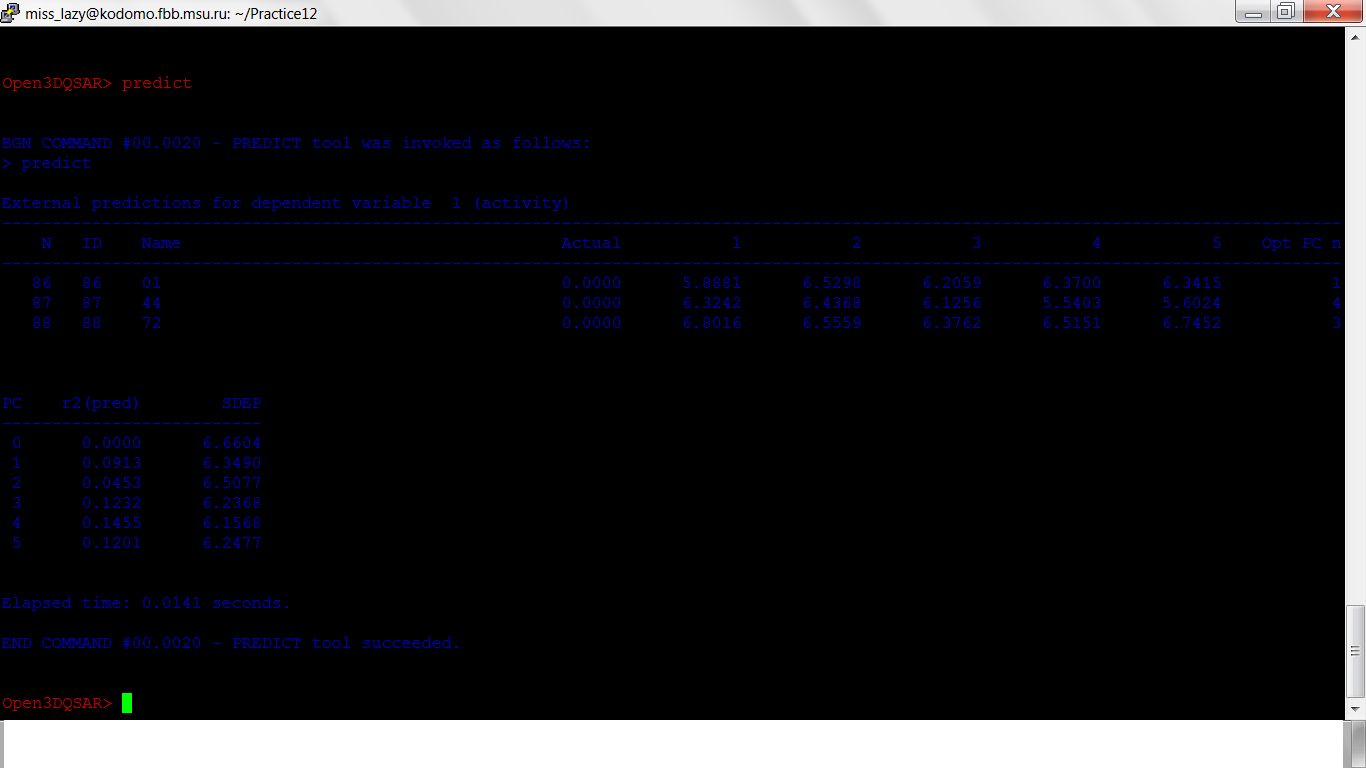
predict
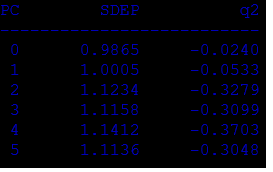
И получаем следующие коэффициенты:
На этот раз пришлось менять кодировку.
Изображение: 
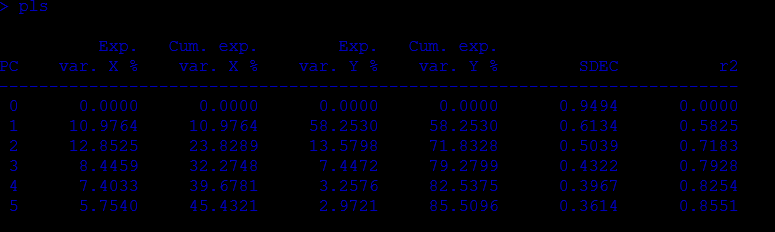
pls:
Кросс-валидация:
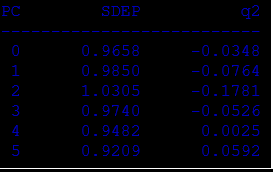
predict: