 --------------->
---------------> 
В отчете приведены результаты анализа качества структуры левансахаразы, расшифрованной методом РСА в 2005 году К.Мартинесом-Флейтесом и др., и содержащимся в PDB под кодом 1w18.
Выявлено, что гидролитическая машинерия левансахараз высоко консервативна, и что сайты связывания ионов кальция в белке Bacillis Subtilis и дисульфидный мостик Cys339-Cys395 занимают эквивалентные позиции в структуре, т.е., возможно, играют сходную роль в ее стабилизации.
 --------------->
---------------> 
Фермент относится к семейству 68 гликозидгидролаз.
Структура была определена с помощью рентгеноструктурного анализа на разрешении 2.5 A и усовершенствована методом молекулярного замещения с использованием гомолога из Bacillus subtilis (PDB-код: 1OYG).
R-факторы составили: 19.6%(R-cryst) и 24.9% (R-free). Для подсчета последнего использовано 5% рефлексов.
Асимметрическая единица содержит две копии цепи, а также в модель вошли 194 молекулы воды и четыре сульфат-иона.
Вот как выглядит эта структура:

C.Martinez-Fleites et al, 2005
На двух нижних изображениях показана структура каталитического центра.
Были обнаружены некоторые маргинальные остатки:
Для цепи А (Asn503):
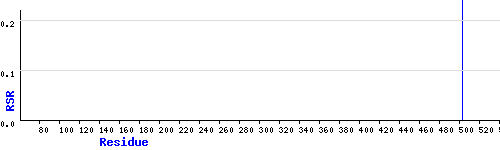
И В - Ala214, Gln232, Ser379, Asp479:
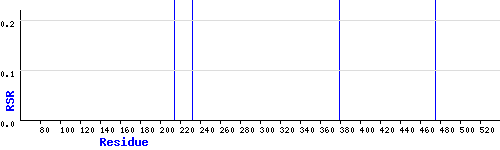
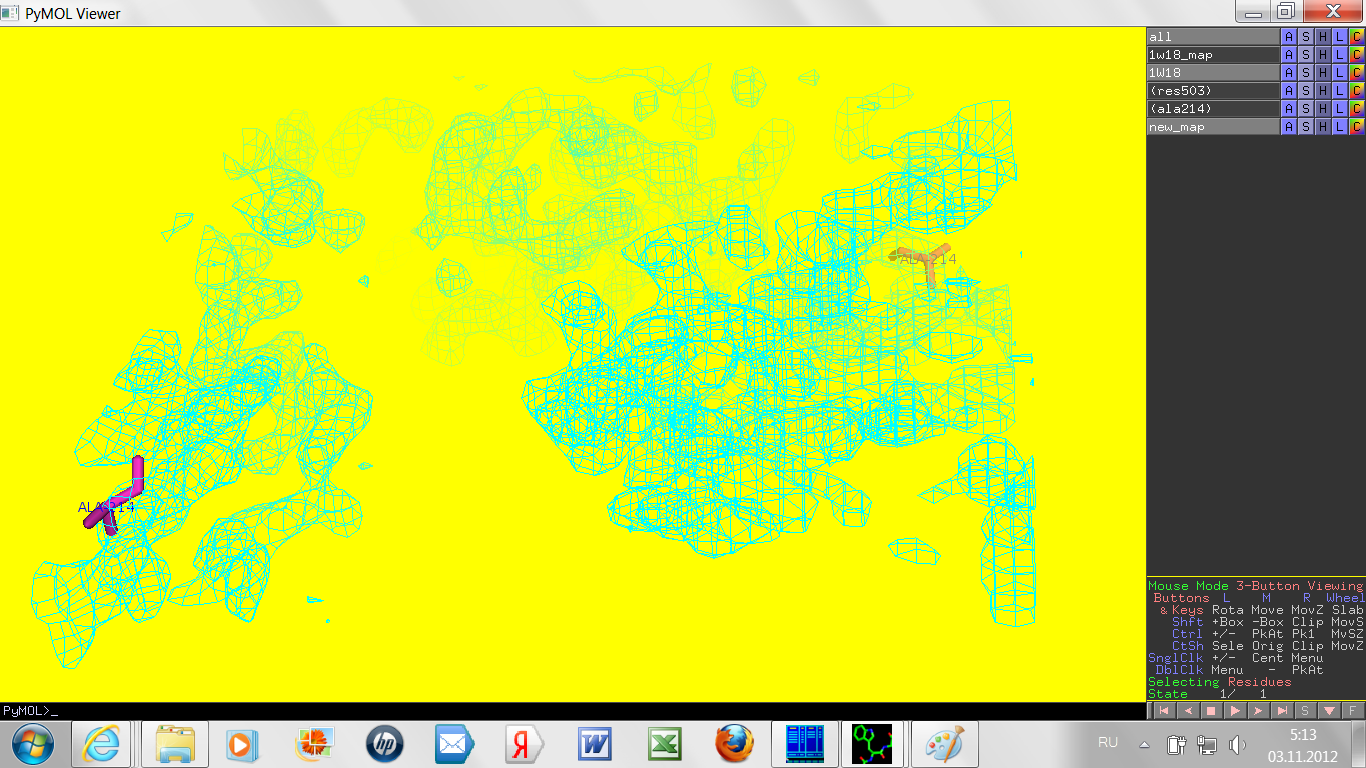
Например, остаток Ala214 имеет RSR 0,28. Вот его электронная плотность (голубым) в сопоставлении со всей молекулой (зеленым):
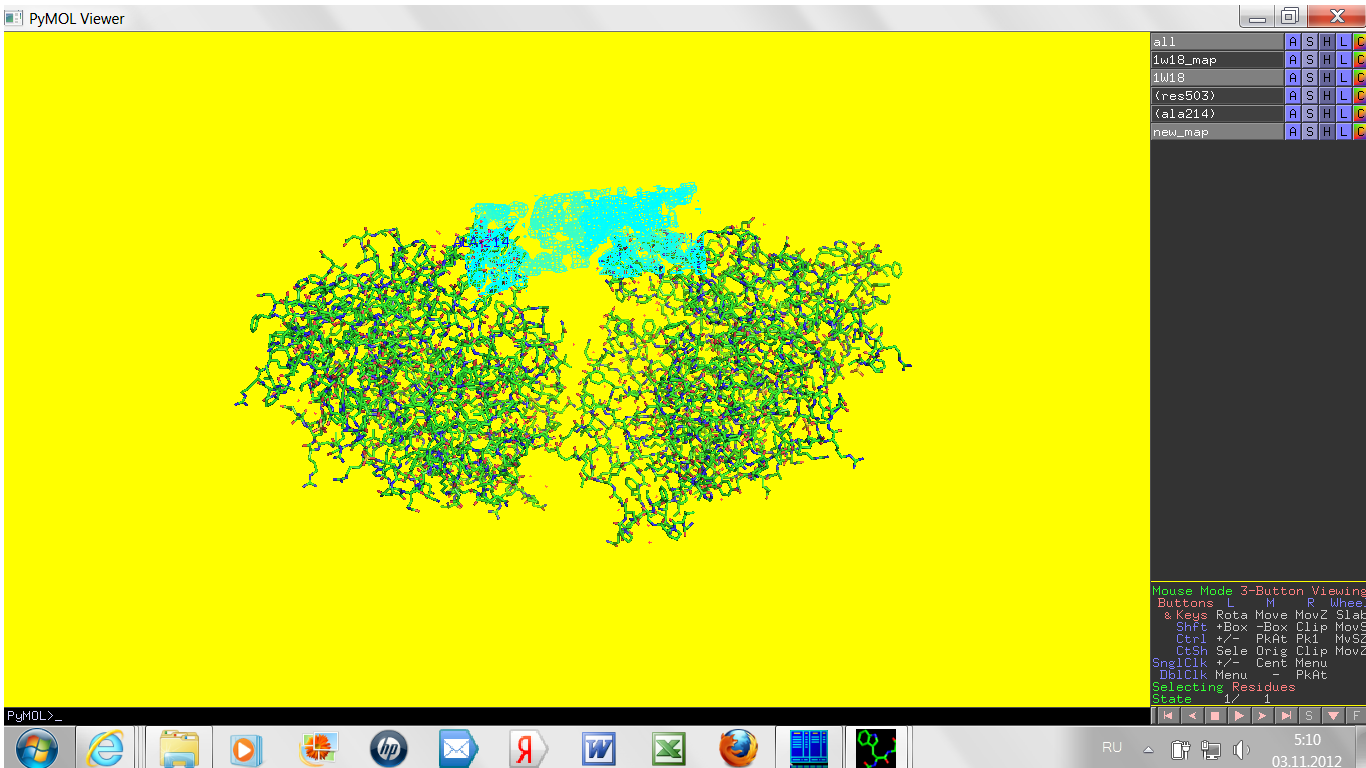
И отдельно:
На картах Рамачандрана: х - глицины, квадратами - пролины, + - остальные остатки.
Для цепи А: 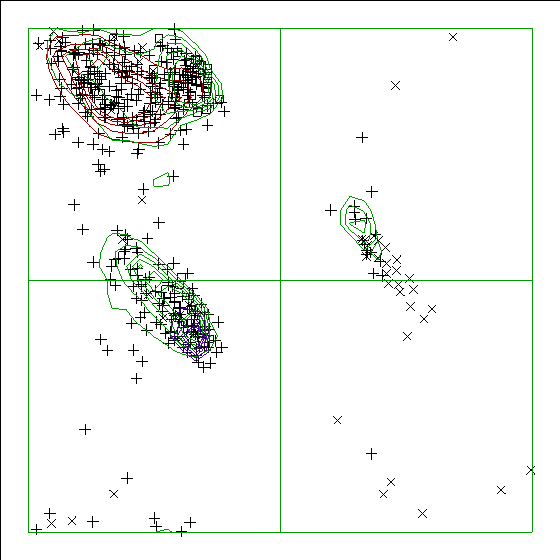
И В: 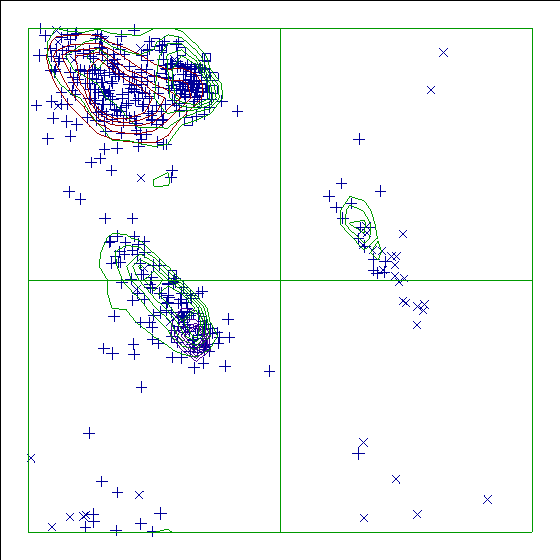
Обнаружены нарушения в конформациях некоторых остатков аргинина, тирозина, фенилаланина, аспарагиновой и глутаминовой кислот; ненормальные длины связей и значения углов; ненормальные конформации остова. Имеются остатки с неправильным окружением:
30 ASP ( 92-) A 61 ASP ( 123-) A 79 ASP ( 141-) A 109 ARG ( 171-) A 121 ARG ( 183-) A 128 ARG ( 190-) A 144 ASP ( 206-) A 182 ASP ( 244-) A 188 ASP ( 250-) A 211 ASP ( 273-) A 230 ASP ( 292-) A 241 GLU ( 303-) A 247 ASP ( 309-) A 265 GLU ( 327-) A 272 ARG ( 334-) A 292 GLU ( 354-) A 296 GLU ( 358-) A 336 ASP ( 398-) A 339 GLU ( 401-) A 358 ARG ( 420-) A 366 ASP ( 428-) A 380 ARG ( 442-) A 399 ASP ( 461-) A 465 ARG ( 527-) A 481 ARG ( 543-) A
HIS, ASN, GLN в неправильной ориентации:
47 ASN ( 109-) A 132 ASN ( 194-) A 158 ASN ( 220-) A 193 ASN ( 255-) A 203 GLN ( 265-) A 235 GLN ( 297-) A 239 GLN ( 301-) A 244 ASN ( 306-) A 260 ASN ( 322-) A 267 ASN ( 329-) A 287 ASN ( 349-) A 295 GLN ( 357-) A 305 GLN ( 367-) A 357 HIS ( 419-) A 387 ASN ( 449-) A 401 ASN ( 463-) A 420 GLN ( 482-) A 491 GLN ( 553-) A 506 GLN ( 75-) B 539 GLN ( 108-) B 540 ASN ( 109-) B 545 GLN ( 114-) B 625 ASN ( 194-) B 686 ASN ( 255-) B 696 GLN ( 265-) B 728 GLN ( 297-) B 732 GLN ( 301-) B 737 ASN ( 306-) B 753 ASN ( 322-) B 760 ASN ( 329-) B 880 ASN ( 449-) B 894 ASN ( 463-) B 913 GLN ( 482-) B 917 HIS ( 486-) B 978 ASN ( 547-) B
Итак, авторы получили более или менее точную модель левансахаразы, однако некоторых недостатков все же не удалось избежать; R-free все же немногим больше 20%, и в предпочтительной области на карте Рамачандрана находятся менее 90% остатков. Некоторые остатки плохо вписались в электронную плотность, не у всех комфортное окружение и правильная ориентация.
Настораживает и то, что с последовательностью гомолога из Bacillus subtilis всего 31% сходства.