Для выполнения задания выбрала домен SesB, который встречается в белках грибов, участвующих в клеточной гибели.
Accession: PF17046
Full: 344
Для данного домена известно 70 архитектур. Возьмем состоящую из PF14479 - PF17046, имеющей двудоменную организацию.
В нём 62 белка.
Я скачала все последовательности, принадлежащие этой доменной архитектуре:
kleshchenko_full_arch.fasta
Затем выровняла в JalView c помощью MUSCLE и обрезала так, чтобы остались только два домена. Затем
я с порогом 87% выделила тренировочную и тестовую выборки.
Затем с помощью команд я запустила на kodomo программы из пакета HMMER 2.3.2:
hmm2build hmmout train.fa
hmm2calibrate hmmout
hmm2search --cpu=1 hmmout kleshchenko_full_arch.fasta > hmm_results_arch.txt
Получила файл hmmout и hmm_results_arch.txt.
Используя таблицу Scores for complete sequences и скрипт
Карина Каримовой
я построила графики.
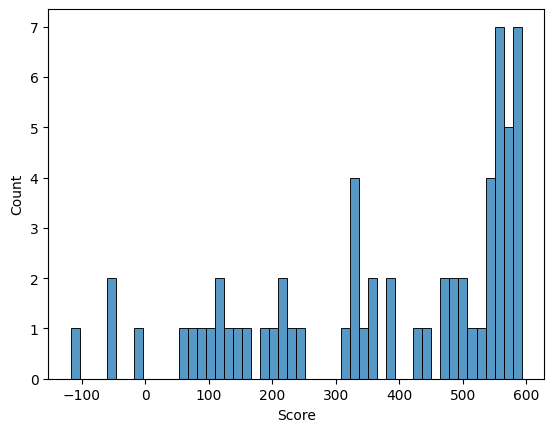
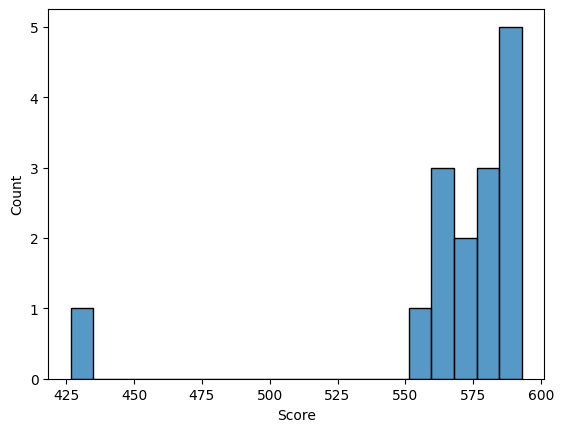
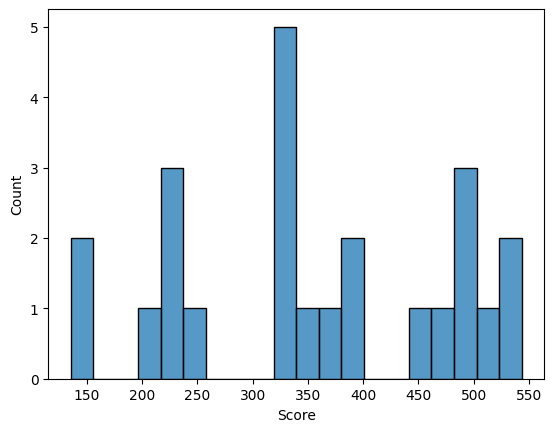
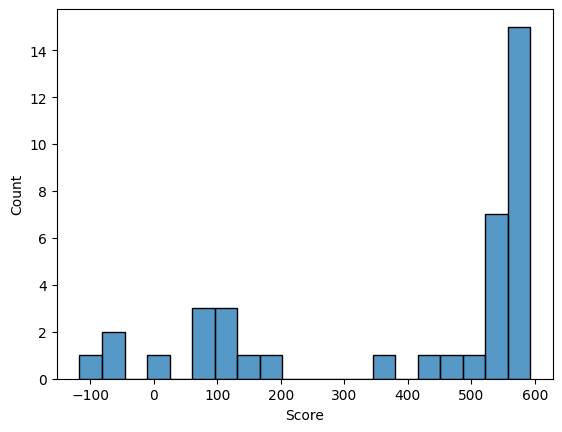
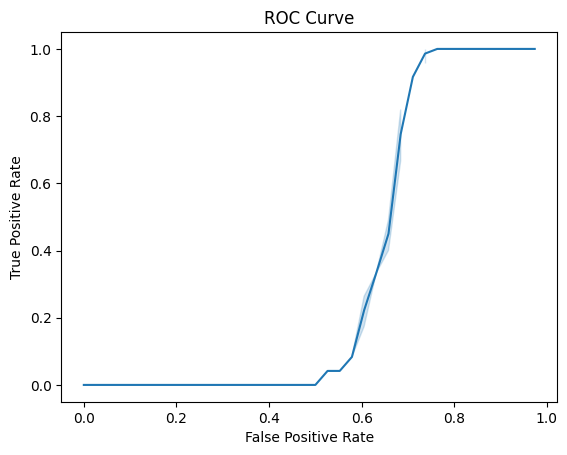
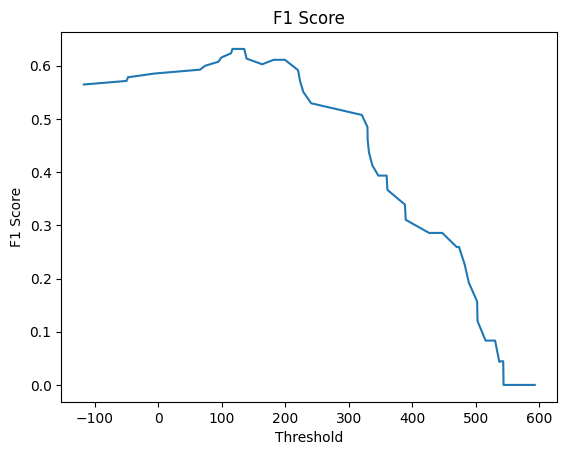
Честно говоря, результаты получились не очень. ROC-кривая вообще на себя не похожа. Так что я могу сказать,
что правильных предсказаний я не вижу.
Большое спасибо за прочтение моего практикума! Хорошего Вам настроения!