Составление списка гомологичных белков, включающих паралоги
Для поиска гомологичных с CLPX_ECOLI белков в бактериях, которые мною были использованы в предыдущих практикумах,
я использовала программу blastp. Для этого на kodomo в директории ~/term4/pr4/ были введены следующие команды:
cat PSEAE.fasta ROSDO.fasta THIDA.fasta YERPE.fasta BARHE.fasta SACD2.fasta PSEMY.fasta AROAE.fasta > ~/term4/pr4/proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out prot
blastp -query clpx_ecoli.fasta -num_threads 4 -db prot -evalue 0.001 -out blast_results.txt
В результате я получила файл, который затем немного отредактировала, дабы убрать лишнюю информацию.
Реконструкция и визуализация
В данный файл были помещены последовательности, которые мы взяли из выдачи BLAST.
Для рыравнивания последовательностей был использован алгоритм MAFFT, а затем для построения дерева - FastMe на интернет-ресурсе NGPhylogeny.fr.
Были выбраны следующие параметры для FastMe:
Gamma distributed rates across sites — NO
Starting tree — BIONJ
No refinement
100 bootstrap реплик
Остальные параметры - по умолчанию.
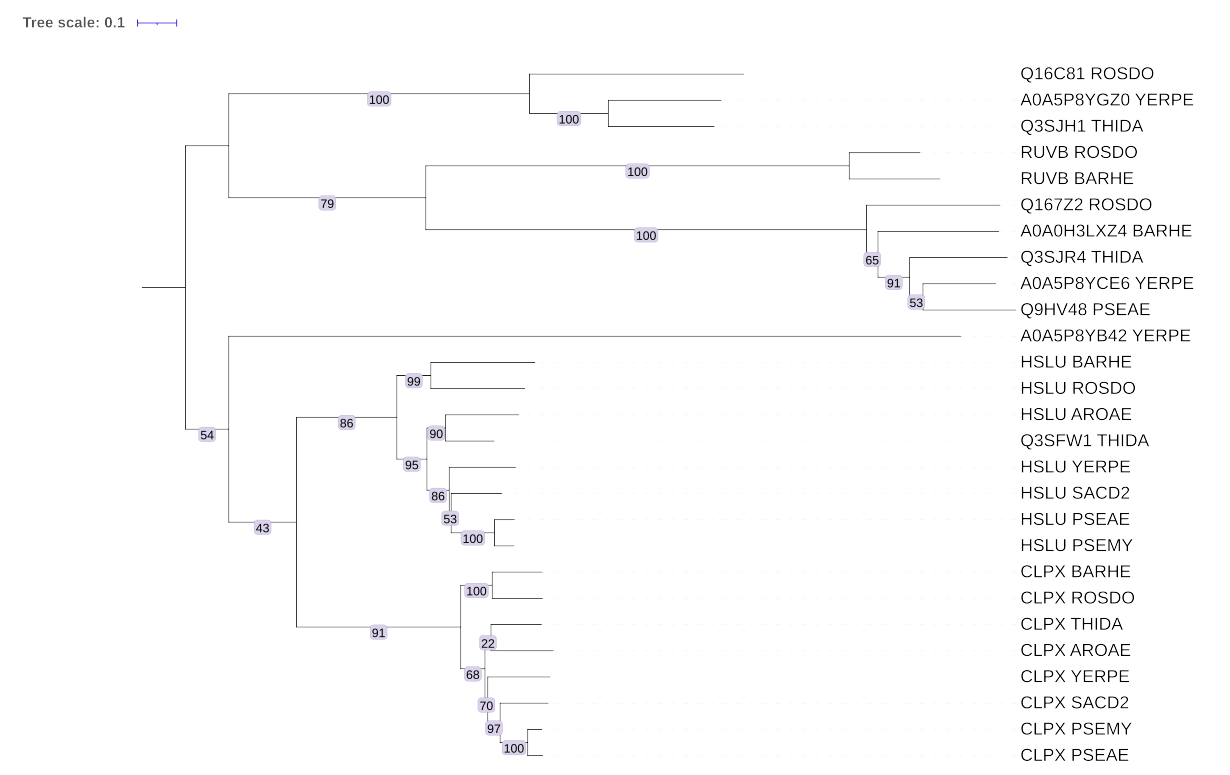
| № | Пара ортологов | |
|---|---|---|
| 1 | RUVB_ROSDO | RUVB_BARHE |
| 2 | HSLU_ROSDO | HSLU_BARHE |
| 3 | CLPX_PSEMY | CLPX_PSEAE |
Табл.1. Ортологи.
| № | Пара паралогов | |
|---|---|---|
| 1 | RUVB_ROSDO | Q167Z2_ROSDO |
| 2 | CLPX_YERPE | A0A5P8YB42_YERPE |
| 3 | Q3SJR4_THIDA | Q3SJH1_THIDA |
Табл.2. Паралоги.
Считая дерево реконструированным верно, я нашла три пары паралогов и три пары ортологов.
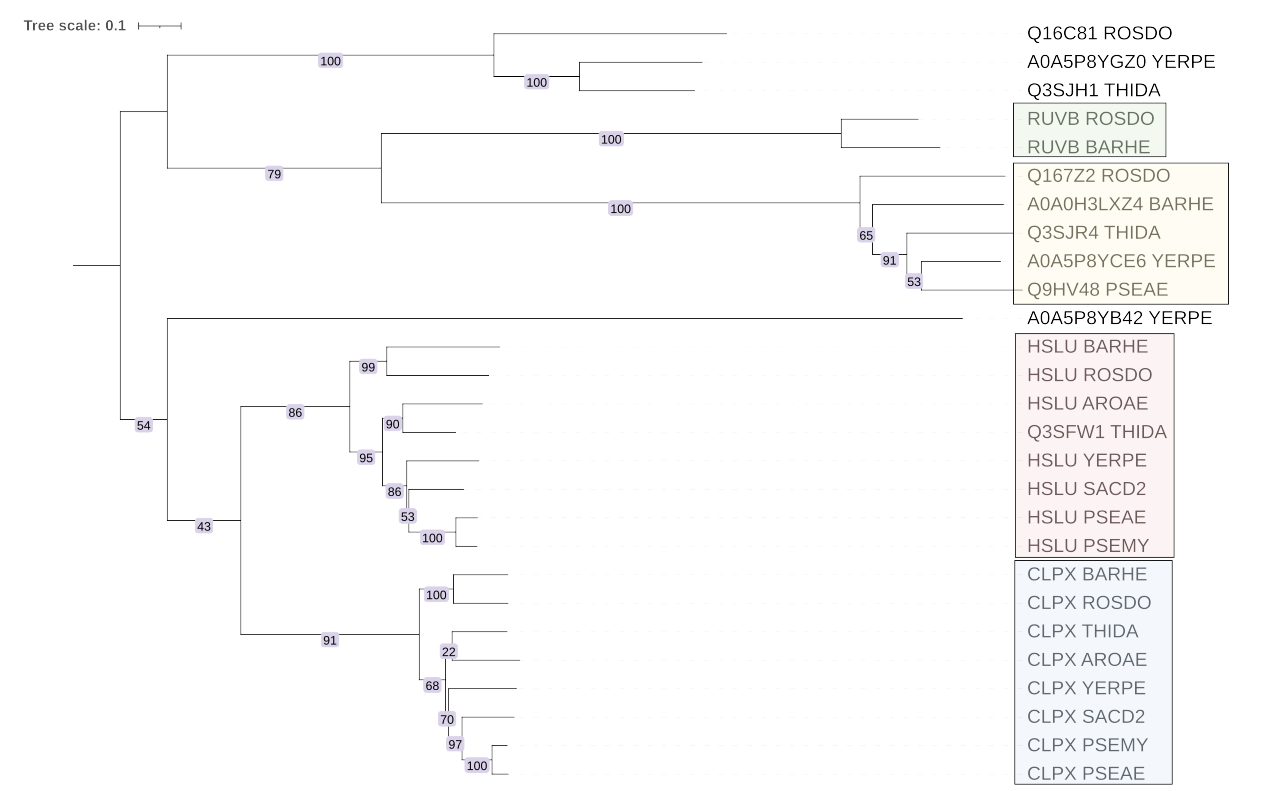
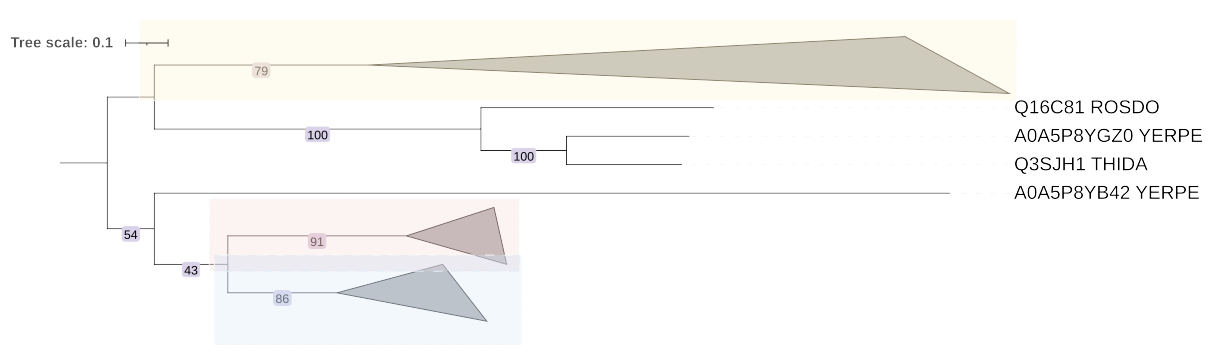
В зеленой группе мне захотелось объединить белки всего лишь двух бактерий, но с одинаковой мнемоникой RUVB.
В желтой группе находятся белки 5 бактерий, все имеют разню мнемонику, причем не очень-то читаемую.
В розовой все кроме одного принадлежат мнемонике HSLU, а выбивающийся - Q3SFW1.
В голубой все из CLPX. Группа, как и предыдущая, состоит из белков восьми бактерий (всех).
Причем филогения голубой группы в точности соответствует той, что мы получили в предыдущий практикумах.
В желтой группе находятся белки 5 бактерий, все имеют разню мнемонику, причем не очень-то читаемую.