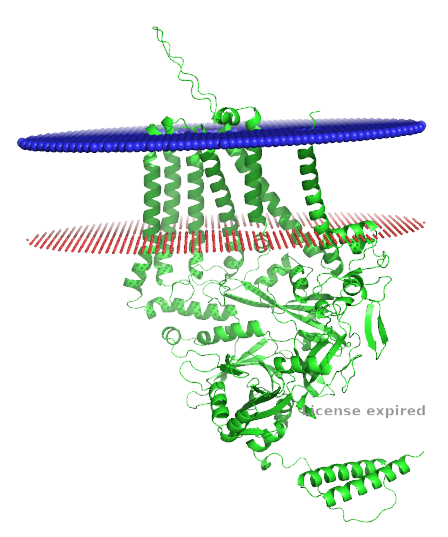
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
В базе данных OFM я выбрала белок Fatty Acid Transporter FadL с мнемоникой
FADL_ECOLI и идентификатором UniProt P10384.
Русское название
(Белок-переносчик длинноцепочечных жирных кислот) отражает функционал этого белка.
Его последовательность в fasta формате из PDB 3PGS.fasta, а идентификатор PDB 3PGS.
Он принадлежит всеми любимой Escherichia coli. На картинке можно посмотреть как он выглядит и как располагается в
билипидном слое: сверху (над красной мембраной) - межклеточное пространство, а под синим - периплазма.
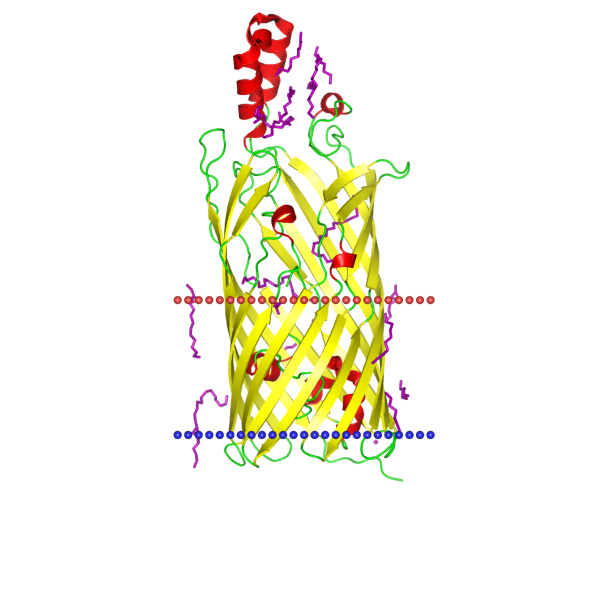
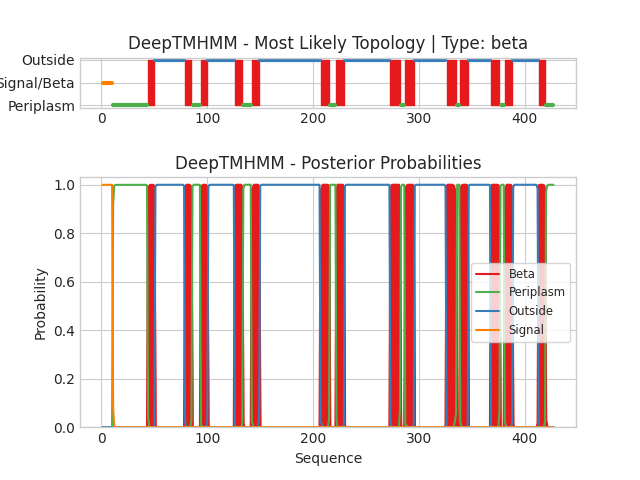
Координаты трансмембранных участков в OPM: 43-53, 78-87, 94-101, 126-135, 142-148, 208-216, 223-229, 274-281, 289-295, 325-334, 341-348, 367-376, 383-390, 411-421
По оси x обозначены номера последовательности. По оси y - вероятность нахождения остатка относительно мембраны там, где показано цветом соответсвенно легенде.
Координаты трансмембранных участков в DeepTMHMM: 44-50, 79-85, 94-100, 126-133, 142-149, 207-215, 223-229, 273-282, 287-295, 326-335, 339-346, 368-376, 381-388, 413-419
Обе программы предсказали по 14 участков β-тяжей, в среднем каждый состоит из 8 остатков. Но DeepTMHMM относителльно OPM уменьшала трансмембранные участки.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок с мнемоникой Y1161_SYNWW и
идентификатором SwissProt Q0AXT4 . Его название UPF0182 protein Swol_1161, честно говоря я не знаю как это
перевести... Выделен он из организма Syntrophomonas wolfei subsp. wolfei (strain DSM 2245B / Goettingen). Его
последовательность хранится в файле: Q0AXT4.fasta.txt.
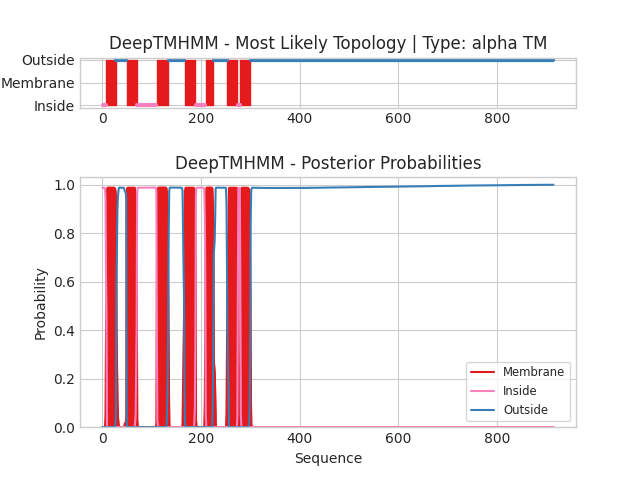
Координаты трансмембранных участков в DeepTMHMM: 8-28, 50-70, 111-134, 167-189, 210-225, 254-274, 280-300.
Предсказано 7 участков трансмембранных альфа-спиралей.
Параметры для запуска: Number of Membranes - 2, Type of membrane - Gram-positive bacteria
inner membrane и Gram-positive bacteria outer membrane (так как DeepTMHMM говорил и про внешнюю
и про внутреннюю чати клетки), Allow curvature - no, Topology (N-ter) - in (основываясь на предсказании DeepTMHMM).
Координаты трансмембранных участков в PPM 3.0: 6- 24, 49- 68, 113- 129, 167- 186, 207- 225, 257- 275, 278- 294.
Снова второй алгоритм стеснительный и предсказывает трансмембранные участки более маленького размера, нежели первый,
но зато их количество и примерное расположение совпадают.