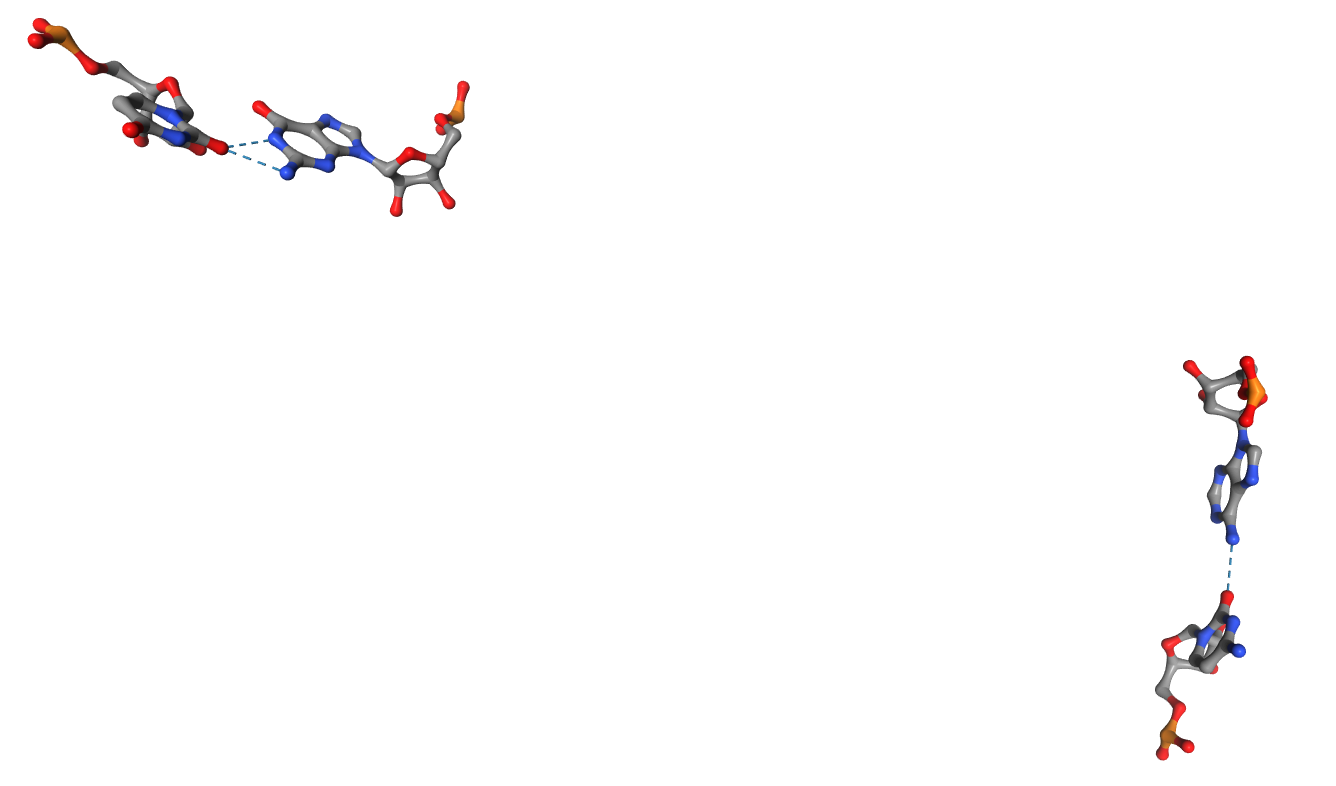
Пакет 3DNA
Пространственные структуры форм ДНК
Анализ структуры B-DNA
Для сравнения и дальнейшего анализа я выбрал B-форму ДНК, так как она показалась мне наиболее красивой. Опуская очевидное различие в длинах эксперементального и теоретически полученного фрагментов спирали, можно скзаать, что теоретическая модель, построенная в 3DNA, выглядит более гладкой и симметричной, правильной. Экспериментальная выглядит, как списанная у отличника работа
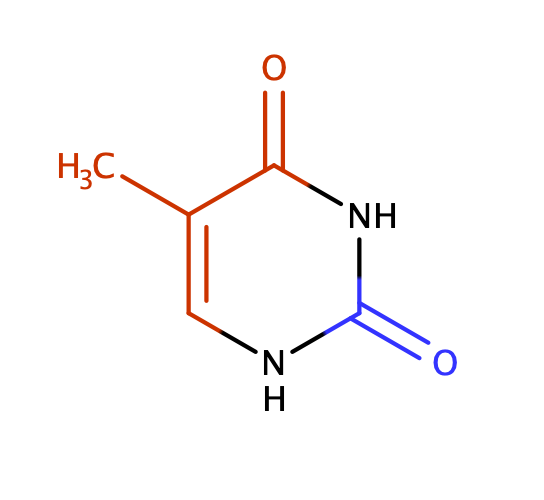
Красным цветом обозначены атом, ориентированные к большой бороздке, синим - к малой.
В сторону большой бороздки ориентированы атомы: [DT]27:B.C4,C5,C6,C7,O4
В сторну малой бороздки ориентированы атомы: [DT]27:B.C2,O2
| A-DNA | B-DNA | Z-DNA | |
|---|---|---|---|
| Тип спирали | Правая | Правая | Левая |
| Число оснований на виток | 11 | 10 | 12 |
| Шаг спирали () | 28,03 | 33,75 | 43,5 |
| Ширина большой бороздки | 16,81 G(5:A.P) | 17,21 G(25:B.P) | * |
| Ширина малой бороздки | 7,98 G(5:A.P) | 11,69 C(12:A.P) | 9,18 C(34:B.P) |
*Анализ структуры Z-ДНК вызвал больше всего вопросов, так что за вычисленное значение ширины бороздки я не ручаюсь, буду ждать комментария. Однако, как я понял из того, что прочитал в Интернете, у Z-формы отсутсвует большая бороздка, присутсвует только аналог малой. Об этом написано тут и тут.
Анализ структуры тРНК 1G59
Определение торсионных углов на примере нуклеотида 20G (Strand 1)>
| α | β | γ | δ | ε | ζ | χ | |
|---|---|---|---|---|---|---|---|
| tRNA(20G) | -66,2 | 172,9 | 62,0 | 87,9 | -158,6 | -79,5 | -164,1 |
Для сравнения приведу таблицу торсионных углов для каждой из форм ДНК, данные для которой получены так же с помощью пакета 3DNA
| α | β | γ | δ | ε | ζ | χ | |
|---|---|---|---|---|---|---|---|
| A-DNA(5G) | -57,2 | 175,6 | 33,3 | 84,4 | -143,5 | -64,1 | -162,0 |
| B-DNA(4G) | -62,9 | -179,9 | 57,2 | 155,7 | -155,3 | -152,5 | -93,4 |
| Z-DNA(2G) | 69,4 | -160,5 | -179,1 | 77,3 | -131,9 | -68,4 | 58,3 |
По полученным данным моя тРНК все же ближе к А-форме ДНК, хотя отдельные значения ближе к В-форме. В связи с выполнением этого задания возникла пара вопросов. Почему, например, некоторые значения углов сильно отличаются от указанных в презентации? Так, например, угол эпсилон у А-формы 178 в презентации, а программа определяет его в примерно тех же абсолютных, но противополжных по знаку значениях? Ну и внутри одной молекулы у одного и того же нуклеотида в разных положениях могут диаметрально отличаться значения углов. Казалось бы, что правильнее сравнивать средние значения, а не какие-то конкретные, и в таком случае стоило бы просто выбросить сильно отличающиейся данные или все же это было бы некорректно?
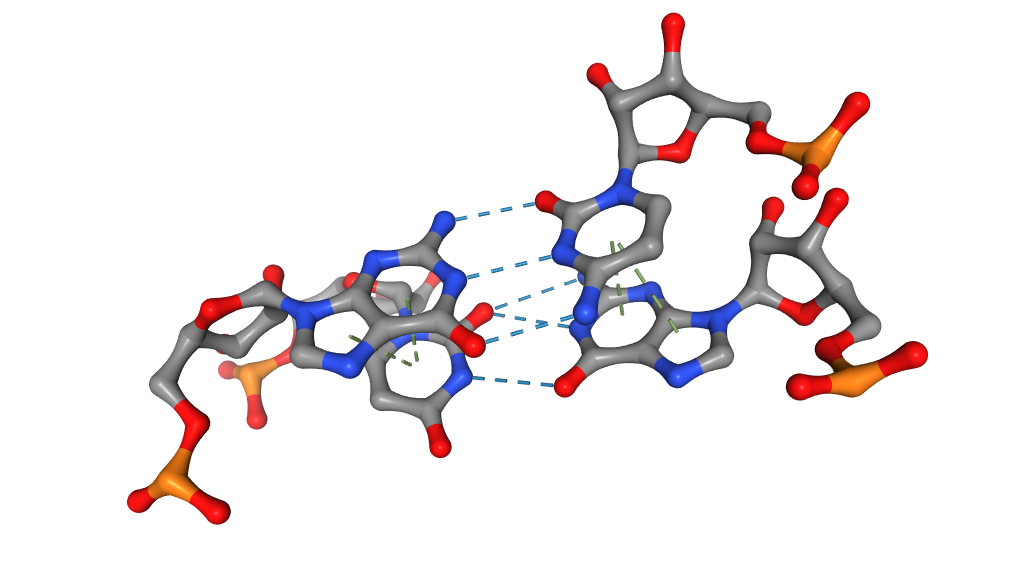
В качестве вспомогательного доказательства сходства именно с А-формой в самой выдаче имеется оценка каждого динуклеотида по ряду параметров. И в случае 1g59 все, за исключением некоторых не уотсон-криковских пар, описаны как принадлежащие к А-форме.
Стебли тРНК
В структуре имеется 4 стебля:
| Strand 1 | Strand 2 | |
|---|---|---|
| Стебель 1 | 501-507 | 566-572 |
| Стебель 2 | 549-553 | 561-565 |
| Стебель 3 | 538-544 | 526-532 |
| Стебель 4 | 510-514 | 521-525 |
Неканонические пары оснований
| Strand 1 | Strand 2 | |
|---|---|---|
| 1 | 502(G) | (U)571 |
| 2 | 554(U) | (A)558 |
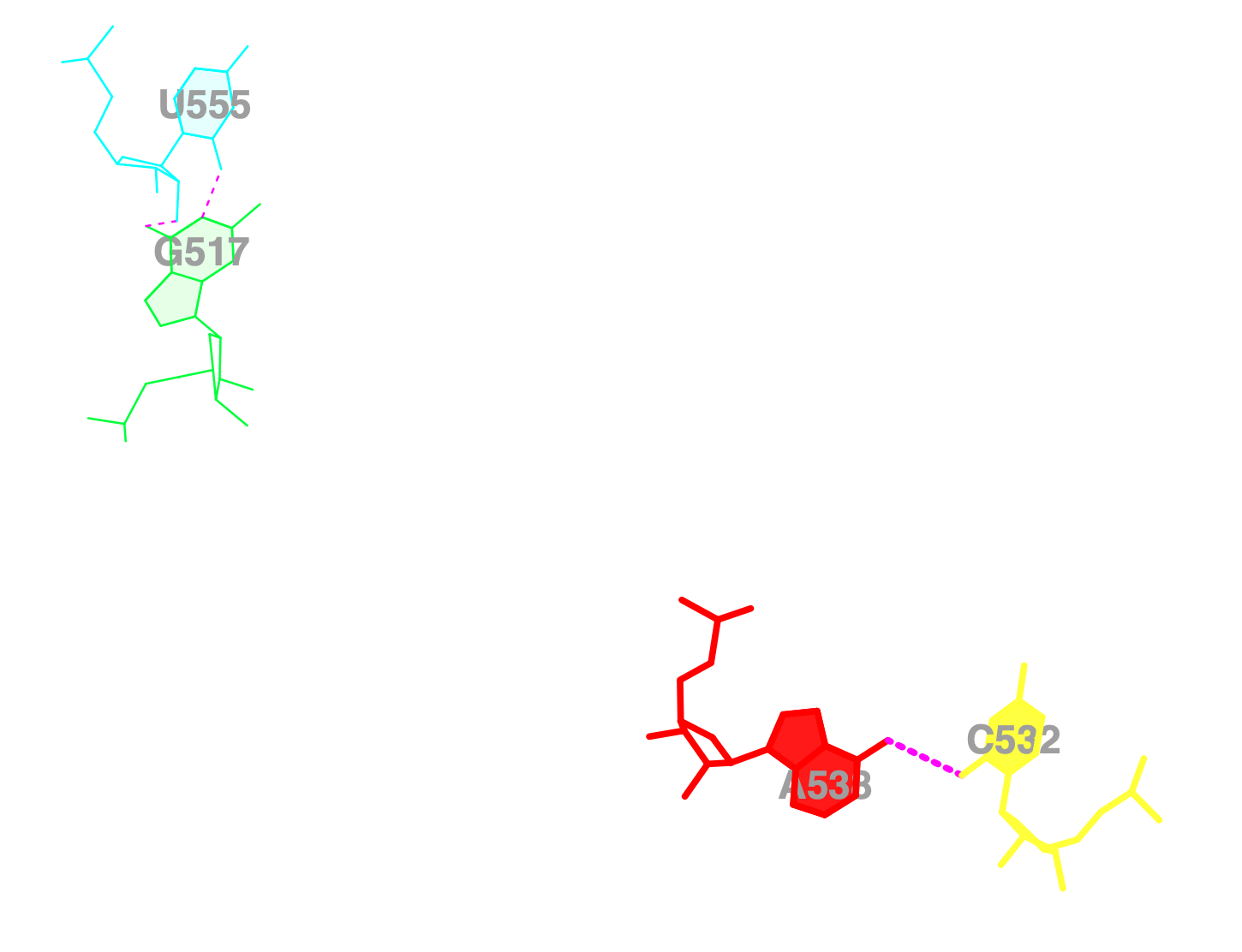
| 3 | 555(U) | (G)517 |
| 4 | 538(A) | (C)532 |
| 5 | 544(A) | (G)526 |
| 6 | 513(U) | (G)522 |
| 7 | 508(U) | (A)546 |
| 8 | 514(A) | (A)521 |
| 9 | 515(G) | (C)548 |
Выделенные жирным связи подсчитываются программой как не уотсон-криковские, но не совсем понятно из-за чего. В файле выдачи между между такими нуклеотидами имеются символы * и +, помимо обычных -.
Дополнительные водородные связи имеются у пары 518(G)-556(C), данная связь изолирована от остальных и не входит ни в одну из имеющихся петель.
Изображение стекинга
Я изобразил 3 наиболее перекрывающиеся группы оснований
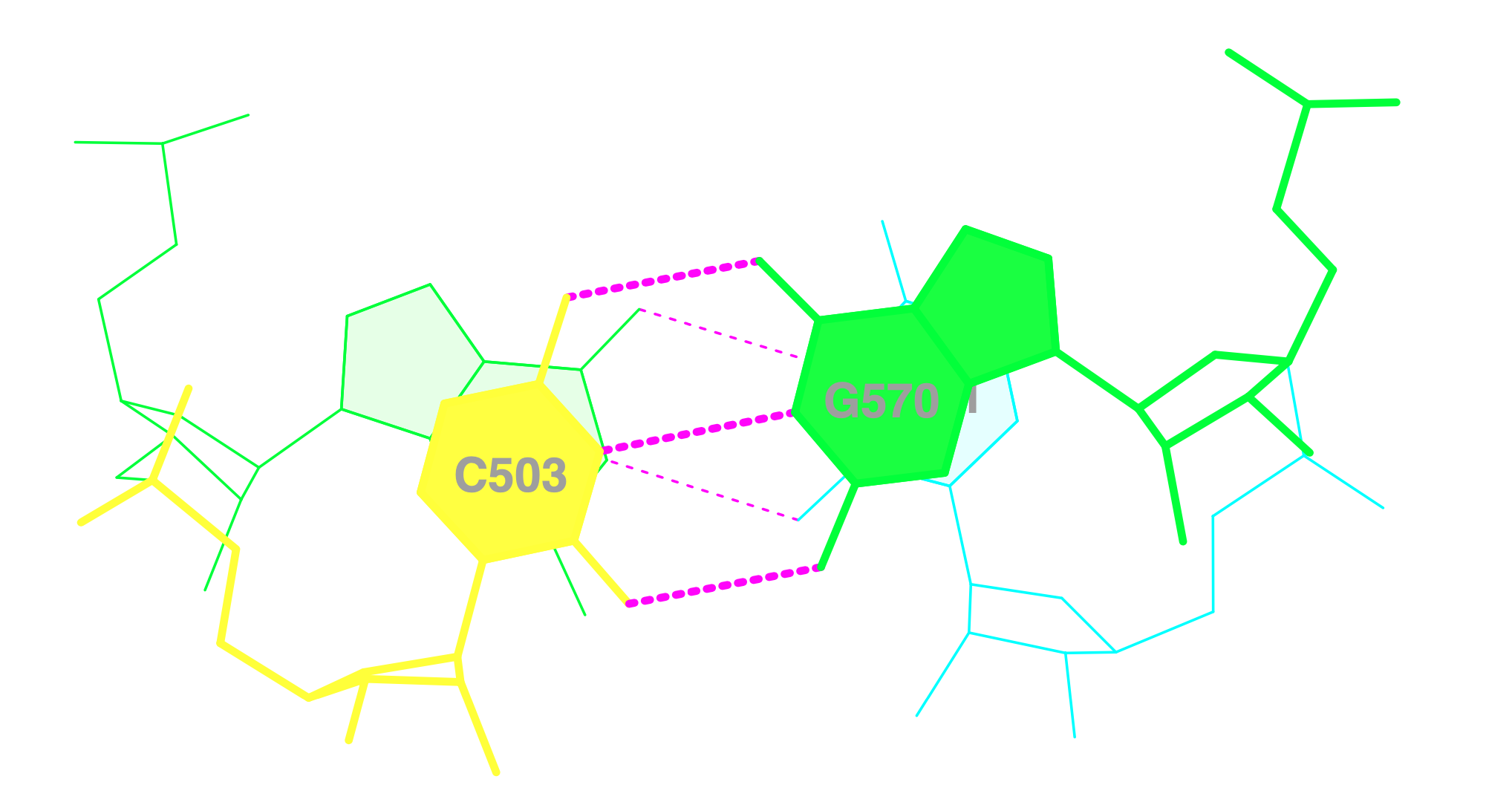
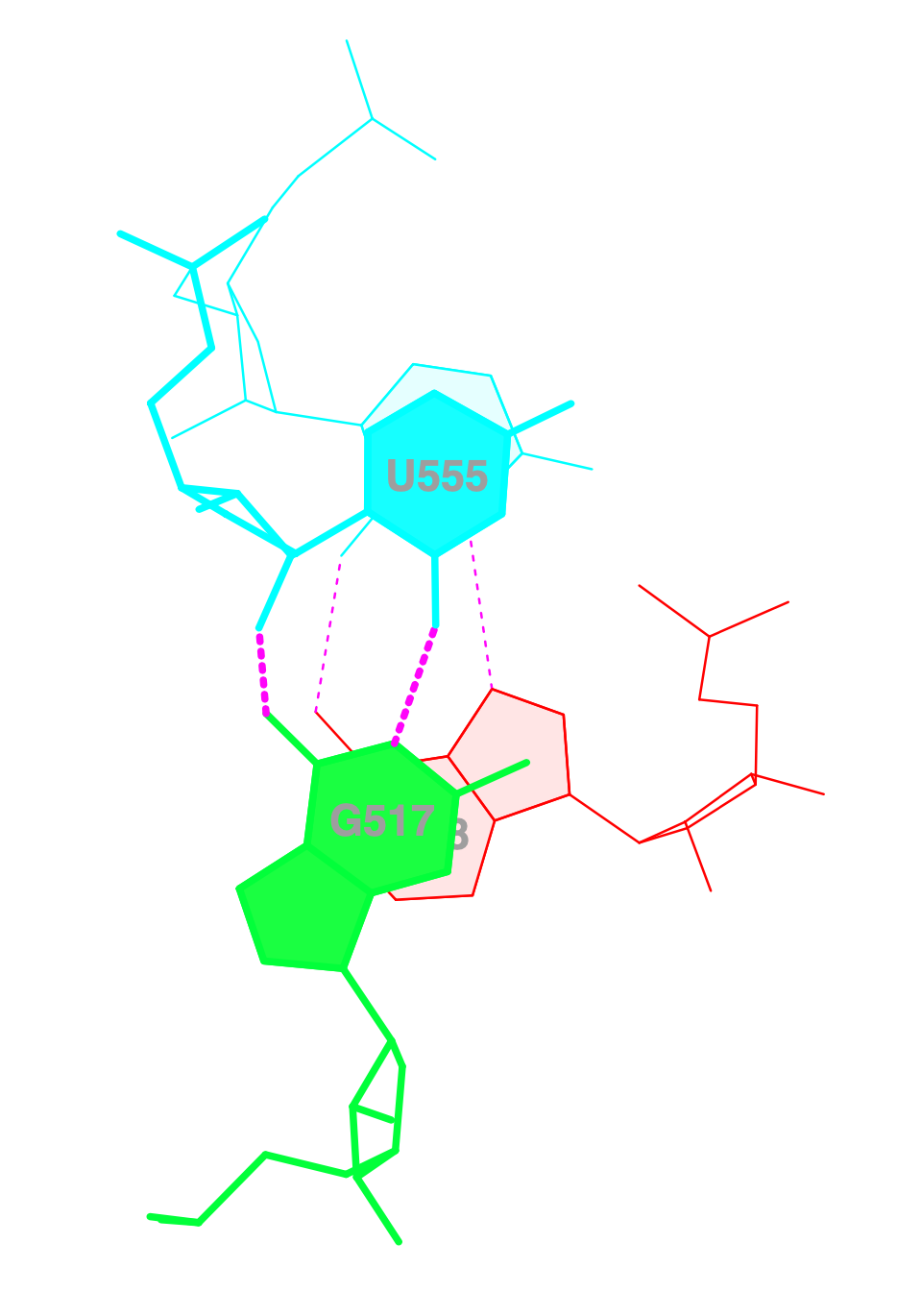
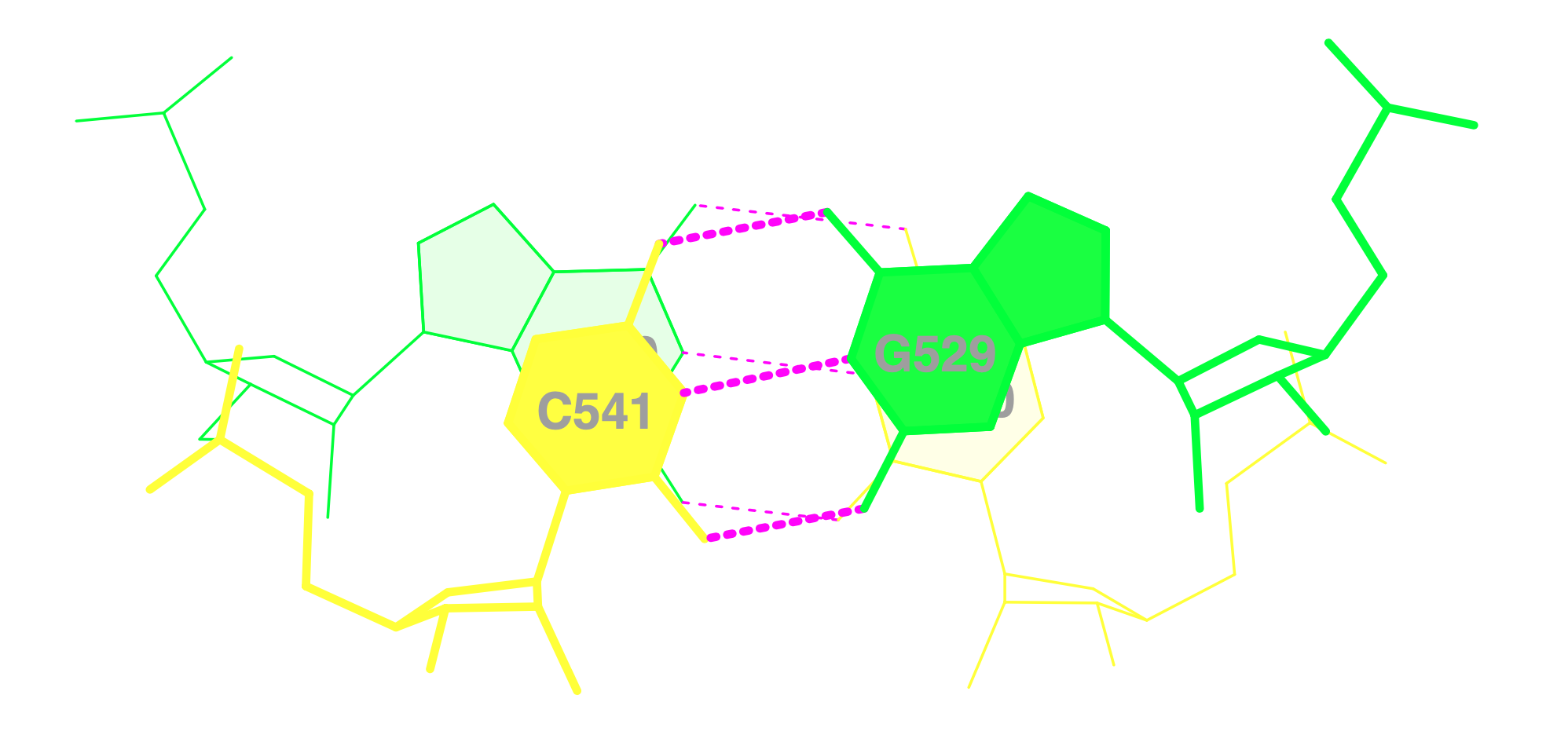
Для сравнения взглянем на группы с меньшей площадью перекрывания вплоть до нулевой:
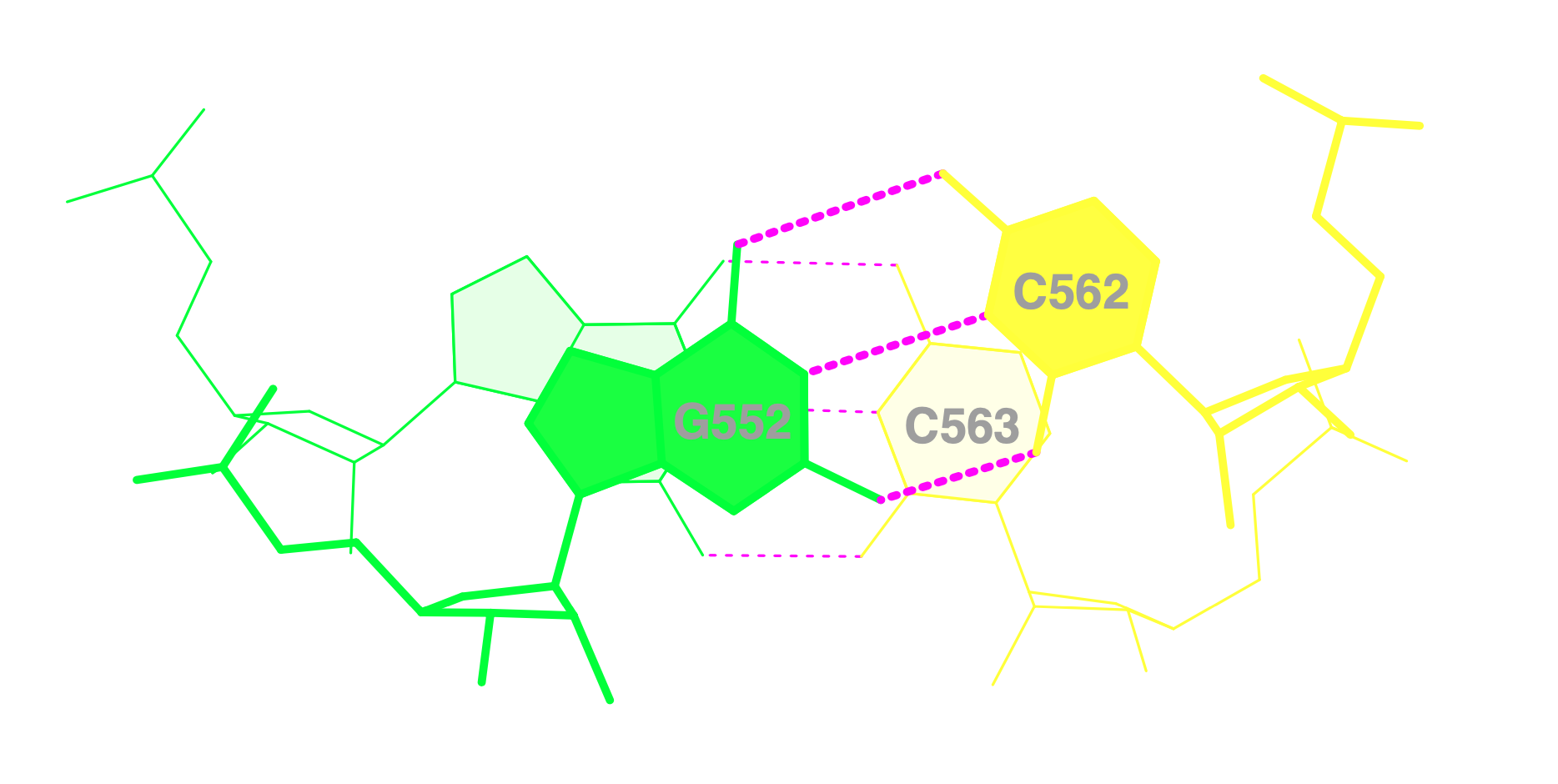
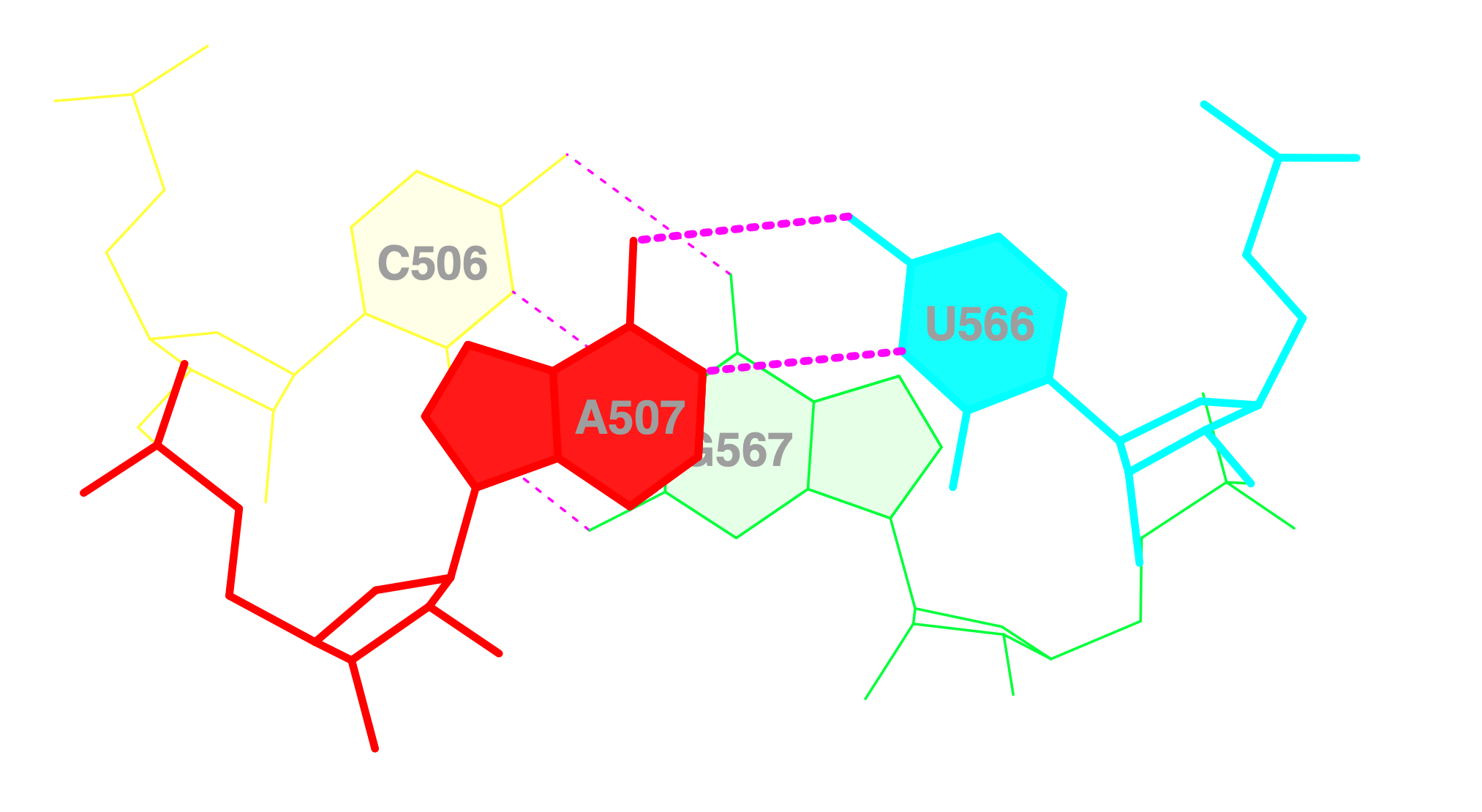
Завершим просмотром визуализации в NGLView двух моделей: с максимальной и минимальной площадями перекрывания