Геном синего кита
Для выполнения практикума я решил выбрать синего кита, так как животное это, конечно, поражающее своим масштабом, своей мощью и в то же время плавностью. К тому же, уровень качества сборки у него среди разных животных, которых я подбирал, у единственного был Chromosome (кстати, совсем недавно, 2 октября, она была аннотирована в RefSeq, а до этого в GenBank).

Немного о ките. Общеизвестно, что синий кит является самым большим животным на планете. При этом питается он планкотоном, которого отцеживает из толщи воды. Интересно, что несмотря на свои невероятные размеры, питается он так же, как и какой-нибудь двустворчатый моллюск. Еще одной особенностью, которая делает китов довольно интересным объектом для изучения - количество клеток. Понятно, что такие большие размеры связаны с куда большим суммарным числом клеток, а, соответственно, с намного большим числом делений. Увеличение числа делений сопряжено с увеличением числа ошибок, мутаций, закрепляющихся в некоторых клетках - а, соответсвенно, и с риском возникновения рака. Так вот, сущесвтует, так называемый, парадокс Пето, сформулированный в 1977 английским эпидемиологом статистиком Ричардом Пето. Пето писал, что хоть человек и намного больше мыши, что казалось бы, должно выливаться в более высокую частоту возникновения раковых заболеваний, в реальности этого не происходит. Подобная проблема отлично демонстрируется и существанием китов, слонов, чьи размеры и продолжительность жизни указвают на выработанные механизмы эффективной борьбы с развитием раковых клеток
Подробнее про это можно так же послушать в подкасте Ильи Колмановского "Голый землекоп" студии "Либо-либо"
В кариотипе у кита 44 хромосомы - в сборке 23. Но, как я понял, в данной сборке представлен гаплотип - то есть, только половина от общего числа, плюс половые хромосомы представлены обе - X и Y, так что получается как раз 22+1=23. В сборке так же присутсвует митохондриальная ДНК, но эта часть была аннотирована только в RefSeq.
Образец был собран в 2016 году на побережье Санта Барбары. Кит - самец, исследовали его фибробалст
Сборку я получил по запросу Balaenoptera musculus (Blue whale). При этом было получено два результата: выбранная сборка и еще одна, но тоже взятая из фибробласта этого же кита. Дело в том, что не случайно в первой сборке оказался гаплоидный набор - на самом деле, в технологии секвенирования третьего поколения есть возможность отделения гаплоидных хромосом - они получают название псевдогаплоидных согласно номенклатуре INSDC. При этом из одной соматической клетки получается возможным выделить два псевдогаплоидных набора. Один из них, более полный и точный называют principal pseudohaplotype - главный псевдогаплотип(?). Другой - альтернативным. Так вот, второй результат выдачи на мой запрос - как раз альтернативный псевдогаплоидный набор из той же клетки. Он же имеет уровень качества Scaffold, что указывает на то, почему он альтернативный, по сравнению с другим. У выбранного мной варианта уровень качества Chromosome - там собраны все или некоторые хромосомы, которые могут, при этом, содержать какие-то пропуски, отдельные кусочки последовательности.
Сборка является референсной, то есть вручную отобранная сборка, которая по сравнению с остальными существующими сборками генома этого организма является наиболее качественной.
| ID GenBank | GCA_009873245.3 |
| ID RefSeq | GCF_009873245.2 |
| Общий размер генома | 2.4 Gb |
| Scaffold N50 | 110.3 Mb |
| Scaffold L50 | 9 |
| Contig N50 | 6.3 Mb |
| Contig L50 | 103 |
Параметр N50 указывает размер контига/скаффолда, длиной меньшей или равной, чем у половины всех контигов/скаффолдов в сборке
Параметр L50 указывает наименьшее число контигов/скаффолодов, в которых содержится половина всех нуклеотидов сборки
Все файлы сборки
| Название файла | Описание |
|---|---|
| GCF_009873245.2_mBalMus1.pri.v3_cross_species_tx_alns.gff.gz | информация о наличии гомологичных генов у других организмов |
| Balaenoptera_musculus_AR100_annotation_report.xml | Отчет по аннотированию в формате XML, информация в числах о результате секвенирования (число экзонов, интронов, минимальные длины, медианные значения и много чего еще) |
| GCF_009873245.2_mBalMus1.pri.v3_assembly_report.txt | общая информация (организм, название проекта, методы сборки и секвенирования,...) и описание элементов, вошедших в сборку (длина, расположение, номера в GenBank и RefSeq, ...) |
| GCF_009873245.2_mBalMus1.pri.v3_assembly_stats.txt | описнаие каждой хромосомы из сборки (длина, число пропусков, значения N50, число скаффолдов, ...) |
| GCF_009873245.2_mBalMus1.pri.v3_cds_from_genomic.fna.gz | кодируемые участки в ДНК |
| GCF_009873245.2_mBalMus1.pri.v3_feature_count.txt.gz | кол-во каждого типа особенности |
| GCF_009873245.2_mBalMus1.pri.v3_feature_table.txt.gz | Таблица особенностей |
| GCF_009873245.2_mBalMus1.pri.v3_genomic.fna.gz | Последовательность генома |
| GCF_009873245.2_mBalMus1.pri.v3_genomic.gbff.gz | последовательности генома + описание каждого контига, структура каждого гена подробно описана |
| GCF_009873245.2_mBalMus1.pri.v3_genomic.gff.gz | описание генов внутри каждой хромосомы и их структуры (учатски экзонов, CDS, что кодируют, ..) + идентификационные номера |
| GCF_009873245.2_mBalMus1.pri.v3_genomic.gtf.gz | похожая таблица, но тут еще приводятся докозательства того, что последовательности получены правильно |
| GCF_009873245.2_mBalMus1.pri.v3_genomic_gaps.txt.gz | описание гэпов в последовательностях (границы, длина, тип и док-во, видимо, того, что этот гэп, действительно, отсюда) |
| GCF_009873245.2_mBalMus1.pri.v3_protein.faa.gz | последовательности протеома |
| GCF_009873245.2_mBalMus1.pri.v3_protein.gpff.gz | таблица особенностей для белков |
| GCF_009873245.2_mBalMus1.pri.v3_pseudo_without_product.fna.gz | последовательности псевдогенов |
| GCF_009873245.2_mBalMus1.pri.v3_rm.out.gz | сводная таблица по повторам |
| GCF_009873245.2_mBalMus1.pri.v3_rm.run | отчет программы RepeatMasker, которая находит в последовательности повторы характерные для предков или всех организмов, конкретного вида и определенной ветки |
| GCF_009873245.2_mBalMus1.pri.v3_rna.fna.gz | последовательность транскриптома* |
| GCF_009873245.2_mBalMus1.pri.v3_rna.gbff.gz | таблица особенностей мРНК |
| GCF_009873245.2_mBalMus1.pri.v3_rna_from_genomic.fna.gz | последовательности учатсков генома, кодирующих мРНК |
| GCF_009873245.2_mBalMus1.pri.v3_translated_cds.faa.gz | последовательности, полученные трансляцией CDS |
| README.txt | информация о файлах, которые есть в сборке (never too late...) |
| README_Balaenoptera_musculus_annotation_release_100 | информация про аннотацию (дата, авторы, идентификационные номера |
| annotation_hashes.txt | сводка дат внесения каких-то изменений в аннотацию |
| assembly_status.txt | статус версии аннотации |
| md5checksums.txt | контрольные суммы для каждого файла |
*показательно, кстати, что файл с геномом весит 716 МB, а с транскриптомом - 30
График
График я ограничил 30-ю первыми скаффолдами, так как дальше уже были совсеми маленькие элементы, которые только уменьшали масштаб графика
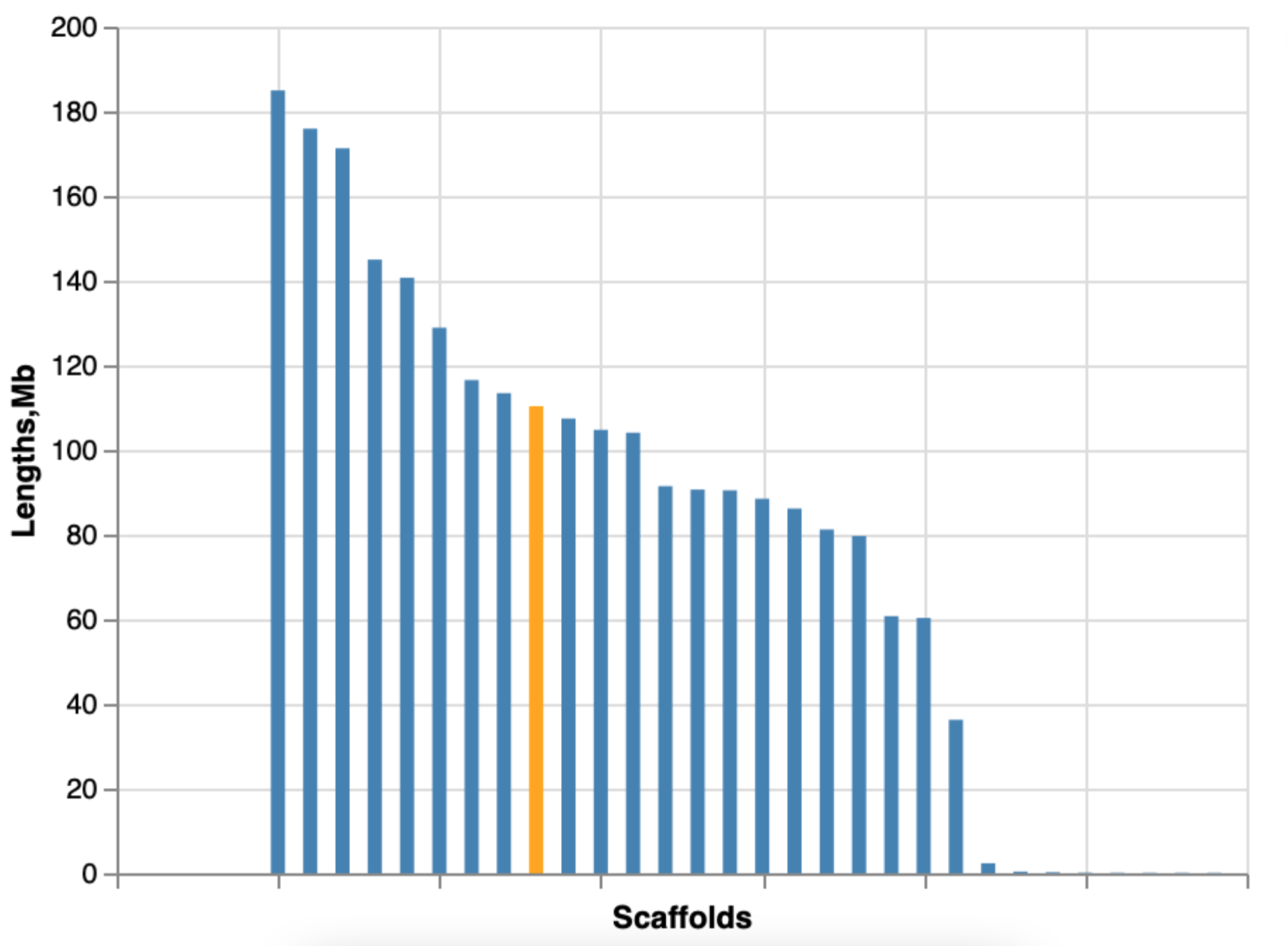
График получен с помощью этого скрипта (не знаю, верным ли было решение использовать altair, но что сделано, то сделано...)
Геном органелл
В этой сборке есть геном митохондрии - обнаружил я это просто пролистав список хромосом, включенных в сборку. Последняя оказалась с названием MT. На наличие дополнительного генома так же указывает то, что в описании сборки указаны 105 скаффолдов, из которых она собрана, но файл генома (FASTA) содержит в себе 106 записей
| Идентификатор записи | Тип органеллы | Число кодирующих последовательностей (CDS) | Число генов рРНК | Число генов тРНК | Число псевдогенов |
|---|---|---|---|---|---|
| NC_001601.1 | Митохондрия | 13 | 2 | 44 | 0 |