Работа в BLAST
Митохондриальный геном
Для выполнения первого задания я решил выбрать митохондрию - ее геном содержит 16402 пн.
Идентификационный номер - NC_001601.1. Последовательность в формате FASTA тут
График
Из графика имеем: 2 участка, кодирующих рРНК, 19 тРНК, 13 генов, один из которых на комплементарной цепи
Выравнивания
megablast
Для начала я попробовал по рекомендации исключить всех хордовых (taxid: 7711). В результате, получилось, что большая часть результатов покрывает введенную последовательность меньше, чем на 10%, а из те, что покрывает больше почти все созданы искусственно. Из природного мира наибольшим сходством при таком поиске обладает почвенный клещ Oppiella nova: покрытие 67%, E-value = 0, результат - 4032 бита. Причем, этот участок соответсвует ядерной ДНК, а не митохондриальной (т.к. содержит интроны).
При исключении позвоночных результат тот же. Убрав только млекопитающих, лучшим результатом стала рыбка Acanthochromis polyacanthus, а точнее ее митохондрия (причем, покрытие составило аж 94%).
Ради интереса я так же решил провереить тот факт, что синие киты наиболее близки к гиппопотамам из наземных млекопитающих. Так что я решил исключить только китообразных. И первым же в списке оказалась митохондрия карликового бегемота! (Hexaprotodon liberiensis).
Про родство китов и гиппопотамов можно почитать вот тут
Пример использования: Обнаружился какой-то участок, содержащий большое количество повторов. Проверить, является ли это распространенным явлением среди других видов или некоторой специфической особенностью
blastn
Тут среди не хордовых клещ снова вырвался в топ, но уже лучше - покрытие 95%, 75% идентичности
Пример использования: Тот же. Но нужно учесть, чсто алгоритм blastn более чувствительный
blastx
Для транлсяции я выбрал генетический год митохондрий позвонончых. Этот код отличается в следующих местах:
| Кодон | Обычный код | Митохондриальный |
|---|---|---|
| AUA | Ile | Met |
| AGA | Arg | STOP |
| AGG | Arg | STOP |
| UGA | STOP | Trp |
Тут поиск оказался очень требовательным, мне не удалось хоть как-то ограничить организмы, чтобы он не завершался ошибкой. Поэтому, к сожалению, выдача состоит только из китообразных. Судя по графику, сходные области соответсвуют двум генам: ND5 и COX1.
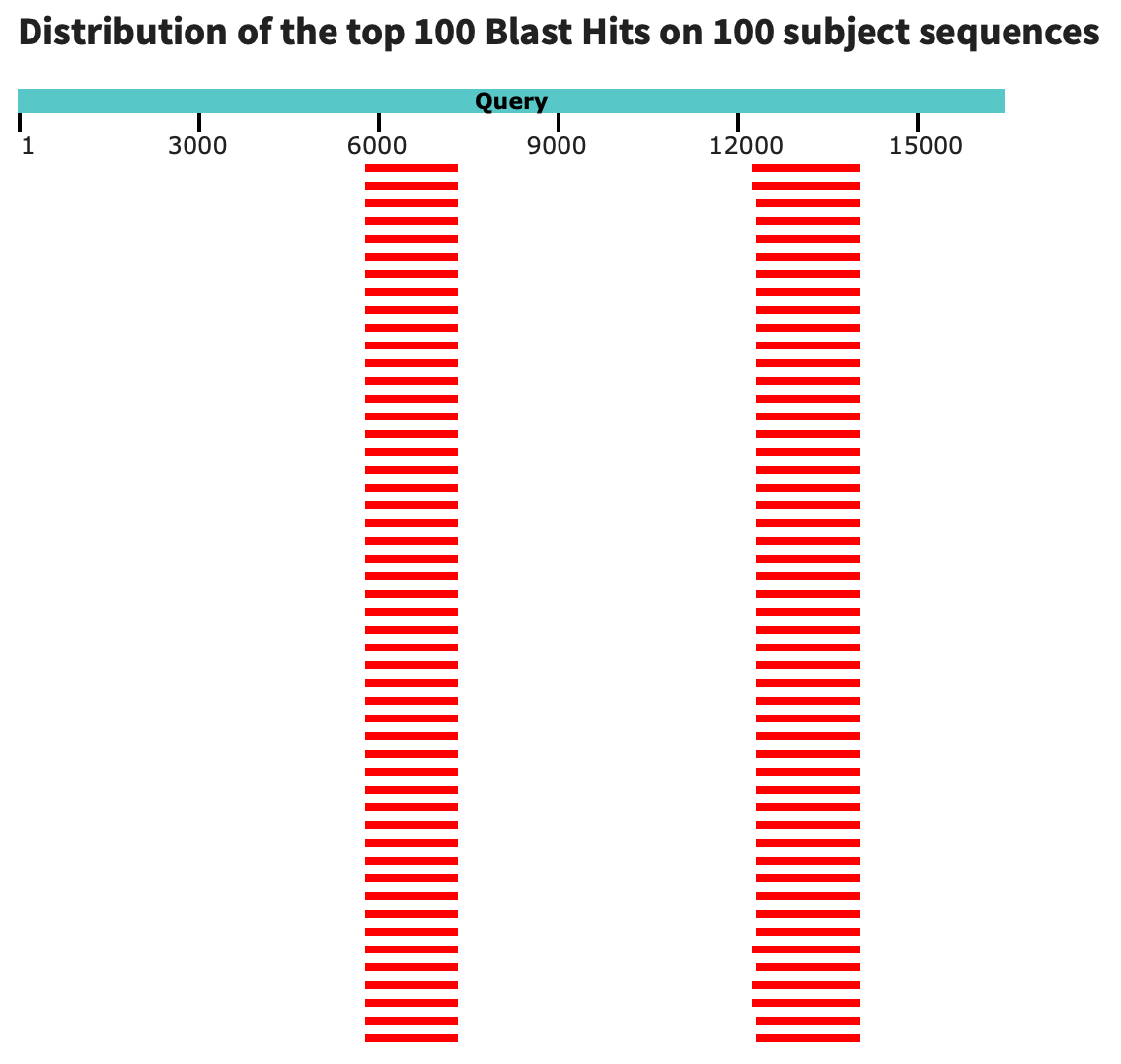
Пример использования: У бактерии нарушена работа фермента А. Известна последовательность, кодируящая его, но непонятно, из-за чего именно возникла поломка.
tblastx
К сожалению, этот алгоритм не выдал никаких результатов совсем. Я пробовал и применять обычный генетический код, и увеличивал количество выдаваемых результатов, и убирал какие-либо ограничения на организмы, но он все равно отказывался выдавать мне хоть один результат.
Пример использования:Попробовать найти гомологов среди далеких предков, так как последовательности аминокислот более консервативны по сравнению с нуклеотидными
Поиск гомологичных генов рРНК у далеких предков
Выданныые последовательности E.coli кодируют 16S и 23S рибосомную РНК. 16S - элменет малой субъединицы рибосомы, 23S - большой. Соответсвтенно, обе эти рРНК выполняют структурную функцию, выстраивая каркас рибосомы. Они взаимодействуют между собой, связывая субъединицы друг с другом.
16S рРНК так же содержит последовательность анти-Шайна-Дальгарно, обеспечивающую связывание с мРНК, участвует в инициации трансляции и обеспечивает правильное взаимодействие кодонов в A-сайте
23S рРНК участвует в связывании пептидил-тРНК в P сайте
blast
Для выполнения задания я выбрал blastn, поскольку он наиболее точный из нуклеотид-нуклеотидных алгоритмов. Команда следующая:
blastn -task blastn -query rRNA_ecoli.fasta -db kit.fasta -out blast.out -evalue 0.05 -word_size 7
В результате работы с 16S рРНК было получено 2 участка
Первый с 22119802 по 22119844 нуклеотид в хромосоме NC_045785.1. Этот участок соответсвует гену SESN2 (его части). Второй с 18256514 по 18256556 в хромосоме NC_045797.1. Там кодирующих учасков не обнаружено
В результате работы с 23S рРНК было получено 8 участков
Среди прочего там оказался и участок митохондриальной ДНК. И он как раз является частью кодирующей 16S рРНК. Интересно, что при работе с 16S E.coli этот участок не был получен)