Построение дерева по белковым последовательностям
В качестве потенциального гомологичного белка для выбранных ранее бактерий была взята АТФ-зависимая Clp протеаза E.coli
Результат работы blastp тут. Получилось 20 результатов, причем наиболее правдоподобными получились записи, содержащие именно этот белок у всех организмов, входивших в запрос
Дерево
Было реконструировано следующее дерево - Newick
Реконструкция проводилас методом минимальной эволюции (FastME) со следующими параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement". Бутстреп строится по 100 репликам.
Примеры паралогов: CLPX_MYCLE -- FTSH_MYCLE | RUVB_BIFLO -- CLPX_BIFLO | B0RHW4_CLAMS -- CLPX_CLAMS
Примеры ортологов: CLPX_MYCTU -- CLPX_MYCLE | Q8G871_BIFLO -- Q82EB8_STRAW | CLPX_CLAMS -- CLPX_LEIX
Дерево с окрашенными группами ортологов:
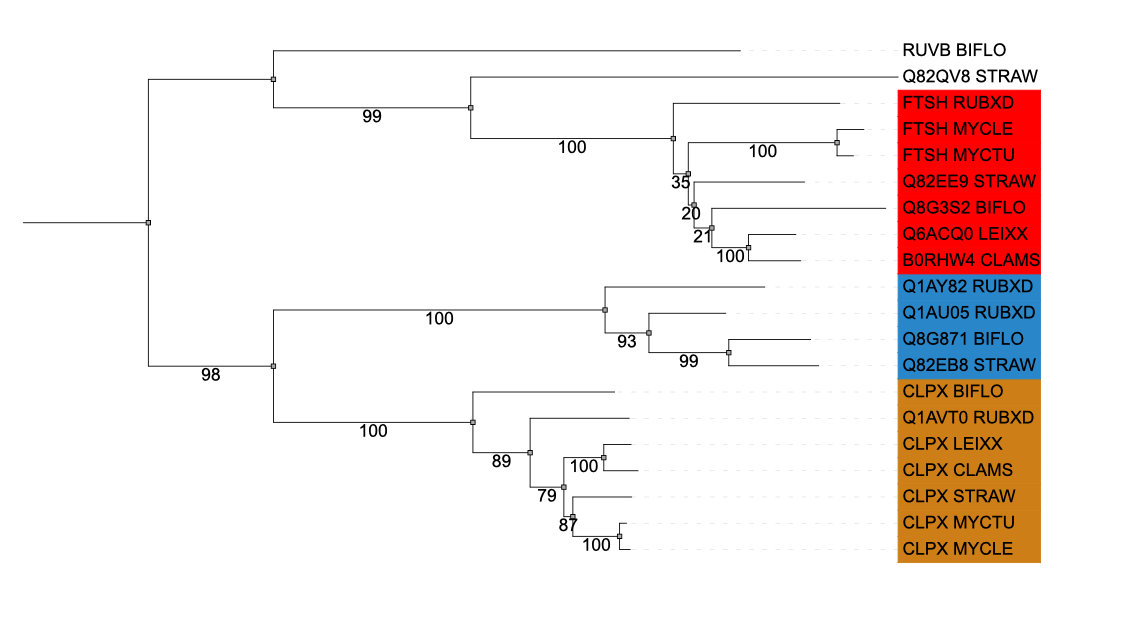
Группы схлопунты:
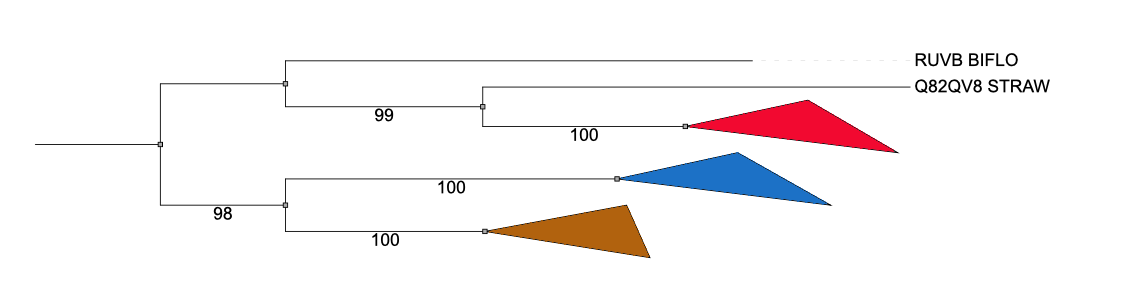
Нижнюю группу составляют АТФ-зависимые Clp протеазы. В синей кладе содержатся АТФ-азы ААА-2, протеазы. В красной - АТФ-зависимые цинковые металлопротеазы FtsH
Полученное дерево я сравниваю с полученным ранее деревом, построенным на основе 16S рРНК, так как оно наиболее репрезентативно в силу консервативности этой последовательности. В кладе АТФ-зависимых Clp протеаз есть нарушения, касающиеся очередности ответвлений: например, в этом дереве первыми ответвляются BIFLO,RUBXD,, а в дереве по 16S рРНК наоборот.
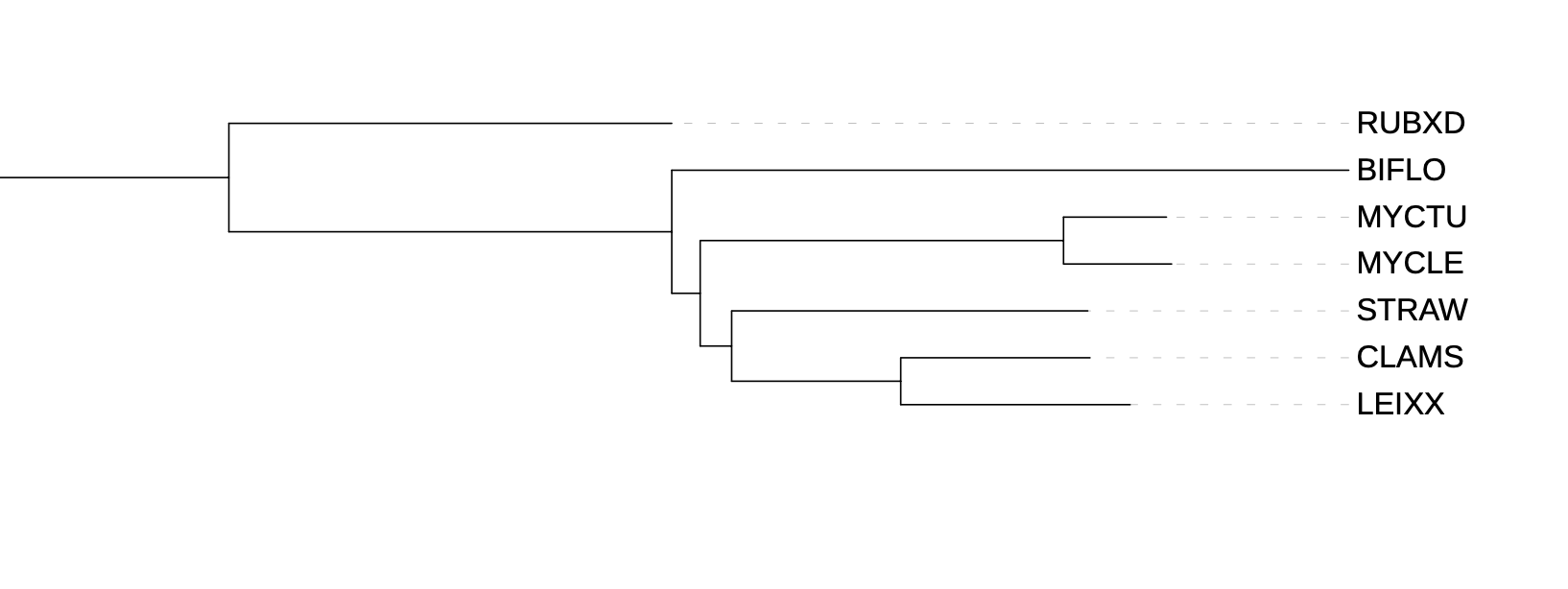
В группе АТФаз присутсвуют не все виды, но среди представленных последовательность ответвлений верная
В группе цинковых металлопротеаз последовательность нарушена, но представлены все