Практикум 6
Анализ сигналов терминации репликации E.coli
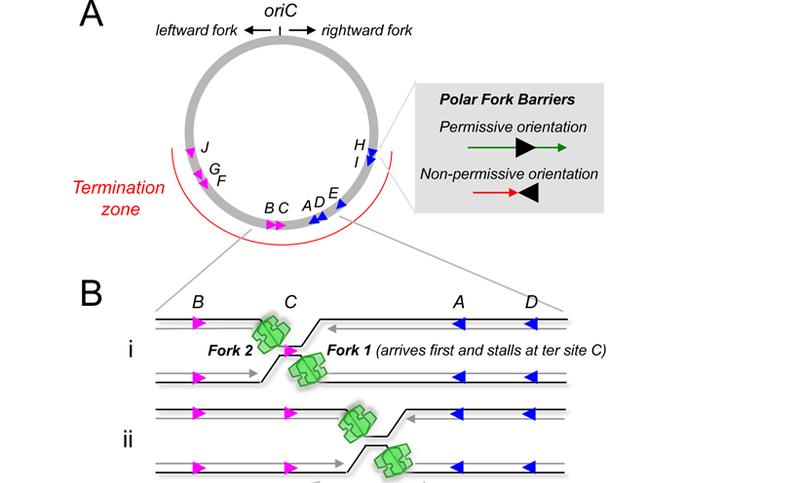
Репликация трансляции E.coli начинается из одного ориджина - oriC - и распространятеся в двух противоположных направлениях с помощью двух репликативных вилок. По мере прохождения циклической ДНК вилки входят в так называемую терминационную зону. Она характерна тем, что в ней находится 10 сайтов (ter сайты) связывания белков из семейства Tus (от terminus utilization substance). Интересно то, что белки эти работают аналогично диодам, пропуская репликативные вилки по направлению к сайту терминации, но не пропуская в обратном. 5 левых белков будут пропускать вилку, двигающуюуся против часовой стрелки, но уже не будутпропускать как ее в обратном направлении, так и другую вилку, идущую против часовой. Ровно наоборот будут работать 5 правых белков. Таким образом, вилки оказываются как бы заперты в этой зоне.
Функция таких сайтов до конца не ясна. Причем если удалить ген tus не будет возникать изменений, указывающих на аномально большие продукты репликации. Получается, ter-сайты не являются неотъемлимой частью терминации репликации. Было также показано, что терминация репликации у E.coli может происходить с образованием одноцепочечных обрывков ДНК на 3'-конце. В таком случае может произойти еще одна инициация репликации на этих участках. Причем в отсутсвие tus ее объемы намного больше. Поэтому, возможно, что функция этих участков именно в том, чтобы не допустить развития этой новой репликации.
Белок Tus связывается с сайтом в мономерном состоянии. Также было показано, что транскрипция этого белка регулируется через один из ter-сайтов - TerB. Ген tus находится дальше сайта TerB на 11 оснований. Связывание белка в этом сайте блокирует сайт посадки рибосомы и участок промотора, таким образом блокируя наработку белка.
Источники информации
1. Dewar JM, Walter JC. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 2017 Aug;18(8):507-516. doi:10.1038/nrm.2017.42. Epub 2017 May 24. PMID: 28537574; PMCID: PMC6386472
2. Neylon C, Kralicek AV, Hill TM, Dixon NE. Replication termination in Escherichia coli: structure and antihelicase activity of the Tus-Ter complex. Microbiol Mol Biol Rev. 2005 Sep;69(3):501-26. doi: 10.1128/MMBR.69.3.501-526.2005. PMID: 16148308; PMCID: PMC1197808
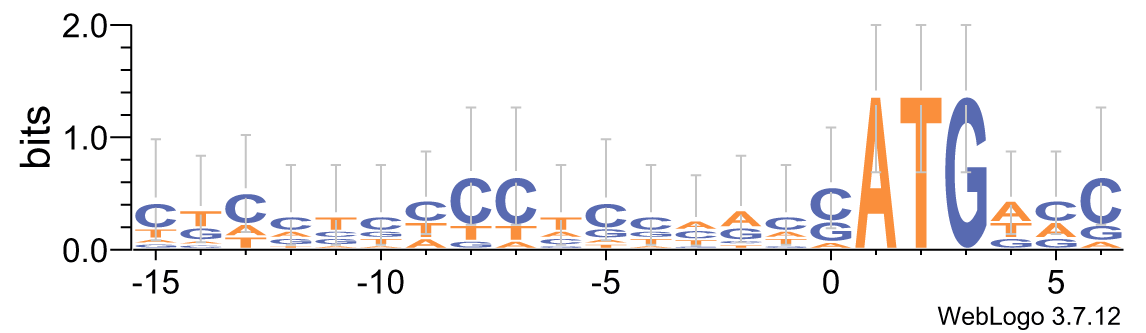
Анализ последовательности Козак
Я взял гены человека, таблицу, приведенную в указаниях к заданиям. Случайным образом выбрал 9 генов, на основе которых строил модель обучения. Я взял участок суммарно в 21 нуклеотидов: 15-3-3. Центральная тройка соответсвует старт-кодону. На основе полученных последовательностей построил выравнивание без гэпов и матрицу весов (PWM).Не совсем понятно, правильно ли я сделал, но псевдоотсчеты я поправил единой для всех "прибавкой" (эпсилон из лекции) в 0.0001. Возможно, теоретически неправильно делать ее одинаковой для A/T и G/C. И еще непонятно, надо ли это делать если речь идеть о старт-кодонах, в которых вариативности не должно быть у человека, если я правильно понимаю. То есть отсутсвие на первой позиции кодона чего-то, кроме аденина, вызвано не малым размером выборки, а правилом генетического кода (аналогично для остальных позиций в старт-кодоне).
Таким же образом я собрал 8 аналогичных участков из других генов. И собрал еще 8 последовательностей случайных участков той же длины, которые не содержали в себе старт-кодоны. Отдельно я этого не проверял, но посмотрю, будут ли какие-то помехи в связи с этим в результатах. Надеюсь, что мне повезло и участки подходят. Построил гистограммы:
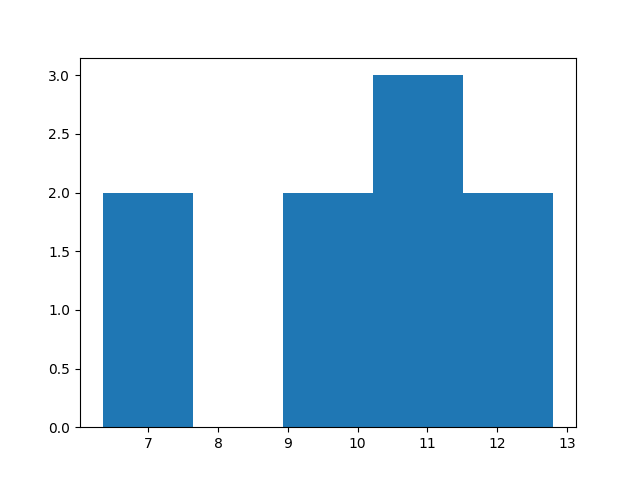
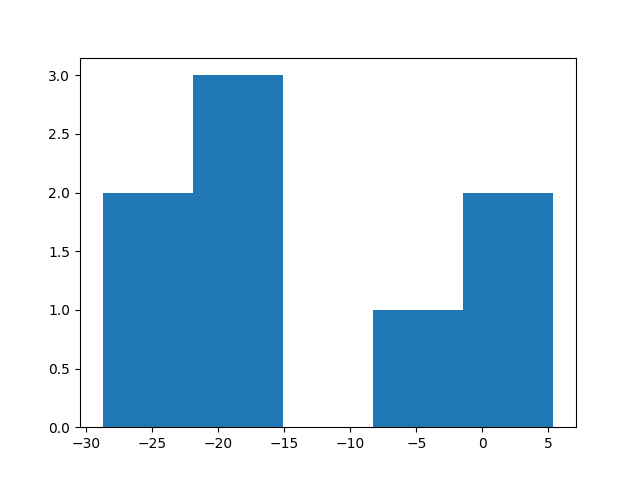
Маловато я взял последовательностей для анализа, из-за того, что вручную выполнял команды в баше, а надо было все же поместить все в скрипт. В целом, видно что веса получились достаточно маленькими. Думаю, что дело может быть в слишком большом хвосте, который я выбрал для окрестности старт-кодона. В 15 предшествующих нуклеотидах меньше половины должно содержаться в консенсусной последовательности, а требований к хоть какой-то схожести среди остальных нет. Тем не менее все равно видно, что случайные участки имеют намного меньший вес, за исключением одного. Но этот участок не соответсвует гену, так что просто совпало.
Порог определять, полагаю, смысла тут нет
Можно выделить следующие минусы в моем выполнении работы (которые я при необходимости исправлю, но публикую что-то для зачета):
Далее я построил матрицу и лого на основе выравнивания модели обучения.