Анализ белкового мотива
Я выбрал семейство Cid1 poly A polymerase. Выравнивание seed доступно по ссылке. В выравнивании выделяется от трех мотивов (возможно, 4-5). Я решил поработать с самым первым из них, в самом начале последовательностей (Рис. 1)
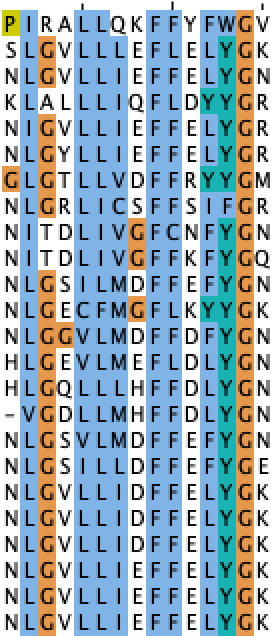
Его паттерн - [ILV]-G-x-[ILV]-[ILVWA](2)-x-[FL](2)-x-[ILFY](2)-G(0,1)
Возможно, он слишком детилизрованный и надо было как-то построже быть, но при попытке найти мотив такого паттерна в выравнивании находится 34 последовательности (всего 77), причем все они расположены в правильной позиции - в начале.
Результат поиска по базе Swiss-Prot доступен для загрузки по ссылкечс. Нашлось больше 400 записей со сходными мотивами, причем все они из совершенно разных ссемейств белков. Надо полагать, найденный мотив не является специфичным для конкретно моего семейства, что вполне нормально, так как среди белков есть какие-то широкораспространенные функции. Вот, возможно, мой мотив является сайтом какого-то "популярного" процесса.
Построив отдельное дерево, я выделил кладу, в которой, к сожалению, не нашлось уникального мотива, но ранее выбранный мотив более консервативен. Этого, в целом, можно было ожидать, так как описание мотива во всех последовательностях должно быть более общим, чем в некотором связанном наборе. (Рис. 2)

PSI-BLAST
Для работы выбрал идентификатор P19954.
Это хлоропластный фактор, связывающий рибосому, PSRP1. Последовательность получена на основе образца из клеток шпината, содержит 302 ак. Участвует в регуляции трансялции, блокируя связывание тРНК в A сайте.
Результаты в таблице. Удалось стабилизировать поиск после третьей итерации. Интересно, что плохая находка ниже порога нашлась только на второй итерации - ее не было ни до, ни после. Ступенька получилась довлольно большой - на второй итерации разница в 9 раз.