HMM-профиль
Выбор домена и доменной архитектуры
Для работы я выбрал домен, чье название на английском звучит так - Holliday junction recognition protein-associated repeat. Accesion записи - PF12346. Название - HJURP_mid. В таблице ниже приведены значения по критериям отбора для данной записи:
| Критерий | Значение |
|---|---|
| Пос-ти в full | 141 |
| Средняя длина | 114.2 |
| Среднее сходство | 63% |
| Средний процент покрытия | 16.54 |
| Число архитектур | 6 |
Доменная архитектура содержит 26 последовательностей и включает два домена. Схема: HJURP_mid, HJURP_C x 2.
В файле содержатся полные последовательности из full
Выравнивание
При получении последовательностей белков выяснилось, что на данный момент четыре из них удалены из UniProtKB и находятся в UniParc. Так что их последовательности в выравнивании не представлены. Само выравнивание построил в JalView методом Muscle (результат до удаления сходных). В нем я решил не удалять никакие последовательноси, поскольку все они были примерно одинаковой длины. Единственное две строки (перемещены в самый низ) содержат крупные делеции, особенно самая последняя - S7N4W0_MYOBR. (летучая мышь Ночница Брандта). Однако после корректировки избыточности ее последовательность удаляется, так что в итоговом варианте ее уже нет. Порог выбрал равный 80%. В результате осталось 12 последовательностей.
По выборке я построил HMM-профиль (HMM). Осуществил поиск и, в результате, получил таблицу, доступную к скачиванию по ссылке.
Оценка порогового значения
По весам полученных находок я построил гистограмму (Рис. 1).
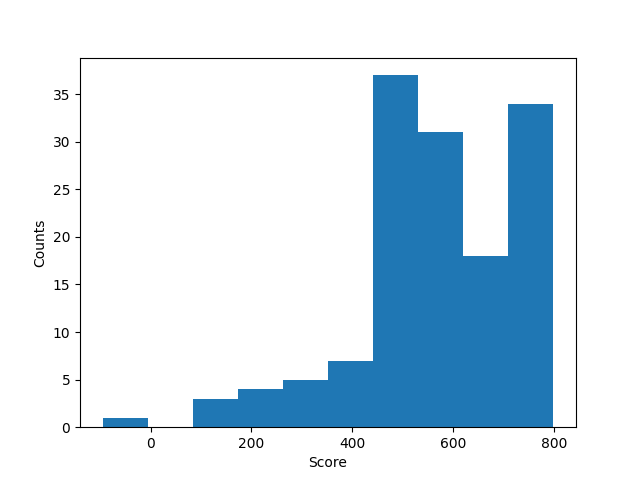
Далее я построил кривую ROC и кривую значений F1 (Рис. 2). Возможно, я чего-то не понял, но минимальное значение F1 получается у меня при минимальном весе, что не указывает на корректный порог. К тому же в интернете (например, в википедии) пишут, что более высокое значение F1 означает более качественный и точный тест. Поэтому порог буду определять по самому большому значению, которое у меня получилось.
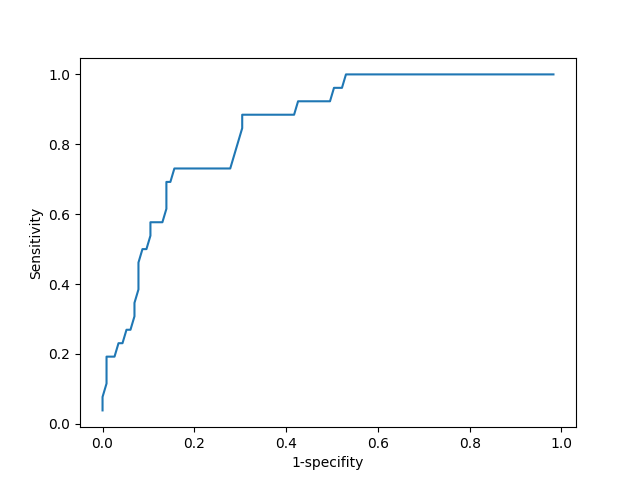
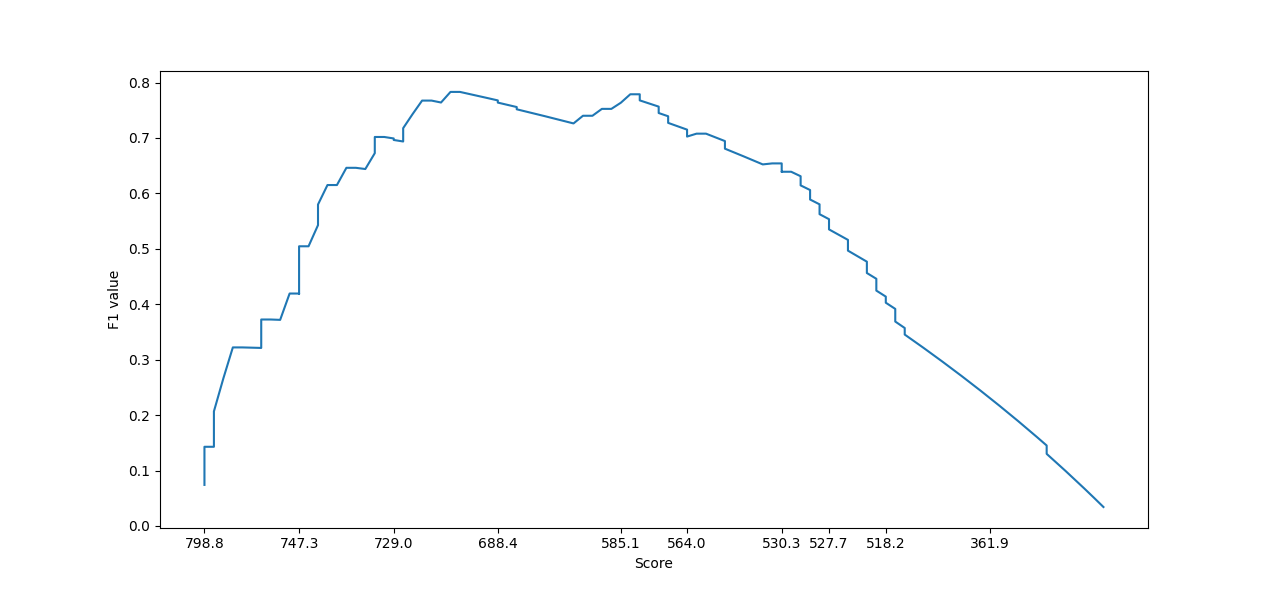
Самое большое значение F1 - 0,783. Оно соответсвует двум точкам веса - 704,7 и 709. При этом специфичность 84%, а чувствительность - 73. Результат, конечно, сомнительный. В общем, мой HMM профиль оказался не очень хорошим. Я оставил при его построении консервативные фрагменты (длиной в несколько десятков аминокислот), не входящие в состав доменов. Возможно, дело в этом.