При изучении данного документа можно найти не только информацию о его трехмерной структуре, но и ряде других полезных вещей. Этот файл содержит информацию о глутаминовой тРНК-синтетазе ассоцированой собственно с глутаминовой тРНК, в документе содержится две цепи A и C белка, а также две цепи тРНК с идентификаторами B и D (в дальнейшем мы будем рассматривать только одну из них, а именно цепь с идентификатором B , так как вторая аналогична).
Для того, чтобы нам сразу было удобно работать только с одной тРНК в RasMol, после удаления всех остальных молекул, была выполнена команда:
save pdb 1G59.pdb
Структура тРНК взята из организма THERMUS THERMOPHILUS и описана в записи PDB.
Ниже приводитсянуклеотидная последователность исследемой тРНК:
75 G G C C C C A U C G U C U 75 A G C G G U U A G G A C G 75 C G G C C C U C U C A A G 75 G C C G A A A C G G G G G 75 U U C G A U U C C C C C U 75 G G G G U C A C C AC помощью команды grep проанализировали как пронумерована цепь РНК в записи ( а именно использовалась команда grep -n 'ATOM' .* P .*' pdb1g59.ent > atoms. txt ), видим, что нумерация идет от 501 до 576 номера, но оснований только 75, одно основание пропущено, а именно под номером 547. В состав данной РНК не входят модифицированные основания, более того из других молекул кроме двух цепей белка и двух цпей РНК в файле не содержится других структур кроме воды.
Результаты программы find_pair
Запустили программу find_pair командой: find_pair -t 1j59.pdb stdout | analyze
Получили файл 1j59.out, который несет информацию о различных, интересующих нас побробностях, И главное, что интересно файл содержит информацию о спиральных участках (двойные спирали). То, что РНК складывается в двойную спираль, дает болшое преимущество, так как расчитывать для нее взаимодействия значительно легче, чем для неопределенной структуры. Кроме файла с расширением out, были получены и некоторые другие, как оказалось один из файлов, называвшийся col_helices.spt содержал информацию о том какие нуклеотиды входят в спирали и каким они соответствуют на комплементарной цепи, более того, сам файл - есть скрипт, для окрашивания в RASMOL этих спиралей, что значительно облегчает нашу работу. Ниже приведена последовательность с раскрашеными участкам, соответствующими спирали:
GGCCCCA UC GUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAANC GGGGGUUCGAUUCCCCCUGGGGUCACCA
Конечно такое изображение расположения спиралей мало чего дает, так как не очень понятно, как это все на самом деле выглядит, можем сказать что, молекула сложно устроена и содержит три спиральных участка, правда одна спираль состоит из одной пары нуклеотидов, что довольно старнно, по-видимому, торсионные углы для нее соответствуют торсионным углам для какого-либо типа спирали. При дальнейшем рассмотрении файла 1j59.out, мы исследовали торсионные углы для участков, которые программа идентифицировала, как спиральные и обратили внимание, что при определенном приближении эти участки все соответствуют А-форме спирали, посмотрев далее мы нашли информацию о том, что считает прогрмма о форме спирали, как оказалось это действительно была А -форма, к тому же известно, что для РНК другие формы гораздо менее устойчивы.
Для того, чтобы теперь лучше представить о пространственном расположении данной молекулы мы воспользовались программмой RASMOL, ниже приведен рисунок в котором спирали отмечены теми же цветами, что и в вышележащей схеме:
Исследуя данную структуру в RASMOL (также можно использовать файл 1j59.out), можно ныйти не только обычные взаимодействия (Утсон-Криковские), но и стекинг-взаимодействия между основаниями (их несколько), ниже приведен яркий пример, два последовательно идущих основания связаны стекинг-взаимодействиями (подписаны номера атомов):
Также определенный интерес предствляют водородные связи между основаниями, не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований. В файле анализа мы можем посмотреть и найти, что есть специальная снока, что в данной молекуле содержатся основания связанные не канонически.
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.011) B:.501_:[..G]G-----C[..C]:.572_:B (0.010) |
2 (0.008) B:.502_:[..G]G-*---U[..U]:.571_:B (0.014) |
3 (0.010) B:.503_:[..C]C-----G[..G]:.570_:B (0.007) |
4 (0.009) B:.504_:[..C]C-----G[..G]:.569_:B (0.009) |
5 (0.011) B:.505_:[..C]C-----G[..G]:.568_:B (0.007) |
6 (0.011) B:.506_:[..C]C-----G[..G]:.567_:B (0.007) |
7 (0.011) B:.507_:[..A]Ax----U[..U]:.566_:B (0.013) |
8 (0.008) B:.549_:[..G]G-----C[..C]:.565_:B (0.010) |
9 (0.006) B:.550_:[..G]G-----C[..C]:.564_:B (0.012) |
10 (0.007) B:.551_:[..G]G-----C[..C]:.563_:B (0.010) |
11 (0.008) B:.552_:[..G]G-----C[..C]:.562_:B (0.009) |
12 (0.010) B:.553_:[..G]G----xC[..C]:.561_:B (0.010) |
13 (0.016) B:.554_:[..U]U-*--xA[..A]:.558_:B (0.008) |
14 (0.017) B:.555_:[..U]Ux**+xG[..G]:.517_:B (0.015) x
15 (0.008) B:.538_:[..A]A-*---C[..C]:.532_:B (0.013) |
16 (0.009) B:.539_:[..G]G-----C[..C]:.531_:B (0.011) |
17 (0.012) B:.540_:[..G]G-----C[..C]:.530_:B (0.010) |
18 (0.012) B:.541_:[..C]C-----G[..G]:.529_:B (0.011) |
19 (0.011) B:.542_:[..C]C-----G[..G]:.528_:B (0.006) |
20 (0.008) B:.543_:[..G]G-----C[..C]:.527_:B (0.011) |
21 (0.007) B:.544_:[..A]Ax*---G[..G]:.526_:B (0.006) |
22 (0.010) B:.510_:[..G]G-----C[..C]:.525_:B (0.010) |
23 (0.014) B:.511_:[..U]U-----A[..A]:.524_:B (0.014) |
24 (0.012) B:.512_:[..C]C----xG[..G]:.523_:B (0.008) |
25 (0.017) B:.513_:[..U]U-**+xA[..A]:.546_:B (0.009) |
26 (0.009) B:.514_:[..A]A-**-xU[..U]:.508_:B (0.013) |
27 (0.008) B:.515_:[..G]Gx**+xC[..C]:.548_:B (0.010) x
28 (0.009) B:.518_:[..G]G-----C[..C]:.556_:B (0.015) +
Возьмем первую из списка пару, которая не является канонической G-U:
Аналогичные структуры мы можем найти рассматривая и другие пары из таблицы, помеченные звездочкой.
Программа einverted
Для сравнения приведем результаты программы einverted из пакета EMBOSS,, которая позволяет найти инвертированные последовательности в НК. Программа запускается простой командой:
einverted 1fcw.fasta - предварительно создали файл с фаста последовательностью РНК (она ктати практически приведена в начале данного задания).
Программа сама спросит о большинстве необходимых услвиях. При стандартных условиях поиск ни к чему не привел, при снижении порога до 15 получили один из участков нашей РНК, который действительно был спиральным ( правда из-за того, что пропущено одно основание, несколько смещена нумерация):
: Score 15: 5/5 (100%) matches, 0 gaps
3 cccca 7
|||||
69 ggggt 65
Несколько странно, что мы не получили второго довольно большого участка, хотя и еще понижали порог.
Программа mfold
Довольно хорошие результаты предсказания спиральных участков дала программа mfold, однако там были показаны не все связи. Программа запускалась при помощи команды:
mfold SEQ=my.fasta P=15
Параметр P показывает, на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального. Но мной было выбрано первое изображение, так как остальные менее подходили под указаные в файле о спиральных участках сведения. Ниже приведено изображение ввиде клеверной структуры:
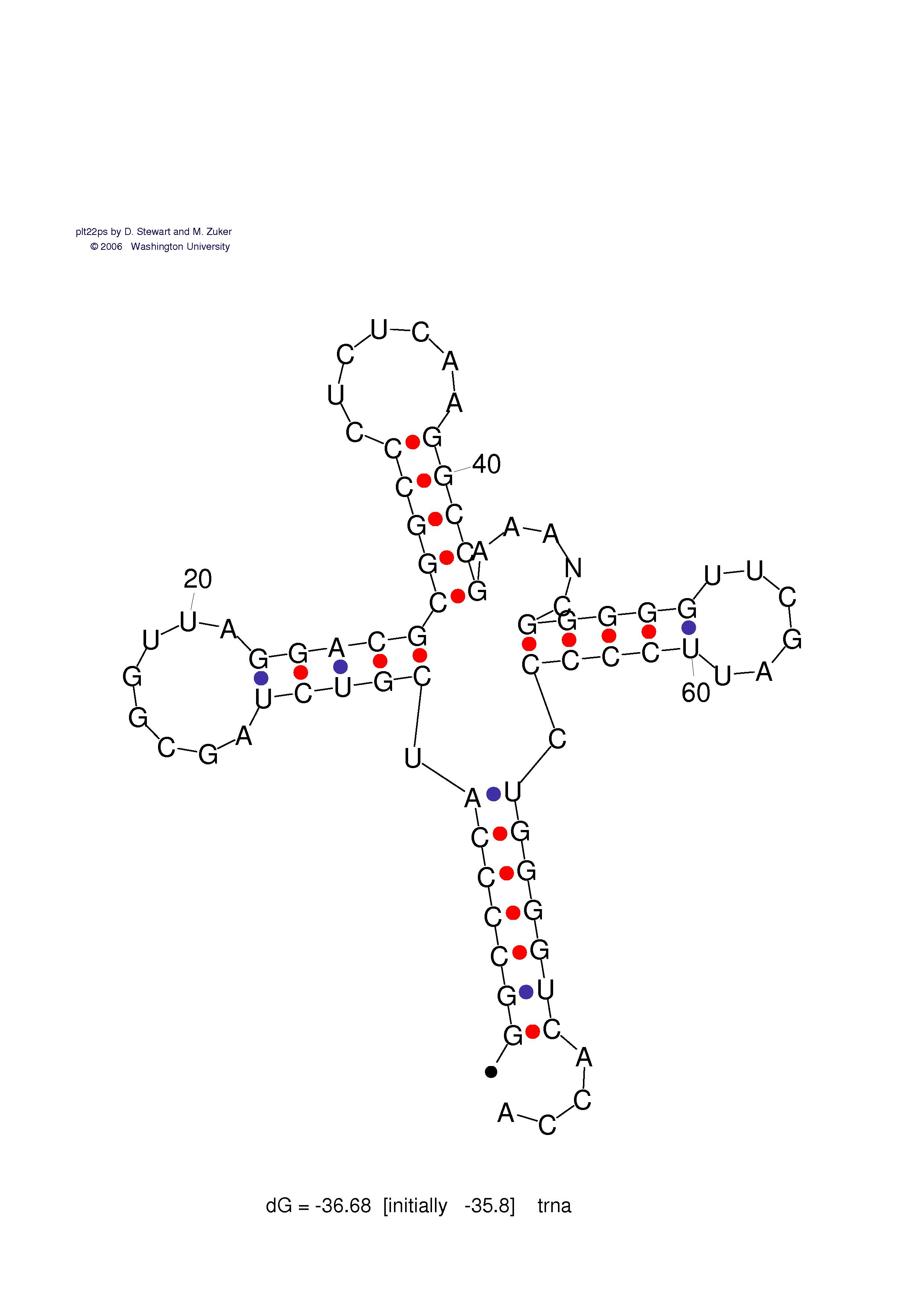
Если сравнить полученное изображение с возможным при использовании известных координат расположения спиралей, то мы найдем значительную степень сходство, да и представить себе что либо более стоящее на бумаге дотаточно сложно без всякого рода перекрываний, так как структура РНК все-таки пространственная, главное надо отметить, что основные параметры, указывающие на то, что это тРНК есть, посмотрев в литературе канонический вид тРНК, я убедился что этот первый вариант, полученный программой является довольно правдоподобным, и основные позиции там указаны верно. Таким образом следует признать, данная программа справилась с поставленной здачей. В следующем файле, вы можете сами убедиться, что полученное изображение ужовлетворяет виду тРНК (особенно если перевернуть на 180 градусов и отразить справа налево).
©Метелев Михаил