Учебный сайт Кузнецовой Марии
Работа в Blast
Был проведен поиск гомологов последовательности с идентификатором Q5QZI4 в банке Uniprot/SwissProt с помощью программы protein blast (blasp). Процесс поиска представлен на рис. 1, первый результат - исходный белок.
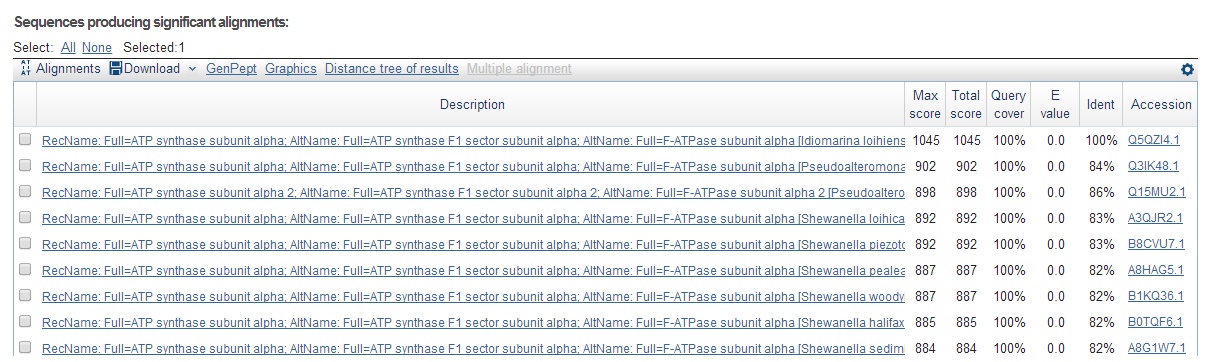 Рис. 1. Поиск белков с гомологичной последовательностью.
Рис. 1. Поиск белков с гомологичной последовательностью.
При этом результаты сравниваются по следующим параметрам:
Max Score - максимальный вес выравнивания одного фрагмента
Total Score - полный вес выравнивания всех фрагментов (совпадает с Max Score, если выравнивается 1 фрагмент)
Query Cover - процент покрытия данной последовательности и белка-гомолога
E-value - количество ожидаемых случайных находок с таким же или, может быть, лучшим счетом
Ident - процент совпадения (идентичности)
Accession - AC number базы Uniprot для данного белка
Строчка выбранного мной гомолога представлена на рис. 2.
![]() Рис. 2. Выбранный гомолог.
Рис. 2. Выбранный гомолог.
 Рис. 3. Выравнивание исходного белка и белка-гомолога.
Рис. 3. Выравнивание исходного белка и белка-гомолога.
Далее была построена карта локального сходства двух белков. Для этого использовался pBLAST, но с функцией Align two sequences, в частности, с помощью вклади Dot Matrix. Как видно из рис. 4 (карты) последовательности довольно хорошо выравниваются по первому фрагменту. Из параметров выравнивания (E-value, Score и т.д.) и карты локального сходства можно предположить, что эти два белка гомологичны. Это подтверждает тот факт, что это белки оба являются α-субъединицей AТФ-синтазы в разных бактериях. Второе выравнивание тоже неплохо совпадает, но, легко понять, что эти фрагменты негомологичны.
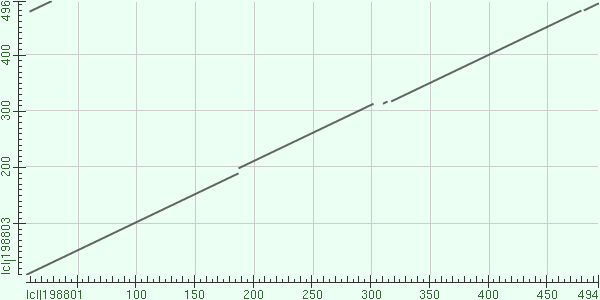 Рис. 4. Карта сходства выравниваний.
Рис. 4. Карта сходства выравниваний.
Далее были найдены гомологи своего белка среди эукариот. Для этого используя pBLAST, был указан в окне Organism - eurakyotes
(taxid:2759). Было найдено 442 эукариотных гомолога. Но все имели процент сходства 24-55%% и разные значения E-value. Были
выбраны 10 гомологов для построения множественного выравнивания. Их последовательности вы можете скачать здесь.
Выбранные гомологи представлены на рисунке 5. Само множественное выравнивание гомологичных белков представлено на рис. 6.
 Рис. 5. Выбранные, для построения выравнивания, гомологи.
Рис. 5. Выбранные, для построения выравнивания, гомологи.
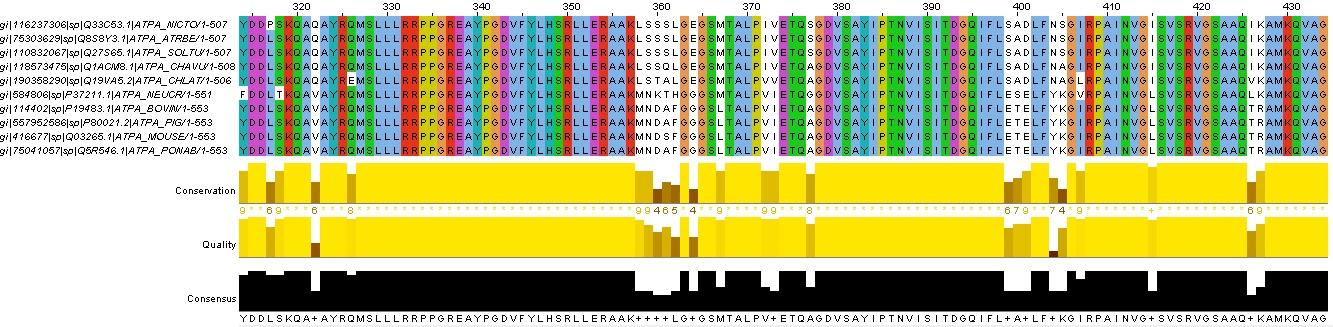 Рис. 6. Множественное выравнивание выбранных белков.
Рис. 6. Множественное выравнивание выбранных белков.
Для выравнивания использована раскраска ClustalX, процент идентичность >70%. Как видно из выравнивания, последовательности действительно можно назвать гомологичными, так как высок процент колонок, где полностью совпадают все последовательности. Скачать множественное выравнивание можно по ссылкам: jar, fasta.
Дата последнего обновления: 15.09.2014
Copyright © Кузнецова Мария, 2013.