Учебный сайт Кузнецовой Марии
ORF Finder

Рис. 1.Открытые рамки считывания, найденные программой ORF Finder.
Таблица 1.Рамки считывания, прошедшие фильтрацию по длине и перекрыванию.
| начало | конец | длина | цепь | функция |
| 449 | 1729 | 1281 | + | Мембранный белок |
| <1 | 446 | <420 | + | Протеин-дисульфид редуктаза |
Ниже приведены выравнивания, на основании которых было сделано заключение о функциях предполагаемых белков:
Идентификатор e-value Организм Длина белка Длина выравнивания Сходство
WP_008622200.1 0.0 Paraprevotella clara 430 427 83%
Query 1 MEKSLREKIIALVKSEVVPAVGCTEPIAVALCVAKATETLGCLPEKITALLSANILKNAM 60
+EK+ RE+II LVK +VVPA+GCTEPI V+LCVA+A E LG P ++ LSANILKNAM
Sbjct 3 IEKTERERIIRLVKQQVVPAMGCTEPICVSLCVARAAEVLGERPVAVSVFLSANILKNAM 62
Query 61 GVGIPGTGMIGLPIAIALGASVGRSEYELEVLKDVTPEAVEAGKQYIAEKRIDVQLKADA 120
GVGIPGTGM+GLPIAIALG G+S Y LEVLKDV P VE GKQYIAE+R+ + LK
Sbjct 63 GVGIPGTGMVGLPIAIALGVVAGKSAYGLEVLKDVMPTDVELGKQYIAERRVKISLKEGI 122
Query 121 PDKLYIEVHVEAK-GDEAVAVISGGHKNFVRIQKNDDVLLEKELASTDEDTDGDDWLTLE 179
+KLY+E VE++ G A AVI+G H +FV ++K+ VLL+ + +E + + L L
Sbjct 123 SEKLYVEAIVESESGHRATAVIAGQHTHFVYVEKDGKVLLDNRMPQGEEAGEDECELNLR 182
Query 180 KVYEFATTAPWDEIDFINEARRLNEEAAEKALHGNYGHSLGKALSRPLGRGIMGDTIFSH 239
KVY+F+ AP +EI+FI EARRLNE A+ +AL G YGH LGK LSRPLGRGIMGDTIFSH
Sbjct 183 KVYDFSVEAPLEEIEFIREARRLNESASRQALKGCYGHELGKTLSRPLGRGIMGDTIFSH 242
Query 240 ILSSTACACDARMAGAMIPVMSNSGSGNQGICATLPVVKYAEENHNTDEEMTRALMLSHL 299
ILSST+CACDARMAGAMIPVMSNSGSGNQGICAT+PVV +AEENHNTDEE+ RALMLSHL
Sbjct 243 ILSSTSCACDARMAGAMIPVMSNSGSGNQGICATMPVVVFAEENHNTDEELVRALMLSHL 302
Query 300 TAIYVKQSLGKLSALCGCVVASTGSSCGITYLMGGSYEQVTFAVKNMIANLTGMICDGAK 359
TAIY+KQSLG LSALCGCVVASTGSSCGITYLMGG YEQV++AVKNMIANLTGMICDGAK
Sbjct 303 TAIYIKQSLGVLSALCGCVVASTGSSCGITYLMGGKYEQVSYAVKNMIANLTGMICDGAK 362
Query 360 PSCALKLASGVSTAVLSAMLAIRNECVTSVEGIIDDSVDKSIHNLTKIGKDAMDETDRCV 419
PSCALKL SGVSTAVLSAMLA++ +CV+SVEGIIDD VDKSIHNLT IG +AM+ETDR V
Sbjct 363 PSCALKLTSGVSTAVLSAMLAMQQKCVSSVEGIIDDDVDKSIHNLTSIGAEAMNETDRKV 422
Query 420 LKIMTSK 426
L+IMT K
Sbjct 423 LEIMTHK 429
Идентификатор e-value Организм Длина белка Длина выравнивания Сходство
WP_021846361.1 7e-65 Bacteroides sp. CAG:598 481 138 84%
Query 2 FTDYEEGMKYAKEHNMPVMIDFTGYGCVNCRKMEAAVFVDQTVADIMTKDYVLIQLYVDE 61
F DY+ GMKYA+EH PVM+DFTGYGCVNCRKME AV+ D VAD++ DYVLI LYVD
Sbjct 343 FDDYDAGMKYAREHGKPVMLDFTGYGCVNCRKMELAVWTDMKVADLINNDYVLITLYVDN 402
Query 62 KTKLAEPIEVVDNGTPRKLRTVGDKWSYLQSSKFGATAQPFYVLLDNEGNPLAKSYSYDE 121
KT+L EP++V++NGT R LRTVGDKWSYLQ KFGA AQPFYVL+DNEG PL KSYSYDE
Sbjct 403 KTRLPEPVKVMENGTERTLRTVGDKWSYLQRVKFGANAQPFYVLIDNEGKPLNKSYSYDE 462
Query 122 DVDKYLVWLRSGLDAYKK 139
D+DKY+ +L++GLD YKK
Sbjct 463 DIDKYVDFLQTGLDNYKK 480
GeneMark
Ниже приведены предсказания, полученные с помощью GeneMark:
Predicted genes
Gene Strand LeftEnd RightEnd Gene Class
# Length
1 + <3 446 444 1
2 + 449 1729 1281 1
3 + 1841 2167 327 1
4 - 2344 2856 513 1
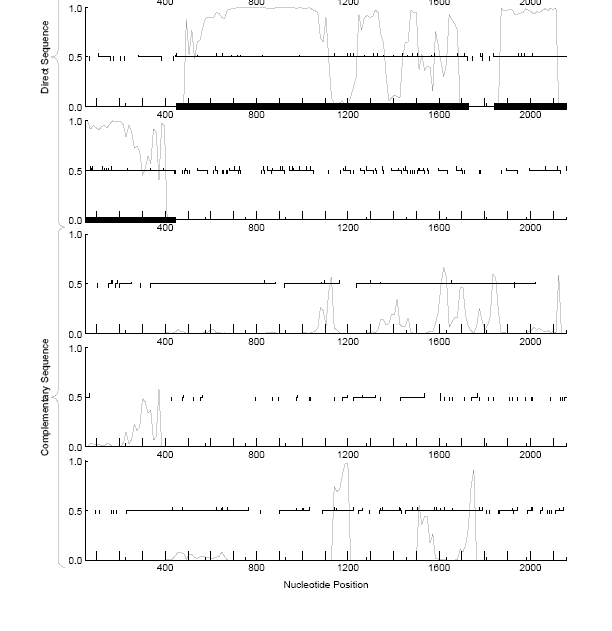
Рис. 2.График кодирующего потенциала.
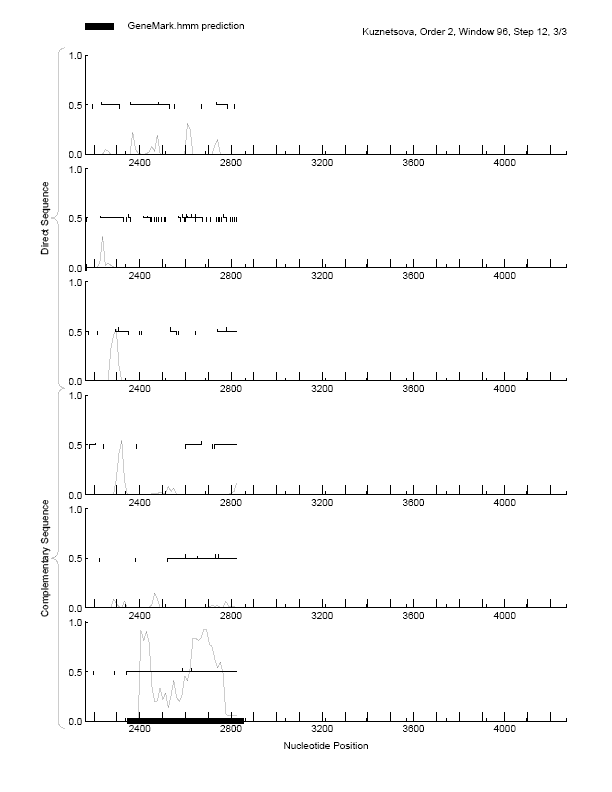
Рис. 3.График кодирующего потенциала.
И с помощью GeneMark, но с измененными эвристическими параметрами:
Predicted genes
Gene Strand LeftEnd RightEnd Gene Class
# Length
1 + <3 446 444 2
2 + 449 1729 1281 1
3 + 1841 2167 327 2
4 - 2344 2856 513 2
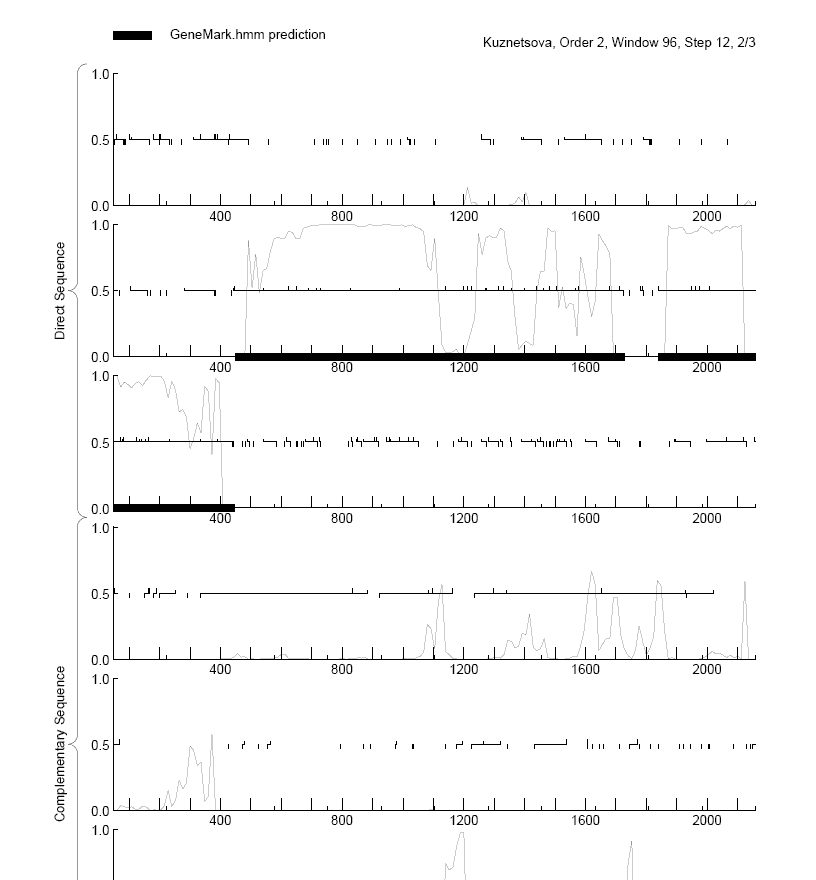
Рис. 4.График кодирующего потенциала.

Рис. 5.График кодирующего потенциала.
Красным в выдачах программ выделены рамки считывания, совпадающие с генами. В графиках кодирующего потенциала видно, что чем больше потенциал, тем больше вероятность того, что данная рамка считывания окажется геном.
Вывод: программа предсказания генов Genemark предсказывает гены с 50% вероятностью.
Изменения эвристических параметров приводят к некоторым изменениям в выдаче программы. Во-первых, изменяется класс предсказанного гена. Во-вторых, график кодирующего потенциала незначительно, но меняется - в некоторых местах линии становятся выше, в некоторых ниже. Хотя это, скорее всего, не имеет значения, так как границы рамок считывания не уточнились.