Учебный сайт Кузнецовой Марии
GENSCAN
В заданном фрагменте генома человека с помощью программы GeneScan были предсказаны следующие экзоны:
Таблица 1.Выдача GENSCAN.
| Начало | Конец | Цепь | Тип | |
| 1157 | 1593 | Внутренний | + | |
| 1699 | 1783 | Внутренний | + | |
| 8843 | 8930 | Внутренний | + | |
| 34135 | 34252 | Внутренний | + | |
| 34813 | 34963 | Внутренний | + | |
| 38185 | 38297 | Конечный | + | |
Genome browser
Нашла свою последовательность в геноме человека. Ее координаты: chr13:108,921,000-108,959,700. С помощью Genome browser было получено графическое изображение выравнивания данного фрагмента генома человека с известными мРНК и EST (на рисунках ниже).
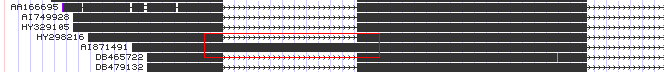
Рис. 1.Удержанный интрон.

Рис. 2.Кассетный экзон.
Чередующиеся экзоны, альтернативные донорные и акцепторные сайты сплайсинга в моей последовательности найдены не были.
BLASTX
Гипотетический белок
| Координаты по белку | Координаты по ДНК | Уточненные координаты по белку | Уточненные координаты по ДНК |
| 35 | 22870 | 28 | 22870 |
| 88 | 22706 | 81 | 22706 |
| 78 | 22664 | 71 | 22664 |
| 106 | 22572 | 99 | 22572 |
| 108 | 22455 | 100 | 22455 |
| 142 | 22351 | 136 | 22351 |
| 176 | 21845 | 169 | 21845 |
| 198 | 21777 | 191 | 21777 |
| 196 | 21696 | 189 | 21696 |
| 217 | 21631 | 215 | 21616 |
| 239 | 21478 | 225 | 21502 |
| 390 | 20936 | 383 | 20936 |
| 384 | 19876 | 365 | 19942 |
| 522 | 19460 | 511 | 19472 |
| 518 | 18590 | 511 | 18590 |
| 624 | 18282 | 618 | 18282 |
Range 1:
Query 22870 NFVVEALVKIKNALNDPRGALNNWDEDSVDPCSWAMITCSSDNLVTGLYVLPSSS 22706
N V+ALV IK ALNDP G LNNWDEDSVDPCSWAMITCS +NLV GL PS S
Sbjct 28 NHEVDALVSIKRALNDPHGVLNNWDEDSVDPCSWAMITCSPENLVIGLGA-PSQS 81
Range 2:
Query 22664 LIFGFYRGAPSQGLSGKLSGMIANLTNLRQV 22572
L+ G GAPSQ LSG LSG I NLTNLRQV
Sbjct 71 LVIGL--GAPSQSLSGTLSGSIGNLTNLRQV 99
Range 3:
Query 22458 LLQNNNISSEIPPEIGVLPNLHTLDLSNNQFSGHMPE 22348
LLQNNNIS IPP++G LP L TLDLSNN+ SG +P
Sbjct 100 LLQNNNISGGIPPQLGSLPKLQTLDLSNNRLSGVIPR 136
Range 4:
Query 21845 SFRDLSYNNLSGPVPKFPARTFK 21777
+F DLSYNNLSGPVPKFPARTF
Sbjct 169 AFLDLSYNNLSGPVPKFPARTFN 191
Range 5:
Query 21696 TCSIVGNSLICGSHSIEGCSGSAFPIP 21616
T ++ GN LICGS S CSGSA +P
Sbjct 189 TFNVAGNPLICGSSSTNVCSGSANSVP 215
Range 6
Query 21502 GKPSSKKLAIALGVSLSFVSLILIVVGFLFRRRNRTKKQTILDIT 21323
GK +SKKLAIALGVS SLIL+ G ++ R+ R K TIL+I
Sbjct 225 GKHNSKKLAIALGVSFGVASLILLAFGLIWYRK-RQKNLTILNIG 268
Query 21322 DLQEEGLMSLGNLRIFTFRELQVATDNFSSKNILGAGGFGNVYKGK 21143
D QEEGL+SLGNLR FTFRELQ AT+NFSSKNILGAGGFGNVYKGK
Sbjct 269 DKQEEGLISLGNLRNFTFRELQQATENFSSKNILGAGGFGNVYKGK 314
Query 21142 LGDSTMVAVKRLKDVIGTAGESQFRTELEMISLAVHRNLLRLTGYCATPNERLLVYPYMS 20963
LGD T++AVKRLKDV GT GESQFRTELEMISLAVHRNLLRL GYCATP ERLLVYPYMS
Sbjct 315 LGDGTVLAVKRLKDVAGTFGESQFRTELEMISLAVHRNLLRLIGYCATPTERLLVYPYMS 374
Query 20962 NGSVASRLR 20936
NGSVASRLR
Sbjct 375 NGSVASRLR 383
Range 7
Query 19942 EQCLWYGFLMNTSVVLIITEKPALDWNTRKRIAIGAARGLLYLHEQCDPK 19763
E+ L Y ++ N SV + EKPALDWNTRKRIAIGAARGLLYLHEQCDPK
Sbjct 365 ERLLVYPYMSNGSVASRLREKPALDWNTRKRIAIGAARGLLYLHEQCDPK 414
Query 19762 IIHRDVKAANILLDDFCEAVVGDFGLAKLLDHADSHVTTAVRGTVGHIAPEYLSTGQSSE 19583
IIHRDVKAAN+LLDDFCEA+VGDFGLAKLLDH+DSHVTTAVRGTVGHIAPEYLSTGQSSE
Sbjct 415 IIHRDVKAANVLLDDFCEAIVGDFGLAKLLDHSDSHVTTAVRGTVGHIAPEYLSTGQSSE 474
Query 19582 KTDVFGFGILLLELITGMRALEFGKTVNQKGAMLEWV 19472
KTDVFGFGILLLELITGMRALEFGK++NQKGAMLEWV
Sbjct 475 KTDVFGFGILLLELITGMRALEFGKSINQKGAMLEWV 511
Range 8
Query 18590 VRKVQQEKKVEQLVDRELGTNYDRIDVGEMLQVALLCTQYLPAHRPKMSEVVRMLEGDGL 18411
V+K+QQEKKVE LVDRELG+NYDRI+VGE+LQVALLCTQYLP HRPKMSEVVRMLEGDGL
Sbjct 511 VKKIQQEKKVEVLVDRELGSNYDRIEVGEILQVALLCTQYLPVHRPKMSEVVRMLEGDGL 570
Query 18410 AEKWAASHNHHLNPTTNFF----LPLKSNPSMVPRHD--DDDDRLSMFG 18282
AEKWAA+HN H NPT N F S P+ P+HD + +D+ SMFG
Sbjct 571 AEKWAAAHN-HTNPTMNNFHTNTKKSTSCPTSAPKHDHEEKNDQSSMFG 618
Белок YORE-YORE
| Координаты по белку | Координаты по ДНК | |||
| 16 | 93124 | |||
| 96 | 96363 | |||
| 93 | 96742 | |||
| 177 | 96981 | |||
| 173 | 98043 | |||
| 284 | 98270 | |||
| 249 | 99864 | |||
| 291 | 99992 | |||
| 289 | 101688 | |||
| 376 | 101951 | |||
| 376 | 102780 | |||
| 412 | 102890 | |||
| 431 | 103685 | |||
| 482 | 103894 | |||
| 482 | 104700 | |||
| 518 | 104810 | |||
| 519 | 105203 | |||
| 576 | 105376 | |||
| 576 | 106105 | |||
| 629 | 106266 |
Range 1:
Query 96124 VQYLIYGPFLAQVLYSRT-QEDSMKNNWCLHILIICFLRACMYQLWNSCSSMLFLNRNRR 96300
++YL+Y P AQV+YS +ED K WC+HILIIC L+A +++LW+ ++MLF+ R R
Sbjct 16 LKYLLYAPLAAQVVYSWVYEEDISKVLWCIHILIICGLKALVHELWSVFNNMLFVTRTLR 75
Query 96301 ILEQGVDFKQIDKEWDW*LYI 96363
I +G+DFKQID EW W YI
Sbjct 76 INPKGIDFKQIDHEWHWDNYI 96
Range 2:
Query 96742 DNFVILQALMASIACSIFP----FLSNLPLWDSRSLIVAIIFHMGVSEPIYYWAHKGFH- 96906
DN++ILQA++ S+ C + P +++LPLW+++ LI I+ H+ SEP+YY+ H+ FH
Sbjct 93 DNYIILQAIIVSLICYMSPPLMMMINSLPLWNTKGLIALIVLHVTFSEPLYYFLHRSFHR 152
Query 96907 GNYLFTHYHSFHHSSPVPQPFTGKD 96981
NY FTHYHSFHHSSPVP P T +
Sbjct 153 NNYFFTHYHSFHHSSPVPHPMTAGN 177
Range 3:
Query 98043 VAAGSVSFLEDIVLSGVIGIPLLGTCLIGCGSIYMIYGYVLMFDLMRCLGHCNVEIVPHQ 98222
+ AG+ + LE+I+L V G+PL+G CL G GS+ IYGY +MFD MRCLGHCNVEI H+
Sbjct 173 MTAGNATLLENIILCVVAGVPLIGCCLFGVGSLSAIYGYAVMFDFMRCLGHCNVEIFSHK 232
Query 98223 LFEKFPTLKYFLYTPT 98270
LFE P L+Y +YTPT
Sbjct 233 LFEILPVLRYLIYTPT 248
Range 4:
Query 99864 YHSLHHTEMDTNYCLFMPLYDALGNTLNSKSWELHKKNSSNSG 99992
YHSLHH EM TN+CLFMPL+D LG+T N SWEL KK ++G
Sbjct 249 YHSLHHQEMGTNFCLFMPLFDVLGDTQNPNSWELQKKIRLSAG 291
Range 5:
Query 101688 NAGKNWRVPDFVFLAHVVDLTSALHSPFVFRSFSSTPFMTKIFLIPFLPIVFMVMLVMWA 101867
+AG+ RVP+FVFLAH VD+ SA+H+PFVFRSF+S P+ T+IFL+P P F VML MWA
Sbjct 289 SAGERKRVPEFVFLAHGVDVMSAMHAPFVFRSFASMPYTTRIFLLPMWPFTFCVMLGMWA 348
Query 101868 KSKTFLFSFYGLRGRLHQTWVVPRFGFQ 101951
SKTFLFSFY LR L QTW VPRFGFQ
Sbjct 349 WSKTFLFSFYTLRNNLCQTWGVPRFGFQ 376
Range 6:
Query 102780 QYFLPFARDGINKQIEEAILRADRLGVKVLSLAALNK 102890
QYFLPFA GIN QIE AILRAD++GVKV+SLAALNK
Sbjct 376 QYFLPFATKGINDQIEAAILRADKIGVKVISLAALNK 412
Range 7:
Query 103685 NEALNGGGTLFVDKHPNLRVRVVHGNTLTAAVILNEIEEDVKEVFLTGATSKLGRAIALY 103864
NEALNGGGTLFV+KHP+LRVRVVHGNTLTAAVIL EI +DV EVFLTGATSKLGRAIALY
Sbjct 413 NEALNGGGTLFVNKHPDLRVRVVHGNTLTAAVILYEIPKDVNEVFLTGATSKLGRAIALY 472
Query 103865 LSTRRVKVLV 103894
L R V+VL+
Sbjct 473 LCRRGVRVLM 482
Range 8:
Query 104700 MLTISSERFQKIQKEVPTEFHKYLVQVTKYQAAKNCK 104810
MLT+S ERFQKIQKE P EF LVQVTKY AA++CK
Sbjct 482 MLTLSMERFQKIQKEAPVEFQNNLVQVTKYNAAQHCK 518
Range 9:
Query 105203 TWIVGKWITPREQSWAPPGAHFHQFVVPPILPFRRDCTYGDLAAMRLPDDVQGLGSCE 105376
TWIVGKW+TPREQSWAP G HFHQFVVPPIL FRR+CTYGDLAAM+LP DV+GLG+CE
Sbjct 519 TWIVGKWLTPREQSWAPAGTHFHQFVVPPILKFRRNCTYGDLAAMKLPKDVEGLGTCE 576
Range 10
Query 106105 QYTMERgvvhachaggvvhSLEGWNHHEVGAIDVERIDLVWRAALKHGLKPVST 106266
+YTMERGVVHACHAGGVVH LEGW HHEVGAIDV+RIDLVW AA+K+GL VS+
Sbjct 576 EYTMERGVVHACHAGGVVHMLEGWKHHEVGAIDVDRIDLVWEAAMKYGLSAVSS 629
Дата последнего обновления: 15.09.2014
Copyright © Кузнецова Мария, 2013.