Учебный сайт Кузнецовой Марии
Программа getorf пакета EMBOSS
Загрузила файл с записью D89965 из банка EMBL с помощью команды entret:
entret embl:d89965 -outfile d89965.entret
Получила файл d89965.entret. После этого получила набор трансляций всех открытых рамок считывания данной последовательности, отвечающих следующим требованиям:
- длиной не менее 30 аминокислотных остатков, то есть не менее 90 нуклеотидов (-minsize 90),
- считая открытой рамкой последовательность триплетов от старт-кодона до стоп-кодона (-find 1),
Для этого выполнила следующую команду:
getorf -minsize 90 -find 1 d89965.entret d89965.orf
Получила файл d89965.orf, содержащий 5 найденных открытых рамок считывания. Сравнив найденные рамки считывания с кодирующей последовательностью, приведённой в поле FT /translation записи D89965 из EMBL, видно, что третья найденная открытая рамка считывания (163 - 432) полностью соответствует кодирующей последовательности с координатами 163 - 435, несмотря на разницу в длине.
Данная запись EMBL ссылается на запись P0A7B8 в Swiss-Prot (/db_xref="UniProtKB/Swiss-Prot:...). Загрузить последовательность этой записи можно с помощью команды:
seqret sw:P0A7B8 P0A7B8.fasta
Получится файл P0A7B8.fasta. Чтобы выяснить, какой из полученных ранее открытых рамок считывания соответствует последовательность P0A7B8.fasta, можно воспользоваться программой blastp:
blastp -query P0A7B8.fasta -subject d89965.orf -evalue 0.01 -outfmt 6 -out P0A7B8_in_d89965_blastp.out
Получится файл P0A7B8_in_d89965_blastp.out. В нем с заданным порогом остается только одна находка: D89965_5 ([294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds). То есть можно сказать, что последовательность записи P0A7B8 соответствует пятой найденной открытой рамке считывания (294 - 1).
Последовательностью записи Swiss-Prot (P0A7B8), на которую ссылается данная запись EMBL (D89965):
>HSLV_ECOLI P0A7B8 ATP-dependent protease subunit HslV (3.4.25.2) (Heat shock protein HslV) MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGGTADAFTLFEL FERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVADETASLIITGNGDVVQPENDL IAIGSGGPYAQAAARALLENTELSAREIAEKALDIAGDICIYTNHFHTIEELSYKA
Пятая найденная в нуклеотидной последовательности D89965 открытая рамка считывания:
>D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Примечание. Запись D89965 из банка EMBL содержит последовательность мРНК крысы, а запись P0A7B8 из банка Swiss-Prot, на которую есть ссылка в записи D89965, содержит последовательность субъединицы HslV АТФ-зависимой протеазы кишечной палочки.
Вывод: последовательность P0A7B8 из банка Swiss-Prot имеет неправильную аннотацию из-за того, что вероятно эксперимент был поставлен неправильно. Исследователи искали белок с определенными свойствами в эпителии желудка крысы. После того, как они заметили активность белка со свойствами, похожими на свойства искомого, ученые выделили и секвенировали мРНК. Однако до этого, последовательность подобного белка уже была известна и хранилась в Swiss-Prot (принадлежала бактерии E.coli), поэтому найденная последовательность была проаннатирована автоматически. Тем самым получается, что белок, полученный из желудка крысы, на самом деле принадлежал E.coli, и, следовательно, авторы проаннотировали найденный белок неверно.
Файлы-списки
С помощью следующей последовательности команд программ пакета EMBOSS: Скачала в файл adh.fasta в fasta-формате все доступные в Swissprot последовательности алкогольдегидрогеназ: их идентификаторы описываются выражением adh*_*. Получила файл с универсальными адресами (USA) этих последовательностей: использовала программу infoseq с параметрами -only и -usa. Получила пример файл-список. Получила из этого файла-списка другой, меньший, с адресами только тех последовательностей, которые взяты из моих организмов. Использовала программу grep с параметром -f, чтобы подать ей на вход список слов для поиска. На основе нового файла-списка получила fasta-файл с последовательностями дегидрогеназ моих организмов. Использовала программу seqret. Список использованных команд ниже.
entret sw:adh*_* выбрала имя файла adh.fasta infoseq -only -usa adh.fasta > list grep -f organisms list > list2 seqret @list2 seq.fasta
Ссылка на полученный файл.
Случайная модель для оценки достоверности выравнивания
Выбрала две алкогольдегидрогеназы из предыдущего задания из далёких друг от друга организмов. Сделала 100 случайных перемешиваний первой аминокислотной последовательности с помощью команды shuffleseq. С помощью программы 'water' пакета EMBOSS сделала парные локальные выравнивания второй последовательности с исходной (неперемешанной) первой и с каждой из 100 перемешанных последовательностей. Получилось 101 парное выравнивание. Получила список весов этих выравниваний. Построила в Excel гистограмму распределения полученных весов (рис. 1).
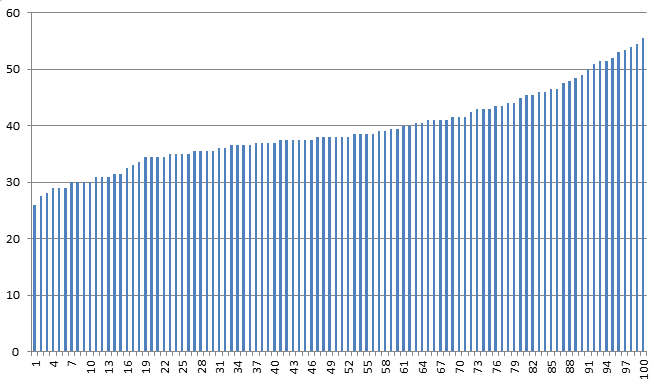
Рис. 1 Гистограмма весов выравнивания.
Видно, что большой вес имеют всего несколько случайных последовательностей, поэтому нельзя с уверенностью говорить о гомологии.
Дата последнего обновления: 15.09.2014
Copyright © Кузнецова Мария, 2013.