Учебный сайт Кузнецовой Марии
A- и B- формы ДНК | Структура РНК
С помощью команды fiber пакета 3DNA на сервере kodomo были построены A-, B- и Z-формы дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "GATC". Полученные структуры в формате .pdb A-, B- и Z-форм можно скачать здесь. здесь и здесь соответственно.
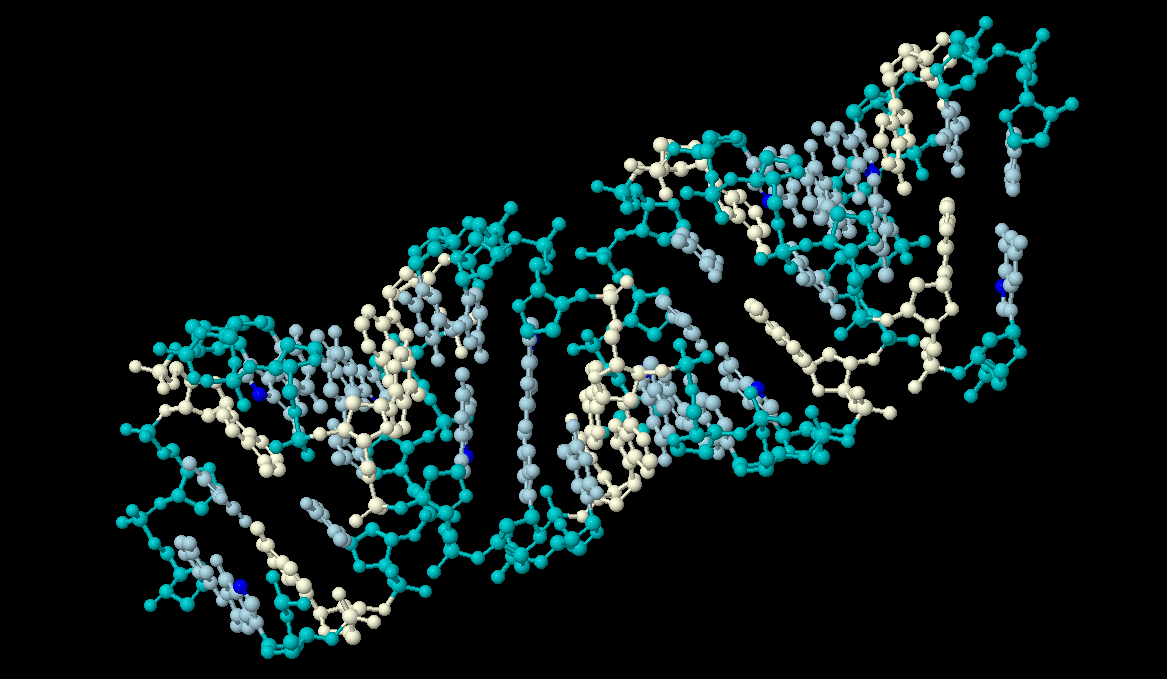
Работа была проведена со структурами ДНК и РНК, которые содержатся в файлах 1CF7.pdb и 1EVV.pdb соответственно. Структуры были визуально проверены на разрывы с помощью программы Jmol. На рис.2 представлены изображения данных цепей.
 Рис. 2. Изображения. полученные при помощи программы Jmol, отображающие изображения цепей ДНК (слева) и РНК (справа).
Рис. 2. Изображения. полученные при помощи программы Jmol, отображающие изображения цепей ДНК (слева) и РНК (справа).
Далее было определено, какие атомы тимина ориентированы в сторону большой/малой бороздки для B-формы дуплекса ДНК. Расположение тимина в данном дуплексе продемонстрировано на рис.3. "Ориентация" относительно бороздок продемонстрирована на рис.4.
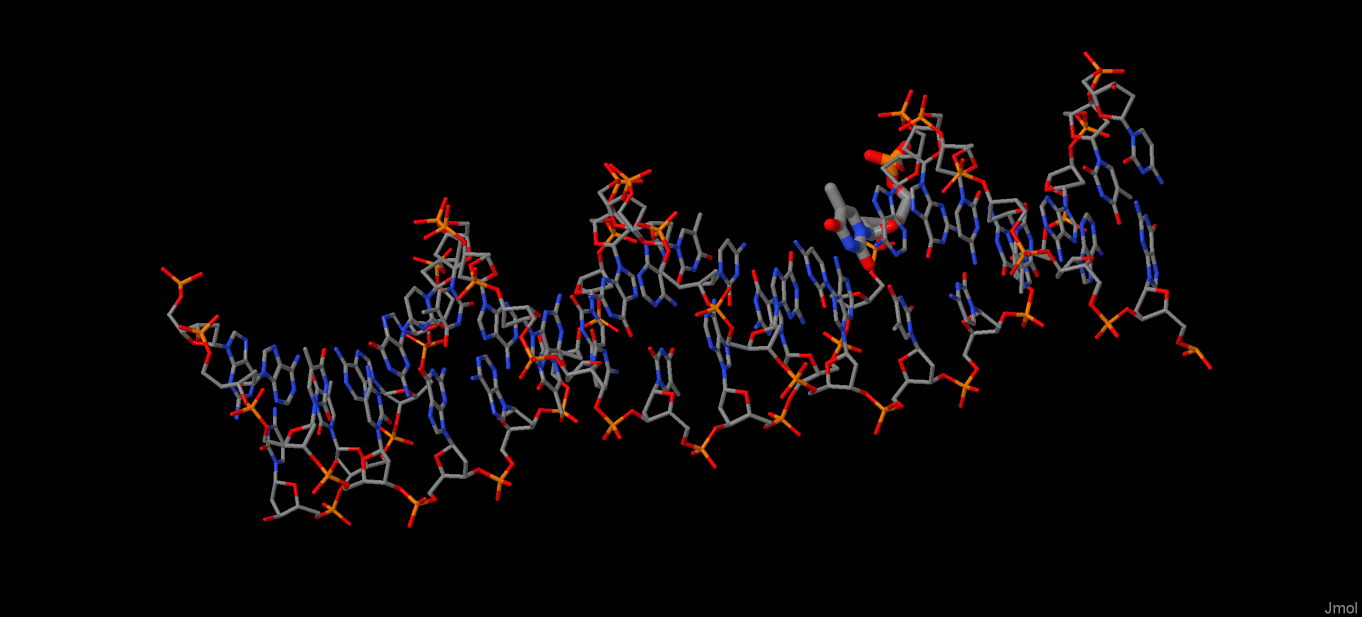 Рис. 3. Положение тимина в B-форме дуплекса ДНК.
Рис. 3. Положение тимина в B-форме дуплекса ДНК.
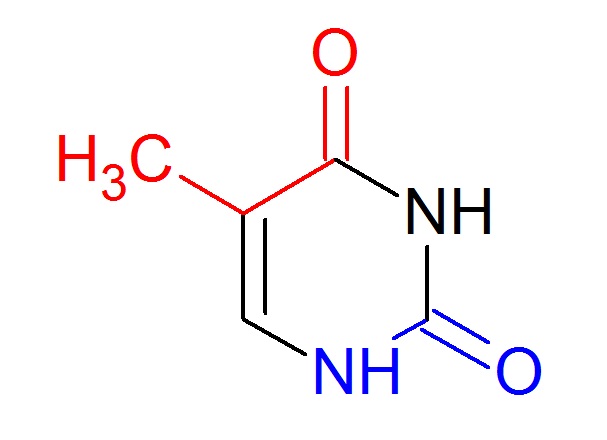
Атомы N1, O2, С2 направлены в сторону
малой бороздки и на рис. 4 обозначены синим цветом. Атомы N4, С4, С5, С6, С7 обращены в сторону большой бороздки и
обозначены на рис. 4 красным цветом. Оставшийся атом - N3 не имеет строгого направления.
Для А-формы дуплекса
ДНК направление атомов тимина такое же, как и в В-форме. В Z-форме тимина нет.
Рис. 4. Тимин. Красным выделены атомы, направленные в сторону большой бороздки В-формы дуплекса ДНК. Синим - в
сторону малой.
Таблица 1. Сравнение параметров различных форм ДНК.
| А-форма | В-форма | Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (A) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (A) | 18.5 (13-32) | 20.58 (3-33) | 23.5 (10-26) |
| Ширина малой бороздки (A) | 9.63 (3-31) | 13.2 (13-33) | 8.7 (15-31) |
Таблица 2. Торсионные углы тимина 7 А- и В-форм дуплекса ДНК.
| α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ζ (O3' - P) |
χ (C1' - N) |
|
| А-форма (из презентации) | 62 |
173 | 52 | 88 или 3 |
178 | -50 |
-160 |
| А-форма (gatc-a.pdb) | -51.7 |
174.8 |
41.7 |
79.1 |
-147.8 |
-75.1 |
22.7/85.2 |
| В-форма (из презентации) | 63 |
171 | 54 | 123 или 131 | 155 | -90 | -117 |
| В-форма (gatc-b.pdb) | -29.9 |
136.3 |
31.2 |
143.3 |
-140.8 |
-160.5 |
-34.4/145.6 |
Чтобы выполнить определить параметры структур нуклеиновых кислот, скаченные pdb-файлы были переведены в старый формат PDB (т. к. пакет 3DNA пока не может работать с новым форматом). Для этого были использованы комманды:
remediator --old ''1CF7.pdb'' > ''1GTR_old.pdb
remediator --old ''1EVV.pdb'' > ''1RH6_old.pdb
В результате были получены файлы 1EVV_old.pdb и 1CF7_old.pdb. Эти файлы были поданы на вход программе find_pair (определяет спаренные основания и положения спиралей в структуре), полученные данные были перенаправлены на вход программе analyze:
find_pair -t 1GTR.pdb stdout | analyze
find_pair -t 1RH6.pdb stdout | analyze
В результате было получено несколько файлов, в том числе наиболее нужные нам файлы 1CF7_old.out
и 1EVV_old.out
Файлы 1CF7_old.out и 1EVV_old.out, полученные ранее, использовались для получения информации о торсионных углах и водородных
связях.
Таблица 3. Торсионные углы ДНК из структуры 1CF7.
| # | base | α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ζ (O3' - P) |
χ (C1' - N) |
| 1 | T | --- | -173.2 | 49.3 | 131.7 | -166.6 | -92.8 | -130.9 |
| 2 | T | -60.3 | 175.5 | 55.3 | 130.0 | -178.7 | -101.1 | -113.4 |
| 3 | T | -66.5 | -175.4 | 55.3 | 140.6 | -161.1 | -117.5 | -109.5 |
| 4 | T | 39.3 | -155.7 | -76.6 | 156.8 | 176.9 | -66.9 | -116.3 |
| 5 | C | -85.8 | -171.2 | 48.6 | 132.6 | -99.1 | 160.8 | -100.5 |
| 6 | G | -79.6 | 144.0 | 56.1 | 132.8 | -178.5 | -75.4 | -114.1 |
| 7 | C | -41.0 | -162.3 | 7.4 | 136.4 | -163.5 | -107.8 | -103.0 |
| 8 | G | -55.6 | 165.8 | 45.8 | 147.3 | -116.5 | 162.2 | -88.1 |
| 9 | C | -62.7 | 143.2 | 45.4 | 135.9 | -172.1 | -50.4 | -116.7 |
| 10 | G | -151.5 | 101.8 | 162.4 | 143.7 | -149.3 | -114.9 | -129.2 |
| 11 | G | -54.7 | 160.9 | 46.6 | 134.6 | -161.2 | -123.9 | -121.4 |
| 12 | T | 37.7 | -164.9 | -66.3 | 158.1 | -170.7 | -111.5 | -115.1 |
| 13 | T | 38.3 | -176.1 | -62.7 | 156.4 | 175.7 | -112.7 | -100.6 |
| 14 | T | 48.0 | -177.9 | -63.2 | 155.5 | 174.6 | 105.5 | -99.9 |
| 15 | T | 52.3 | 179.9 | -69.9 | 159.5 | --- | --- | -103.7 |
| 1 | A | -135.8 | 137.2 | 136.6 | 136.1 | --- | --- | -142.3 |
| 2 | A | 173.8 | -151.7 | 162.5 | 127.8 | -161.3 | -79.5 | -133.3 |
| 3 | A | -25.8 | 139.9 | 44.8 | 132.5 | 167.1 | -109.7 | -113.8 |
| 4 | A | -70.7 | 137.9 | 48.5 | 143.5 | -161.3 | -164.0 | -107.4 |
| 5 | G | -67.3 | 167.2 | 50.0 | 147.0 | -111.3 | 177.9 | -85.5 |
| 6 | C | -61.8 | 145.0 | 43.8 | 138.4 | -158.1 | -98.1 | -104.8 |
| 7 | G | -50.5 | 162.1 | 38.6 | 146.0 | -117.5 | 167.0 | -85.2 |
| 8 | C | 73.4 | -170.4 | -110.6 | 139.3 | -143.2 | -103.9 | -113.7 |
| 9 | G | 112.6 | -141.6 | 166.0 | 133.7 | -153.9 | -91.6 | -139.1 |
| 10 | C | -89.3 | 178.4 | 56.7 | 137.0 | -89.8 | 125.3 | -106.8 |
| 11 | C | 62.5 | -160.4 | -95.0 | 144.9 | -166.0 | -73.0 | -126.7 |
| 12 | A | 43.1 | -172.7 | -66.0 | 151.8 | -176.2 | -100.4 | -108.3 |
| 13 | A | -66.2 | -169.8 | 59.7 | 136.5 | -172.9 | -118.5 | -104.6 |
| 14 | A | -53.8 | 179.9 | 44.9 | 134.5 | 167.1 | -89.5 | -108.9 |
| 15 | A | --- | -127.5 | 59.8 | 135.5 | -168.4 | -105.7 | -131.8 |
| # | среднее значение |
-19.8 | -5.7 | 26.8 | 141.3 | -88.5 | -50.9 | -110.2 |
С помощью Microsoft Excel были проанализированны торсионные углы ДНК структуры 1CF7. было получено среднее значение для каждого угла (см. Таблицу 3). а так же найден нуклеотид. суммарное отклонение каждого торсионного угла от среднего значения максимально. Этот нуклеотид - 14 тимин из первой цепи.
Таблица 4. Торсионные углы тРНК из структуры 1EVV.
| # | base | α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ζ (O3' - P) |
χ (C1' - N) |
| 1 | G | --- | -172.2 | 178.7 | 101.1 | -166.1 | -43.9 | 169.2 |
| 2 | C | -134.3 | -120.6 | 52.8 | 82.5 | -175.1 | -59.6 | -154.2 |
| 3 | G | -99.8 | 173.3 | 82.9 | 78.5 | -144.4 | -83.7 | -169.1 |
| 4 | G | -69.9 | 161.6 | 66.3 | 81.2 | -153.8 | -64.8 | -175.4 |
| 5 | A | -73.5 | -175 | 51 | 83.9 | -151 | -66.3 | -160.8 |
| 6 | U | -69 | 164.8 | 64.2 | 79.6 | -151.4 | -75.9 | -172 |
| 7 | U | -53.6 | -180 | 58 | 138.4 | --- | --- | -127.8 |
| 8 | C | --- | 172.6 | 40.7 | 84.7 | -152.7 | -77.3 | -172.3 |
| 9 | U | -48.3 | 177.4 | 37 | 85 | -149.6 | -79.8 | -165.1 |
| 10 | G | -41.5 | 174.5 | 30.8 | 87.2 | -154.6 | -73 | -154.9 |
| 11 | U | -62.9 | 167.1 | 50.5 | 81.4 | -160 | -66.5 | -167.7 |
| 12 | G | -59.6 | 173.1 | 53.8 | 83.4 | -167 | -60.4 | -173.3 |
| 13 | U | -85.6 | -167.6 | 58 | 80.1 | -136 | -68.8 | -156.1 |
| 14 | P | -53.7 | 170.2 | 44.2 | 79.7 | --- | --- | -153.2 |
| 15 | A | --- | 149 | 58.5 | 80.1 | --- | --- | -154.5 |
| 16 | A | --- | -174.6 | 60.3 | 75.3 | -146.6 | -62.2 | -173.4 |
| 17 | P | -82.9 | 179.9 | 69.4 | 84.4 | -151.5 | -69.3 | -172.9 |
| 18 | C | -90.8 | 168.7 | 74.2 | 81.3 | -141.7 | -96.3 | -176.3 |
| 19 | U | 41.1 | 162.3 | -47.4 | 99.2 | -152.5 | -68.5 | -155.8 |
| 20 | G | -57.3 | 162.1 | 58.1 | 83.3 | -156.7 | -80.8 | -170.1 |
| 21 | G | -53.1 | 168.4 | 51.4 | 83.8 | -151.2 | -77.8 | -160 |
| 22 | A | -71.8 | 156.4 | 70 | 84.2 | --- | --- | -158.3 |
| 23 | G | --- | 148.4 | 57 | 91.2 | -134.6 | -76.3 | 173.4 |
| 24 | C | -75.1 | 179 | 58.4 | 89.4 | -145.8 | -71.1 | -161.4 |
| 25 | U | -69.4 | 173 | 49.4 | 82.8 | -162 | -73.4 | -160.1 |
| 26 | C | 138 | -155.7 | -163.9 | 79.3 | -169.9 | -79.1 | -168.2 |
| 27 | A | 131.3 | 177.1 | -153.1 | 91.4 | -117.3 | -65.5 | -175.3 |
| 28 | G | -50 | 160.2 | 41.4 | 80.4 | --- | --- | -166.8 |
| 29 | G | --- | 176.6 | 59.6 | 157.4 | --- | --- | -79.7 |
| 1 | C | -91.7 | -160.7 | 61.3 | 81.3 | --- | --- | -160 |
| 2 | G | -78.2 | 170.1 | 61.7 | 81.8 | -166.5 | -54 | -164 |
| 3 | C | -70.1 | 168.4 | 54.8 | 77.5 | -156 | -69.4 | -159.6 |
| 4 | U | -59.2 | 171.4 | 47.2 | 82.2 | -154.8 | -78.8 | -162.4 |
| 5 | U | -60.4 | 173.4 | 46.4 | 82.3 | -155.3 | -64.4 | -161.8 |
| 6 | A | -62.7 | 171.9 | 48.5 | 89.2 | -158.3 | -77 | -156.3 |
| 7 | A | -65.7 | 175.3 | 58.8 | 80.3 | -148.2 | -74.4 | -166.2 |
| 8 | G | -41.4 | 157.8 | 48.1 | 79.9 | -154.7 | -74.5 | -172.5 |
| 9 | A | -56.7 | 176.1 | 38 | 77 | -146 | -75.8 | -160.7 |
| 10 | C | -81.5 | 175.4 | 62.8 | 82.4 | -150.5 | -75.6 | -157.3 |
| 11 | A | -73.1 | -173 | 51.5 | 83.1 | -157.4 | -67.8 | -161.9 |
| 12 | C | --- | 179.3 | 33.8 | 83.2 | -157.5 | -69.9 | -170 |
| 13 | A | --- | -150.7 | 69 | 155.7 | --- | --- | -85.3 |
| 14 | G | --- | 137.1 | -67.9 | 177.7 | --- | --- | -95.6 |
| 15 | U | -98.3 | -149.6 | 56.2 | 87.7 | --- | --- | -145.6 |
| 16 | C | -71.3 | -179.5 | 48.3 | 85.7 | -164.2 | -49.8 | -144.8 |
| 17 | A | -71.6 | -175.1 | 49.1 | 87.7 | -143.4 | -62.1 | -158.2 |
| 18 | G | -58.6 | -176.7 | 38.8 | 82 | -166.6 | -56.7 | -157.4 |
| 19 | A | -56.4 | 165.2 | 52.3 | 82.8 | -159.9 | -72.3 | -164.8 |
| 20 | C | -72 | 175 | 53.7 | 81.3 | -147.3 | -76.3 | -162.5 |
| 21 | C | -60.7 | 171.5 | 48.8 | 81.8 | -148 | -73.6 | -163.5 |
| 22 | G | -85.1 | -177.4 | 62.6 | 81.8 | -136.5 | -71.6 | -163.1 |
| 23 | C | -57.7 | 169 | 47.8 | 77.5 | -161.1 | -49.3 | -161.7 |
| 24 | G | -51.9 | 175.1 | 32.1 | 88.3 | -147.4 | -66.7 | -158.2 |
| 25 | A | -59.8 | 179.9 | 47.3 | 84.3 | -152 | -73.4 | -161.4 |
| 26 | G | --- | 161.3 | 178.2 | 87.3 | -148.6 | -78.5 | -176.7 |
| 27 | U | --- | -140.9 | 50.3 | 81.2 | --- | --- | -159.6 |
| 28 | C | --- | -175.3 | 153.2 | 138.4 | --- | --- | -143.1 |
| 29 | C | --- | 175.1 | 47.1 | 85.8 | --- | --- | -163.9 |
| # | среднее значение |
-56.5 | 75.9 | 45.3 | 88.4 | -152.4 | -70.6 | -153.2 |
С помощью Microsoft Excel были проанализированны торсионные углы тРНК структуры 1EVV, было получено среднее значение для каждого угла (см. Таблицу 4). а так же найден нуклеотид, суммарное отклонение каждого торсионного угла от среднего значения максимально. Этот нуклеотид - 28 цитозин из второй цепи.
Рассмотрим структуру тРНК, приведенную в общем виде на рис. 5.
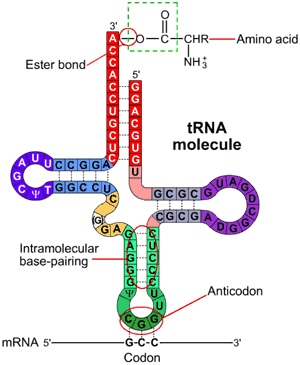
Акцепторный стебель(выделен красным, 7 нуклеотидов) формируется за счет спаривания 5'- и 3'-концевых нуклеотидов (на 3'-конце
неспаренные CCA для прикрепления аминокислоты).
D-петля (D-loop, выделена фиолетовым, 4 нуклеотида) содержит дигидроуридин.
Антикодоновая петля (выделена зеленым, 5 нуклеотидов) содержит антикодон.
T-петля (T-loop, выделена синим, 5 нуклеотидов) содержит ТψС последовательность, где ψ - псевдоуридин.
Рис. 5. Структура тРНК в общем виде.
Далее приведем результаты, полученные из 1EVV_old.out по структуре тРНК.
Strand I Strand II Helix
1 (0.003) A:...1_:[..G]G-----C[..C]:..72_:A (0.005) |
2 (0.004) A:...2_:[..C]C-----G[..G]:..71_:A (0.006) |
3 (0.008) A:...3_:[..G]G-----C[..C]:..70_:A (0.006) |
4 (0.009) A:...4_:[..G]G-*---U[..U]:..69_:A (0.007) |
5 (0.009) A:...5_:[..A]A-----U[..U]:..68_:A (0.008) |
6 (0.007) A:...6_:[..U]U-----A[..A]:..67_:A (0.016) |
7 (0.007) A:...7_:[..U]Ux----A[..A]:..66_:A (0.012) |
8 (0.017) A:..49_:[5MC]c-----G[..G]:..65_:A (0.017) |
9 (0.015) A:..50_:[..U]U-----A[..A]:..64_:A (0.015) |
10 (0.011) A:..51_:[..G]G-----C[..C]:..63_:A (0.009) |
11 (0.014) A:..52_:[..U]U-----A[..A]:..62_:A (0.022) |
12 (0.015) A:..53_:[..G]G----xC[..C]:..61_:A (0.006) |
13 (0.011) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.048) |
14 (0.018) A:..55_:[PSU]Px**+xG[..G]:..18_:A (0.025) x
15 (0.007) A:..36_:[..A]Ax*---U[..U]:..33_:A (0.008) |
16 (0.007) A:..38_:[..A]A-*---c[OMC]:..32_:A (0.008) |
17 (0.021) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.006) |
18 (0.010) A:..40_:[5MC]c-----G[..G]:..30_:A (0.009) |
19 (0.008) A:..41_:[..U]U-----A[..A]:..29_:A (0.006) |
20 (0.012) A:..42_:[..G]G-----C[..C]:..28_:A (0.005) |
21 (0.011) A:..43_:[..G]G-----C[..C]:..27_:A (0.006) |
22 (0.008) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.010) |
23 (0.017) A:..10_:[2MG]g-----C[..C]:..25_:A (0.006) |
24 (0.006) A:..11_:[..C]C-----G[..G]:..24_:A (0.017) |
25 (0.009) A:..12_:[..U]U-----A[..A]:..23_:A (0.013) |
26 (0.011) A:..13_:[..C]C----xG[..G]:..22_:A (0.011) |
27 (0.017) A:..14_:[..A]A-**-xU[..U]:...8_:A (0.008) |
28 (0.018) A:..15_:[..G]Gx**+xC[..C]:..48_:A (0.013) x
29 (0.018) A:..19_:[..G]G-----C[..C]:..56_:A (0.012) +
В соответствии с вышеприведенными данными:
акцепторный стебель состоит из участка 1 - 7 и комплементарного ему участка 66 - 72
Т-петля состоит из участка 53 - 59 и комплементарного ему участка 61 - 65
антикодоновый стебель состоит из участка 38 - 44 и комплементарного ему участка 26 - 32
D-стебель состоит из участка 10 - 13 и комплементарного ему участка 22 - 25
Неканонические пары составляют (отмечены в файле звёздочками) 4 G-U, 13 U-A, 14 P-G, 15 A-U, 16 A-C, 17 P-A, 22 A-G, 27 A-U,
28 G-C. Среди них есть как и некомплементарные пары, так и комплементарные, но с неканоническими водородными связями.
Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру составляют 13 U-A, 14 P-G, 15 A-U, 27 A-U,
28 G-C, 29 G-C (это комплементарные пары, не имеющие отношения к стеблям).
Изображение заданной структуры тРНК с окрашенными стеблями приведено на рис. 6.
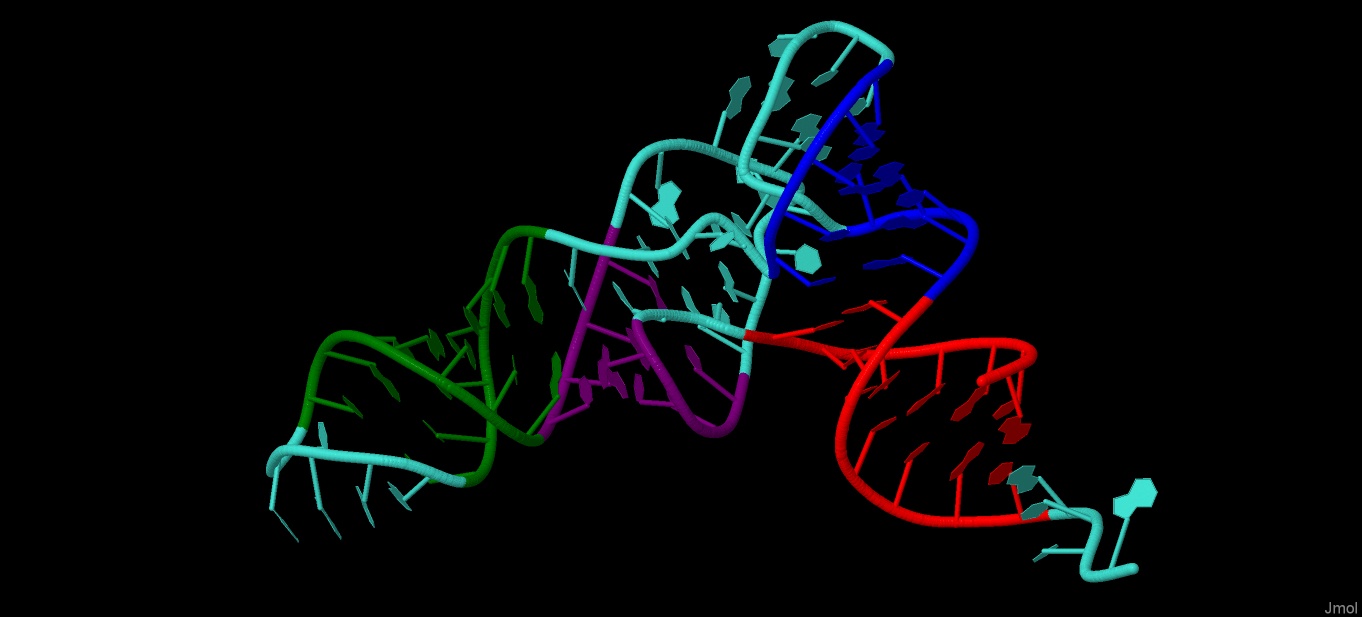 Рис. 6. тРНК из файла 1EVV с выделенными стеблями.
Рис. 6. тРНК из файла 1EVV с выделенными стеблями.
Чтобы определить пары нуклеотидов, для которых в наибольшей степени возможно стекинг-взаимодействие, была проанализирована информация о перекрывании нуклеотидов, полученная из файла 1EVV_old.out:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GC/GC 2.64( 0.51) 0.00( 0.00) 0.00( 0.00) 5.78( 2.74) 8.42( 3.25)
2 CG/CG 0.76( 0.01) 0.00( 0.00) 3.20( 0.72) 0.46( 0.00) 4.42( 0.73)
3 GG/UC 2.38( 0.91) 0.00( 0.00) 0.08( 0.00) 0.33( 0.00) 2.79( 0.91)
4 GA/UU 3.72( 2.19) 0.00( 0.00) 0.00( 0.00) 2.58( 0.89) 6.30( 3.08)
5 AU/AU 4.79( 3.43) 0.00( 0.00) 0.00( 0.00) 3.04( 1.74) 7.83( 5.18)
6 UU/AA 0.78( 0.01) 0.00( 0.00) 0.00( 0.00) 3.15( 2.66) 3.93( 2.66)
7 Uc/GA 0.82( 0.00) 0.00( 0.00) 0.00( 0.00) 3.37( 1.85) 4.19( 1.85)
8 cU/AG 0.00( 0.00) 0.00( 0.00) 0.43( 0.00) 3.87( 2.83) 4.29( 2.83)
9 UG/CA 0.16( 0.00) 0.00( 0.00) 2.60( 1.28) 0.13( 0.00) 2.89( 1.28)
10 GU/AC 6.87( 3.88) 0.00( 0.00) 0.00( 0.00) 3.53( 1.98) 10.40( 5.86)
11 UG/CA 0.00( 0.00) 0.00( 0.00) 4.46( 2.20) 0.00( 0.00) 4.46( 2.20)
12 Gu/aC 8.81( 4.20) 0.00( 0.00) 0.00( 0.00) 4.17( 0.85) 12.98( 5.06)
13 uP/Ga 5.72( 1.89) 0.00( 0.00) 0.00( 0.00) 8.48( 3.73) 14.20( 5.63)
14 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 AA/cU 0.65( 0.00) 0.00( 0.00) 0.00( 0.00) 2.75( 1.18) 3.40( 1.18)
16 AP/Ac 4.50( 1.95) 0.00( 0.00) 0.00( 0.00) 6.77( 3.74) 11.27( 5.68)
17 Pc/GA 1.75( 0.00) 0.00( 0.00) 0.00( 0.00) 5.13( 3.30) 6.88( 3.30)
18 cU/AG 0.07( 0.00) 0.00( 0.00) 0.71( 0.00) 0.72( 0.72) 1.49( 0.72)
19 UG/CA 0.07( 0.00) 0.00( 0.00) 3.07( 1.76) 0.04( 0.00) 3.18( 1.76)
20 GG/CC 2.97( 1.48) 0.00( 0.00) 0.36( 0.00) 0.19( 0.00) 3.53( 1.48)
21 GA/gC 3.66( 1.01) 0.00( 0.00) 0.00( 0.00) 5.47( 2.64) 9.13( 3.65)
22 Ag/Cg 0.00( 0.00) 0.00( 0.00) 1.27( 0.04) 0.34( 0.07) 1.61( 0.11)
23 gC/GC 3.38( 0.62) 0.00( 0.00) 0.00( 0.00) 7.14( 3.85) 10.52( 4.46)
24 CU/AG 1.05( 0.02) 0.00( 0.00) 0.03( 0.00) 2.47( 2.16) 3.55( 2.18)
25 UC/GA 1.26( 0.30) 0.00( 0.00) 0.00( 0.00) 1.00( 0.06) 2.26( 0.36)
26 CA/UG 0.00( 0.00) 1.98( 0.00) 2.24( 0.19) 0.00( 0.00) 4.22( 0.19)
27 AG/CU 2.43( 0.70) 0.00( 0.00) 0.16( 0.00) 0.00( 0.00) 2.58( 0.70)
28 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
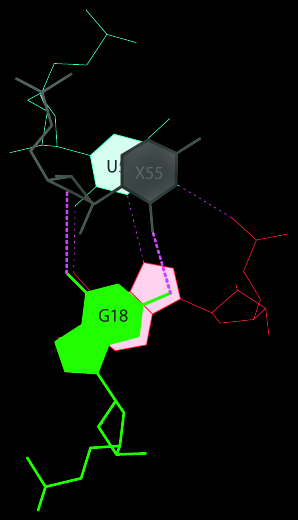
Очевидно, что наибольшее перекрывание наблюдается для пар 13 uP/Ga.
Из файла stacking.pdb с помощью комманды
ex_str -13 stacking.pdb st13.pdb
был вырезан файл со структурой нужных нам пар.
Затем с помощью команды
stack2img -cdolt st13.pdb st13.ps
было получено изображение стэкинг-взаимодействия пар 13 uP/Ga (приведено на рис. 7).
Рис. 7. Стэкинг-взаимодействие пар 13 uP/Ga.
Дата последнего обновления: 15.09.2014
Copyright © Кузнецова Мария. 2013.