Учебный сайт Кузнецовой Марии
KEGG
Задания данного практикума выполнялись для реакции 3.6.1.6 метаболизма пиримидинов (запись KEGG). Графическое изображение этого метаболического пути с выделенной реакцией можно увидеть на рис. 1.
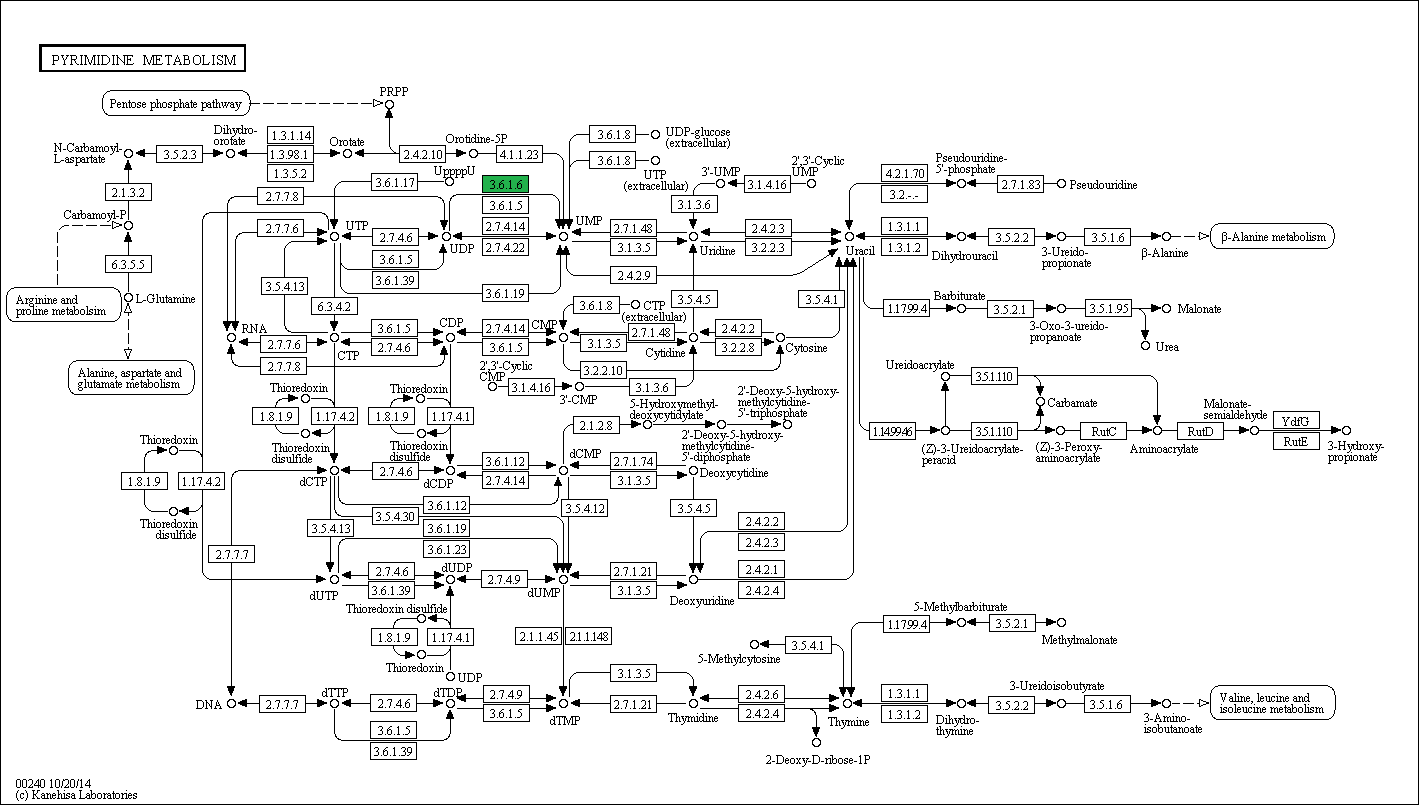 Рис. 1. Изображение метаболизма пиримидинов. Зелёным выделена реакция 3.6.1.6. Изображение получено с помощью базы данных KEGG.
Рис. 1. Изображение метаболизма пиримидинов. Зелёным выделена реакция 3.6.1.6. Изображение получено с помощью базы данных KEGG.
Реакцию 3.6.1.6 катализируют три ортологических ряда белков: ectonucleoside triphosphate diphosphohydrolase 5/6 (82 белка), soluble calcium-activated nucleotidase 1 (79 белков) и ectonucleoside triphosphate diphosphohydrolase 4 (30 белков).
Я скачала последовательности этих белков из Uniprot, переименовала их и построила множественное выравнивание с помощью Muscle. Проект выравнивания - здесь.
Затем я проанализировала это выравнивание в программе MEGA методом Neighbor-Joining с 100 бутстреп-репликами. Получилось дерево, которое можно увидеть на рис. 2.
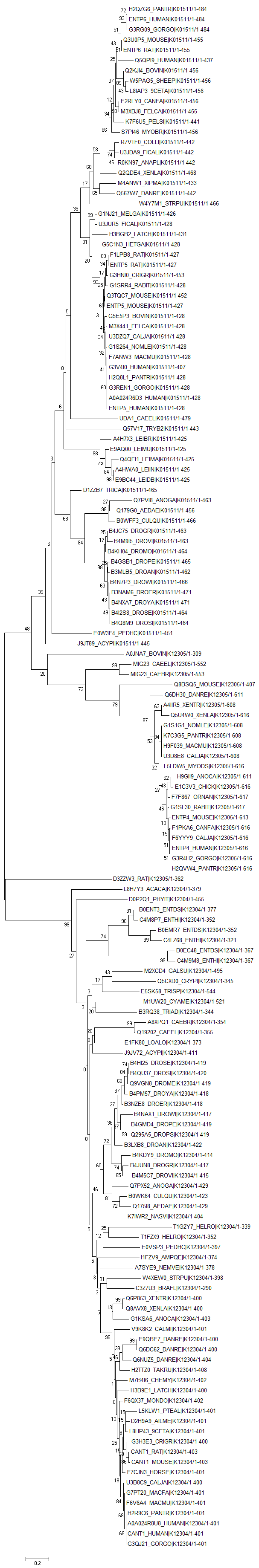 Рис. 2. Филогенетическое дерево белков, катализирующих реакцию 3.6.1.6. Получено с помощью программы MEGA (метод Neighbor-Joining с 100 бутстреп-репликами) на основе Muscle-выравнивания.
Рис. 2. Филогенетическое дерево белков, катализирующих реакцию 3.6.1.6. Получено с помощью программы MEGA (метод Neighbor-Joining с 100 бутстреп-репликами) на основе Muscle-выравнивания.
Три больших ветви дерева соответствуют трём ортологическим рядам (за исключением последовательности D3ZZW3_RAT, которая, видимо по ошибке попала в ветвь ряда K12304, но потом сразу отделилась от неё, если посмотреть, эта последовательность сильно выбивается из выравнивания и не гомологична даже своему ряду). Однако, в целом выравнивание последовательностей и дерево (в котором нет очень сильно выбивающихся последовательностей) позволяют предполагать гомологию этих трёх белков.
Дата последнего обновления: 15.09.2014
Copyright © Кузнецова Мария, 2013.