Учебный сайт Кузнецовой Марии
Поиск регуляторных мотивов
В рамках данного практикума я искала регуляторные мотивы в промоторных последовательностях E. coli (MEME07.txt) с помощью программы MEME. Для поиска использовались эксперементально установленные сайты посадки PurR. Результат задания в файле seq.docx. Программа meme была запущена дважды, в режимах One per sequence и Zero or one per sequence. В обоих случаях программа сработала довольно хорошо. При поиске с параметром One per sequence, точность предсказания оказалась 100%-ой для последовательностей, действительно содержащих сайты связывания. Напротив, в случае с параметром Zero or one per sequence был упущен один из реальных сайтов, а те, которые нашлись, были сдвинуты на один нуклеотид. Каждый раз следующие параметры оставались неизменны: Minimum length = 16; Maximum length = 16; Maximum number of motifs to find = 1. В итоговом файле экспериментально подтвержденные сайты выделены синим, сайты, полученные в режиме One per sequence - курсивом, Zero or one per sequence - полужирным. Все взятые на рассмотрение сайты помещены на серую подложку.
PePPER (задание выполнено совместно с Лозинским Ярославом)
PePPER - вебсервер для предсказания прокариотических промоторов и регулонов.
На этом сервере используется новый подход, который включает в себя все методы анализа данных для поиска промоторов и регулонов в любом бактериальном геноме. Он позволяет определить предположительные регулоны, составить полные аннотации межгенных регионов любых бактерий на основе уже существующих знаний по родственным организмам.
С помощью сервера PePPER можно искать прокариотические промоторы, мотивы в ДНК и сайты посадки транскрипционных факторов (этим трём возможностям соответствуют три режима работы сервера). В режиме поиска прокариотических промоторов нужно в одно поле ввести fasta-последовательность генома, а в другое - таблицу с названиями генов, координатами и указаниями цепи.
В режиме поиска мотивов в межгенных промежутках нужно выбрать активный геном и затем сгенерировать PFM (Position Frequency Matrix) для этого генома на основе выравнивания. Также может производиться поиск по собственноручно заданной PFM. Поиск мотивов происходит с использованием алгоритма MOODS.
В режиме поиска сайтов связывания транскрипционных факторов можно произвести поиск одного из уже имеющихся на сервере сайтов известных бактерий. Поиск можно производить либо в выбранном геноме (одном из уже имеющихся) либо самому ввести fasta-последовательность генома, в котором нужно найти сайт.
Мы провели работу с геномом бактерии Bifidobacterium adolescentis, для начала сменили активный геном. Это можно сделать, нажав по ссылке "Change active genome here". Перед нами открылся другой сервер, представленный на рисунке 1, куда мы ввели название нужной нам бактерии.
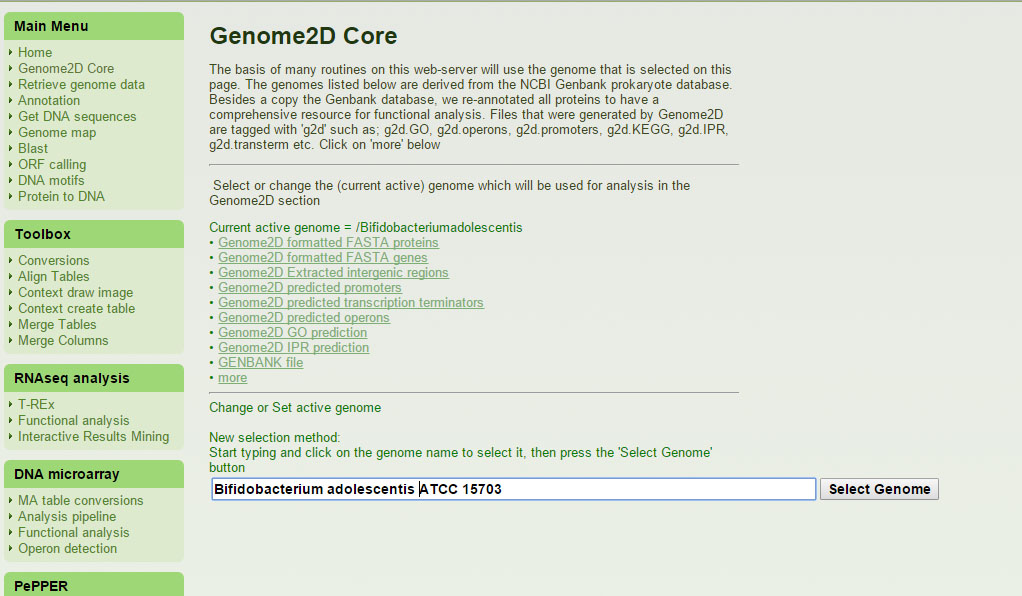
Рисунок 1. Смена активного генома.
Вернулись на исходную страницу. К сожалению, геномную последовательность нельзя загрузить целым файлом, её нужно скопировать и вставить в белое окошко. Далее выбирали интересующий
нас сайт связывания. Напротив него написано, на основе данных по какому организму будет осуществляться поиск. Общий вид показан на рисунке 2. Нажимаем на "Start RUN".
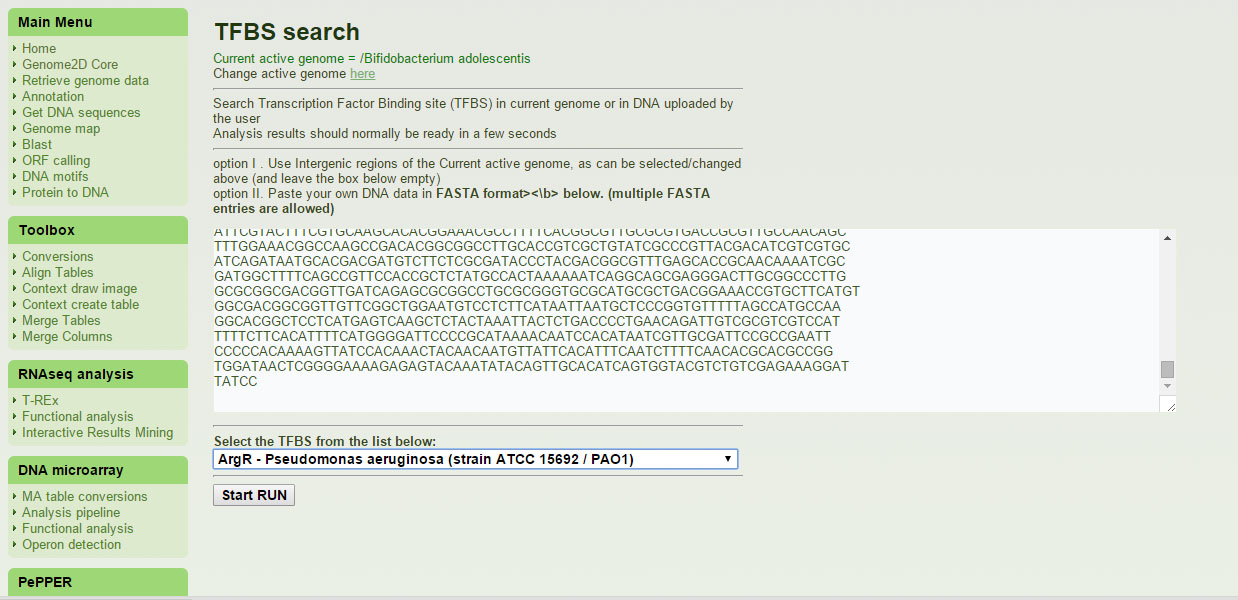
Рисунок 2. Поиск сайта связывания purR в геноме Bifidobacterium adolescentis.
Через несколько секунд сайт перезагрузился и мы увидили результат (рисунок 3). Нажав на "Download the results file here", мы получили информацио о координатах найденных сайтов в
txt-формате. Конкретно для нашего примера - output.txt. Программу можно запустить заново, нажав на "Start a new run".
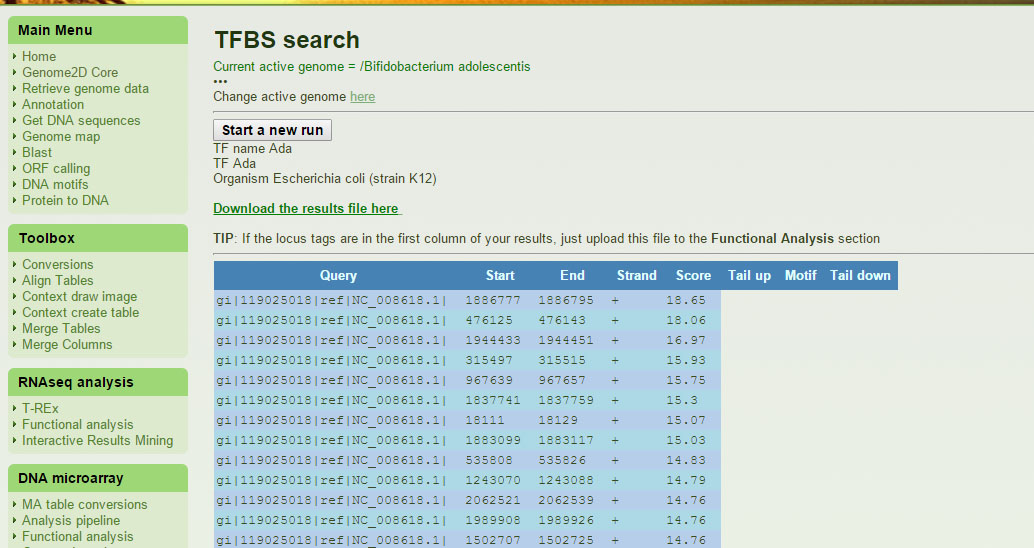
Рисунок 3. Результат поиск сайтов связывания purR в геноме Bifidobacterium adolescentis.
Дата последнего обновления: 15.09.2014
Copyright © Кузнецова Мария, 2013.