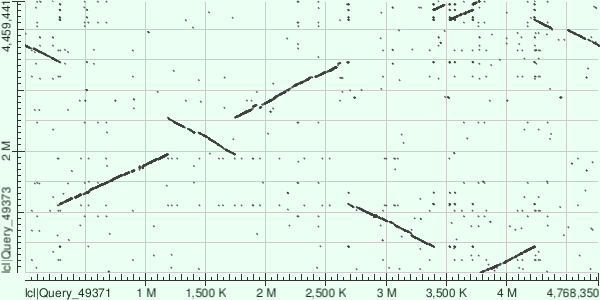
Определение функции и таксономии нуклеотидной последовательности
Для исследования последовательности решено было сравнить выдачи megablast и blastn со стандартными параметрами:
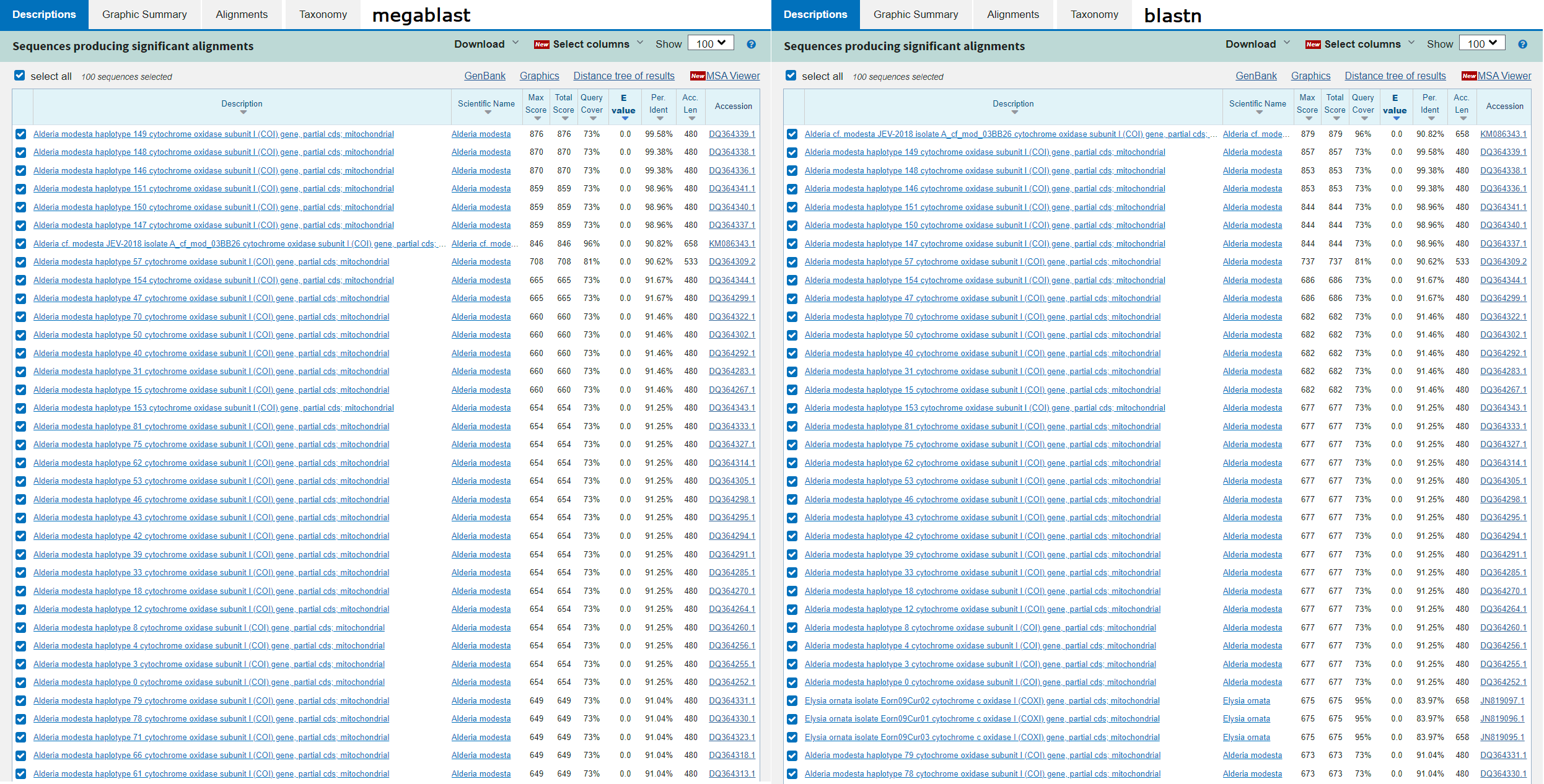
Насколько можно видеть, результаты являются близкими к идентичным, изменение параметров бласт в общем меняет выдачу в пределах погрешности, однако замечаем, что в числе лучших находок blastn помимо гена из Alderia modesta содержится также 3 находки из генома Elysia ornata с крайне высокими показателях покрытия при чуть более низкой идентичности.
Результаты выдачи крайне однородны и содержат множество находок с параметрами сходства более 90% при высоком покрытии и E-value равном машинному нулю, из чего делаем вывод, что исходная последовательность является кодирующей для первой субъединицы белка CO (cytochrome oxidase), принадлежащего гастроподе Alderia modesta