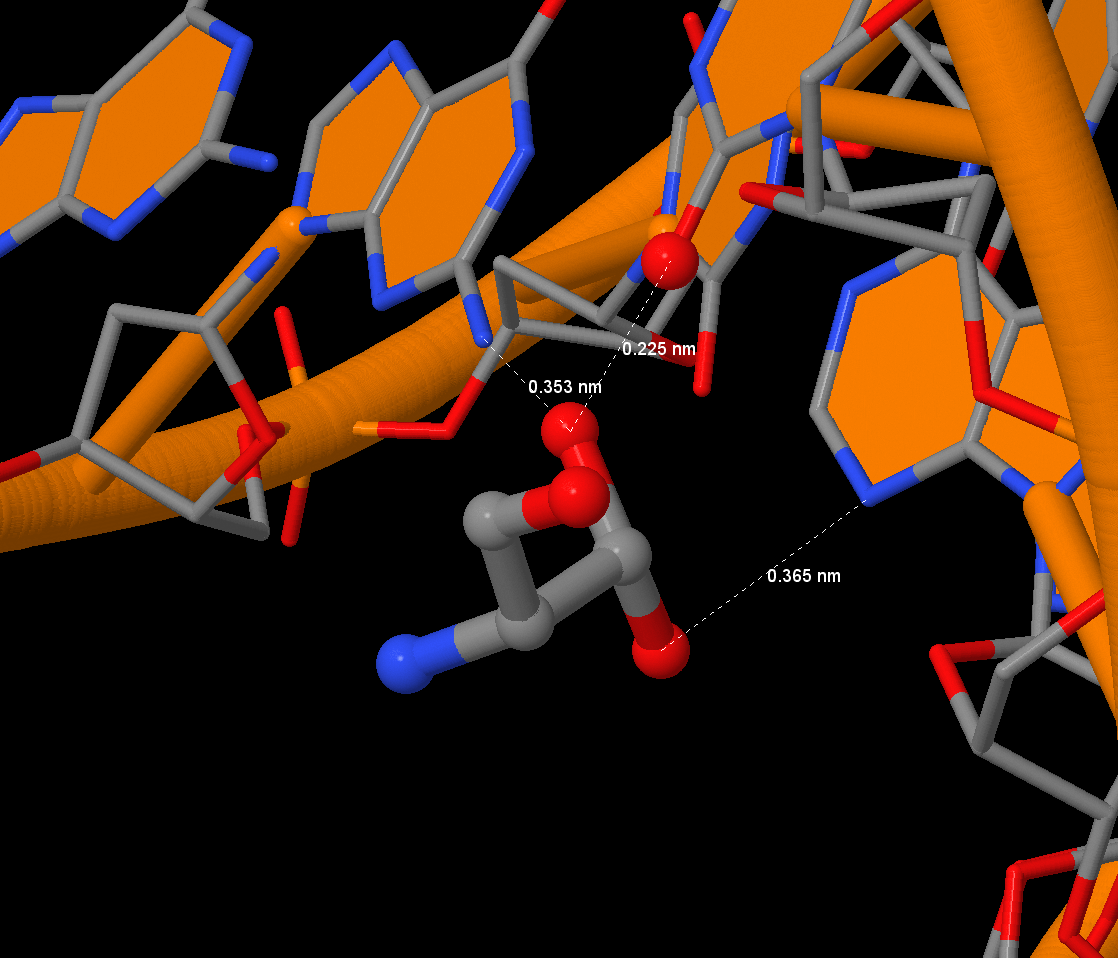
Упражнение 1
Скрипт, задающий множества
Скрипт, показывающий множества
Упражнение 2
Для подсчета связей использовались следующие команды:
Полярные с остатками 2'-дезоксирибозы: select within(3.5, (*.O? or *.N?) and protein) and set1
Неполярные с остатками 2'-дезоксирибозы: select within(4.5, (*.S? or *.C? or *.P?) and protein) and *.C?'
Полярные с остатками фосфорной кислоты: select within(3.5, (*.O? or *.N?) and protein) and set2
Неполярные с остатками фосфорной кислоты: select within(4.5, (*.S? or *.C? or *.P?) and protein) and *.P
| Контакты атомов белка с |
Полярные |
Неполярные |
Всего |
| остатками 2'-дезоксирибозы |
8 |
46 |
54 |
| остатками фосфорной кислоты |
15 |
14 |
29 |
Упражнение 3
Выдача nucplot
Упражнение 4
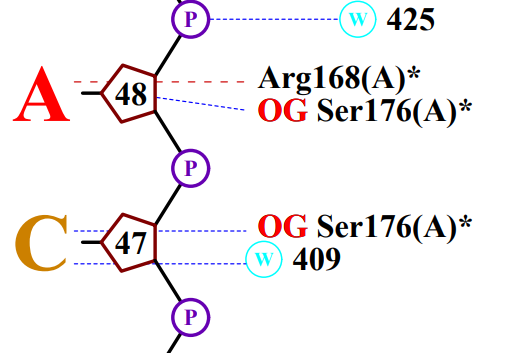
Многие анимокислотные остатки имеют 2 контакта с цепью ДНК, но не один не имеет трех, однако главным для определения цепи я выбрал Ser176(A)*, так как он контактирует в 2 местах с азотистыми основаниями, а не с сахарами остова.