Анализ трансмембранного белка
Из каталога OPM был выбран белок TOLC_ECOLI, являющийся эффлюксной помпой и фактором устойчивости к ампициллину для Escherichia coli. Именно этот белок был выбран из-за того что в процессе выполнения курсовой работы в лаборатории мне довелось работать со штаммом Escherichia coli_TOLC, который имел устойчивость к ампициллину именно благодоря этому белку.
Для работы были получены последовательность в Fasta формате и PDB файл 1ek9 с трехмерной структурой.
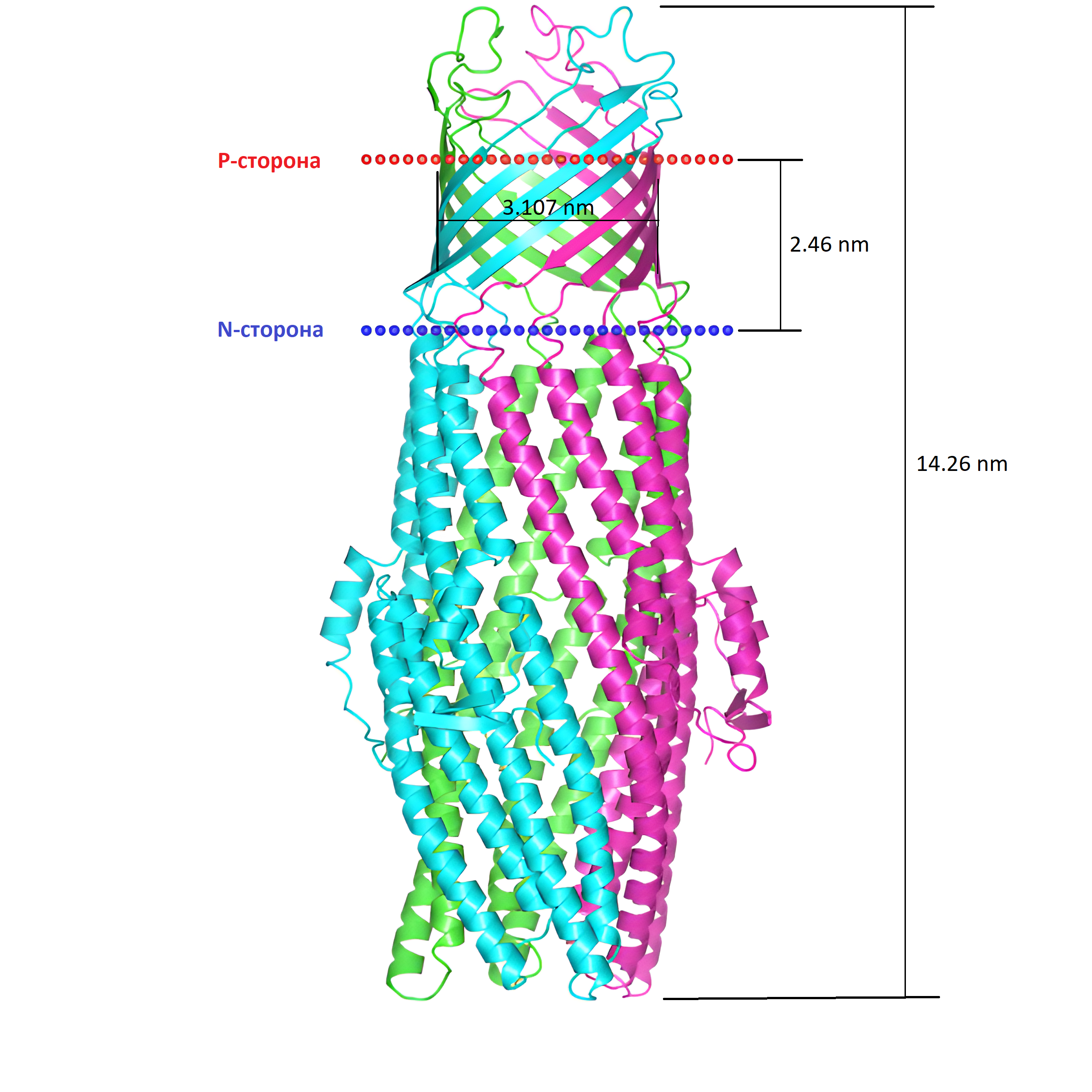
Толщина внутримембранной части белка: 2.46 nm (2.25 nm по моим измерениям в Jmol)
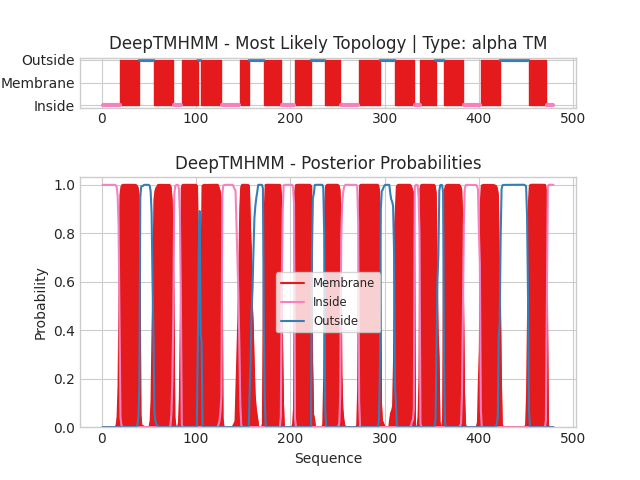
Трансмембранные участки: 1(40-50),2(65-77),3(246-256),4(282-290)
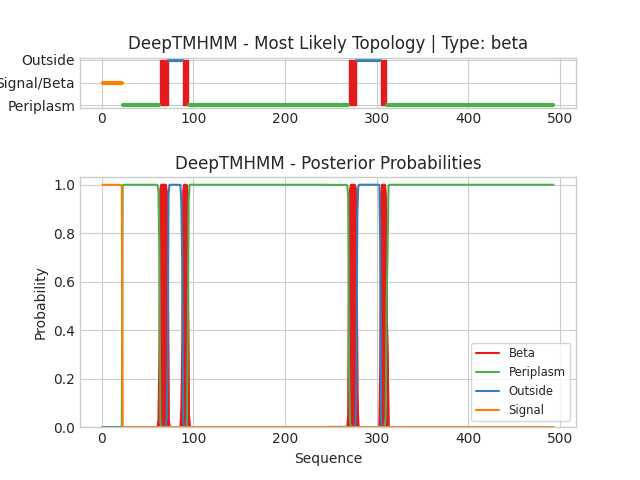
Трансмембранные участки(по DeepTHMHMM): ( 64 - 72 ) ( 89 - 94 ) ( 270 - 278 ) ( 305 - 310 )
Длины участков совпадают с небольшой погрешностью, однако можно заметить "смещение" на 24 аминокислоты - это след сигнального белка локализации, который читается и распознается DeepTHMHMM, однако не входит в OPM, так как отсутствует в конечной структуре белка.
Uniprot ID: P02930
Количество остатков в B-тяже: от 11 до 16
Из трехмерной структуры можем видеть, что белок имеет структуру, состоящую из 2 частей - внутриклеточной, собранной преимущественно из A-спиралей и внутримембранной, представляющей из себя B-бочонок 2.46 nm длины и 3.1 nm в поперечном сечении. Белок располагается на мембране, разграничивающей внешнюю среду и периплазму грамм-отрицательной Escherichia coli.