BLAST
1. Терминал linux.
В ОС Windows запустили Putty и зашли на сервер kodomo, используя свой логин и пароль.
Командой pwd убедились, что находимся в домашней директории: /home/students/y11/morozova_ea
2. Создаем директорию Pr1.
Командой mkdir в домашней директории создаем директорию Term2 (mkdir ~/Term2), в ней - директорию Block1 (mkdir ~/Term2/Block1), в ней - директорию Practices (mkdir ~/Term2/Block1/Practices), в ней - директорию Pr1 (mkdir ~/Term2/Block1/Practices/Pr1). Командой (ls ~/путь к директории) можно проверить ее содержимое и убедиться, что папки появились.
Для выполнения заданий перейдем в директорию Pr1 (cd ~/Term2/Block1/Practices/Pr1).
3. Получим информацию о своем белке.
Команда infoseq sw:guad_bacsu выдает на терминал информацию о записи GLOX_BACSU банка SwissProt.
Запишем информацию об этом белке в файл с именем GLOX_BACSU.info (infoseq sw:glox_bacsu 1> GLOX_BACSU.info) GLOX_BACSU.info
(Командой infoseq sw:GLOX_BACSU -html 1>html.info можно представить эту информацию ввиде таблицы)
| USA | Database | Name | Accession | Type | Length | Organism | Description |
|---|---|---|---|---|---|---|---|
| sw-id:GLOX_BACSU | sw | GLOX_BACSU | O31616 | P | 369 | Bacillus subtilis | Glycine oxidase (GO) (1.4.3.19) |
4. Запишем информацию о программе infoseq в файл.
Для этогго можно использовать разные опции:
infoseq –help 2> infoseq.info (infoseq.info)
man infoseq >infoseq1.info (infoseq1.info)
5. Используя "*" в имени последовательности найдите такие же белки в других видах рода Bacillus (infoseq sw:guad_bac* 1> other1.info). Таких не оказалось.
6. Повторите команду из предыдущего задания, используя разные опции изменения выдачи результата.
7. The description of command infoseq and it's qualifiers.
| Qualifier | Function | Example |
| -outfile | If you enter the name of a file here then this program will write the sequence details into that file. | infoseq sw:GUAD_BACSU -outfile outfile.infooutfile.info |
| -html | Format output as an HTML table | infoseq sw:GUAD_BACSU -html 1> html.infohtml.info |
| -[no]columns | Set this option on to print the sequence information into neat, aligned columns in the output file. Alternatively, leave it unset (N), in which case the information records will be delimited by a character, which you may specify by using the -delimiter option. In other words, if -columns is set on, the -delimiter option is overriden. | infoseq sw:GUAD_BACSU -columns 1> columns.infocolumns.info infoseq sw:GUAD_BACSU -nocolumns 1> nocolumns.infonocolumns.info |
| -delimiter | This string, which is usually a single haracter only, is used to delimit individual records in the text output file. It could be a space character, a tab character, a pipe character or any other character or string. | infoseq sw:GUAD_BACSU -nocolumns -delimiter "))" 1> delimiter.infodelimiter.info |
| -only | This is a way of shortening the command line if you only want a few things to be displayed. | infoseq sw:GUAD_BACSU -only -length 1> onlylength.info/onlylength.info |
| -[no]heading | Display column headings | infoseq sw:GUAD_BACSU -heading 1> heading.infoheading.info(по умолчанию) infoseq sw:GUAD_BACSU -noheading 1> noheading.infonoheading.info |
| -usa | Display the USA of the sequence. Only with -only | infoseq sw:GUAD_BACSU -only -usa 1> usa.infousa.info |
| -database | Display 'database' column. Only with -only | infoseq sw:GUAD_BACSU -nodatabase 1> nodatabase.info(по умолчанию эта колонка есть) gi.info |
| -name | Display 'name' column. Only with -only | infoseq sw:GUAD_BACSU -only -name 1> name.infogi.info |
| -accession | Display 'accession' column. Only with -only | infoseq sw:GUAD_BACSU -only -accession 1> accession.infogi.info |
| -type | Display 'type' column. Only with -only | infoseq sw:GUAD_BACSU -only -type 1> type.infogi.info |
| -length | Display 'length' column. Only with -only | infoseq sw:GUAD_BACSU -only -length 1> onlylength.infogi.info |
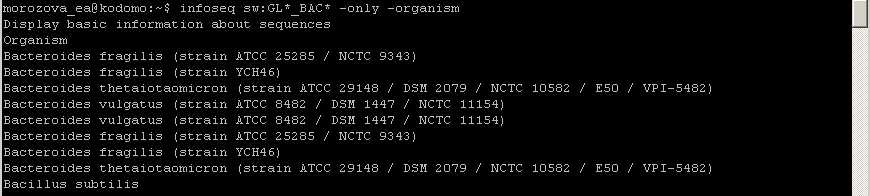
| -organism | Display 'organism' column. Only with -only | infoseq sw:GUAD_BACSU -only -organism 1> organism.infogi.info |
| -description | Display 'description' column. Only with -only | infoseq sw:GUAD_BACSU -only -description 1> description.infogi.info |
| -gi | Display 'GI' column | infoseq sw:GUAD_BACSU -gi 1> gi.info(по умолчанию не выводится) gi.info |
| -seqversion | Display 'version' column | infoseq sw:GUAD_BACSU -seqversion 1> seqversion.infogi.info |
| -pgc | Display 'percent GC content'column. Only with -only | infoseq sw:GUAD_BACSU -pgc 1> pgc.infogi.info |
| -help | Report command line options and exit. More information on associated and general qualifiers can be found with -help -verbose | infoseq sw:GUAD_BACSU -help 2> help.infohelp.info |