Предсказание генов прокариот.
1. Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для плазмиды.
В задании рассматривается плазмида Rhodovulum sulfidophilum (штамм SNK001). Rhodovulum sulfidophilum — это фотосинтезирующая несерная пурпурная бактерия, найденная в морских прибрежных зонах Вишакхапа́тнама (города на юго-восточном побережье Индии). Эта бактерия может жить как в аэробных условиях в темноте, так и в анаэробных условиях на свету. Эта бактерия выпускает внутриклеточные макромолекулы, такие как ДНК и РНК, во внеклеточную среду, что приводит к формированию клеточных агрегатов, а сам процесс известен как флокуляция.
С помощью нижеследующих команд пакета EMBOSS была получена последовательность в форматах cp015422.gff и cp015422.fasta.
seqret embl:CP015422 -feature cp015422.gff
seqret embl:CP015422 cp015422.fastaВ итоговом файле .gff были сохраненны особенности, которые нужны для дальнейшей работы. Для извлечения информации о начале, конце, ориентации генов из файла .gff, использовали команду:
grep 'CDS' cp015422.gff | awk '{print $4,$5,$7}' >> genbank.txt
Был получен файл . Команда grep (акроним от search globally for lines matching the regular expression, and print them) находит в файле строки с паттерном CDS, затем awk (Это утилита для извлечения данных; awk читает за один раз одну строку, выполняет определенные действия в зависимости от заданных опций, и выводит результат. Одним из самых простых и популярных способов использования awk является выбор столбца из текстового файла или из вывода другой команды) находит 4-й, 5-й и 7-й столбцы gff-файла, где содержатся соответствующие координаты, и записывает в файл .
Далее, для предсказания генов в данной плазмиде, воспользовались алгоритмом Prodigal (Prokaryotic Dynamic Programming Genefinding Algorithm).
В командной строке задаем:
prodigal.windows.exe -i cp015422.fasta -f sco -o prodigal -s prodical_genes
Длина плазмиды в соответствии с полученными данными равна 113 522 п.о., что вполне приемлимо для полноценной работы Prodigal.
prodigal.fasta| sed "s/_/ /g" | awk '{print $2,$3,$4}'>> prodigal-1.out
Команда grep ищет строки, где содержится паттерн ">". Затем в найденных строках команда sed заменяет "_" на пробел: s (substitute) заменяет первое встреченное соответствие первому шаблону, на второй шаблон; g (global) гарантирует выполнение команды всеми найденными соответствиями внутри каждой из строк.
Полученные файлы для дальнейшей работы: genbank.out и prodigal-1.out.
Далее, с помощью скрипта, были оценены следующие показатели:
Комментарии к скрипту:
1) Скрипт запрашивает на вход три файла (любых) (в нашем случае мы даем на вход файлы GenBank и Prodigal с координатами и файл с результатами) и создает два списка genbank (+ по 2 списка с координатами С- и N-концов) и prodigal( с аналогичным набором списков), содержащих строки с координатами из исходных файлов.
2) После этого для каждой координаты N-конца в n_genbank ищется такая же координата в n_prodigal. Если она найдена, проверяется совпадает ли координата C-конца и в зависимости от ответа подсчитывается число совпадающих генов (если координаты совпадают) или генов с разными C-концами (у белка). Далее для каждой координаты С-конца в c_genbank ищется такая же в c_prodigal, так что соответствующие координаты N-концов не совпадают. Таким образом подсчитывается число генов с по-разному предсказанными N-концами белков.
3) Все результаты записываются в указанный файл - results.out.
Результаты предсказания Prodigal Примеры, когда аннотация на одном из концов гена не совпала.
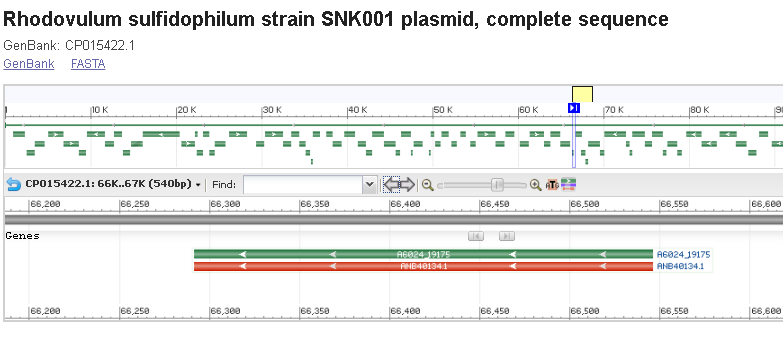
1) Ген предсказанный в Genbank, не найденный Prodigal: координаты 66 546-66 292 (-). Аннотация к данному гену в Genbank "Derived by automated computational analysis using gene prediction method: GeneMarkS+". Исходя из вышесказанного, сложно сказать является ли анатированный ген в gb правомерным в полной мере.
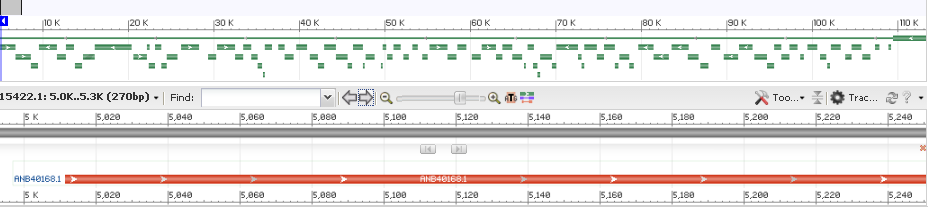
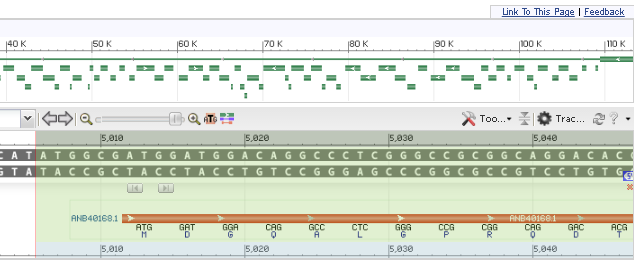
2) Пример расхождения предсказаний. Рассмотрим ген с координатами 5012-6868, аннотированный в Genbank.
N-конец этого гена в Prodigal аннотирован с 5006 позиции, где также наблюдается кодон ATG.