Предсказание генов эукариот..
1. Описание контига и одного из его генов, для которого предсказан альтернативный сплайсинг.
В задании представлен скэффолд NW_003303700.1, принадлежащий организму Phytophthora infestans T30-4.
Фитофтора — род грибоподобных протистов из отдела Oomycota. Вызывают фитофтороз — заболевание растений, в основном семейства паслёновых (Solanaceae). Название вида Фитофтора инфестанс происходит от греческого «истребитель растений».
Домен
Eukaryota
Тип
Heterokonta
Класс
Oomycota
Порядок
Peronosporales
Семейство
Pythiaceae
Род
Phytophthora
Вид
P. infestans
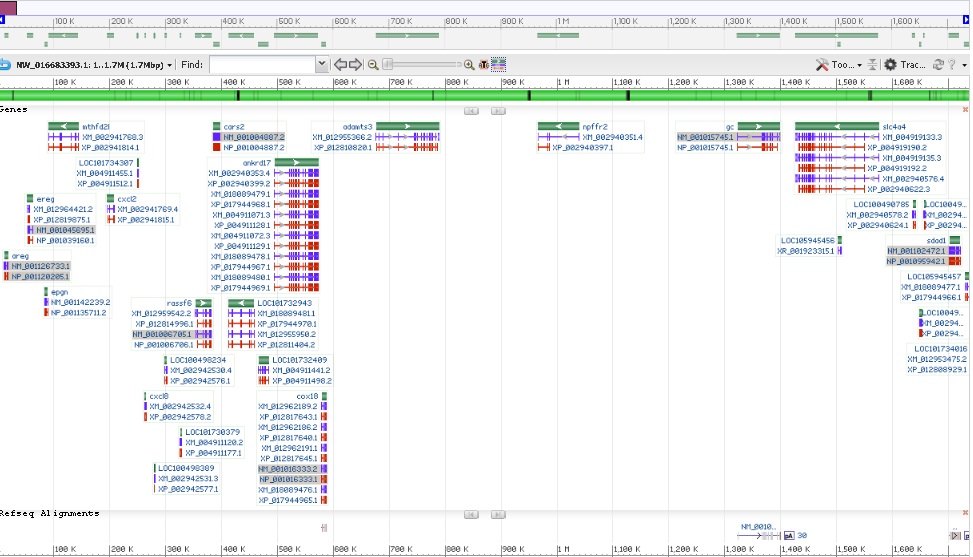
Так как, в данном мне образце не было найдено участков альтернативного сплайсинга для работы был взят другой скэффолд (из резерва) NW_016683393.1, принадлежащий организму Xenopus tropicalis.
Характеристики скэффолда:
Длина:118 935 п.о.
Число генов и ген-кодирующих последовательностей: 27 и 26 соответственно.
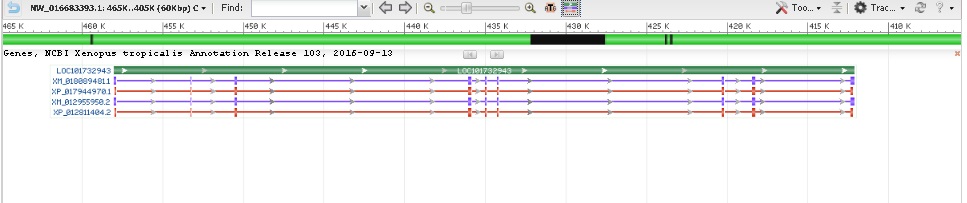
Рассмотрим ген LOC101732943, для которого известен альтернативный сплайсинг.
Расположение: 412102..458006
Количество экзонов: 9
С этого гена транскрибируется 2 различных формы мРНК: XM_018089481.1 (XP_017944970.1 ) и XM_012955950.2 (XP_012811404.2 )
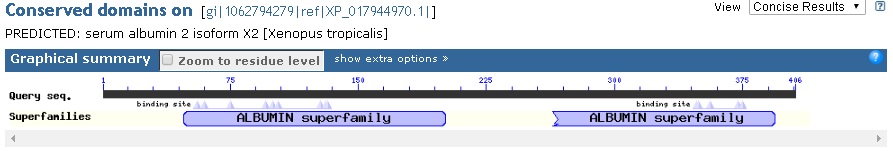
Описание: кодирует изоформы Х1 и Х2 сывороточного альбумина.
Альбумины (лат.: albus, белый) — простые растворимые в воде белки, умеренно растворимые в концентрированных растворах соли и свёртывающиеся при нагревании (денатурация белка). Их относительная молекулярная масса составляет примерно 65000, не содержат углеводов.
Общая площадь поверхности множества мелких молекул сывороточного альбумина очень велика, поэтому они особенно хорошо подходят для выполнения функции переносчиков многих транспортируемых кровью и плохо растворимых в воде веществ. К веществам, связываемым сывороточным альбумином, относятся билирубин, уробилин, жирные кислоты, соли желчных кислот и т. д.
В обоих изоформах белка присутствуют по 2 консервативных альбуминовых домена (содержит 5 или 6 внутренних дисульфидных связей).
2. Предсказание генов и белок-кодирующих областей в контиге.
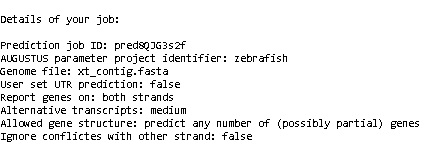
Для предсказания генов в контиге организма Xenopus tropicalis, воспользуемся помощью web-сервера AUGUSTUS.
В качестве модели был взят вид Danio rerio, будучи наиболее близким из предложенных. Параметры модели необходимы для того, чтобы получить требуемое предсказание.
Предсказание UTR (предсказание нетранслируемых участков) для выбранной модели недоступно, поэтому выбрано значение параметра false.
Мы знали о наличии нескольких альтернативно сплайсируемых генов и поэтому поставили параметр medium в соответствующем поле.
Значения остальных параметров были взяты по умолчанию.
Описание полученных данных
Последовательности белков в формате fasta, полученные после трансляции предсказанных генов
Предсказания генов в формате .gff
Предсказание генов в формате .gtf (второй версии gff)
Файл с координатами для Gbrowse
Предсказание кодирующих регионов ДНК в формате fasta
Предсказание экзонов в формате fasta
Из GenBank скачали таблицу с параметрами контига в формате gff3 (xtinfo.gff). С помощью команды
grep 'CDS' xtinfo.gff | awk '{print $4,$5,$7}' >> xtinfo.txt
из файла с особенностями был получен файл xtinfo.txt.Аналогичный файл aug.txt для предсказаний AUGUSTUS был получен командой
grep 'CDS' augustus.gff | awk '{print $4,$5,$7}' >> aug.txt.Далее скриптом из предыдущего практикума было проведено сравнение, в результате которого выяснилось, что AUGUSTUS не смог предсказать правильно ни одного гена (даже частично: по С- или N- концу).