Филогенетические деревья.
Занятие 1. Филогенетические деревья.
Для построения дерева были отобраны следующие бактерии.
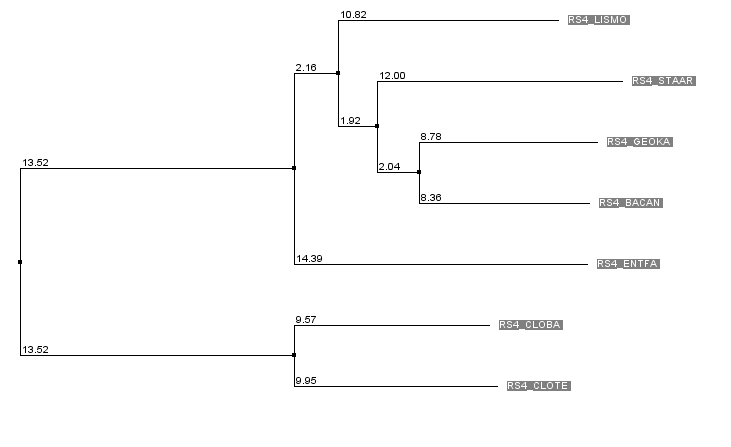
Скобочная формула дерева ((CLOTE,СLOBA),(ENTFA,((BAСAN,GEOKA,LISMO),STAAR)));
Изображение дерева
Данное дерево содержит 3 нетривиальные ветви.
-
{CLOTE,CLOBA} vs {ENTFA,BACAN,GEOKA,LISMO,STAAR}
-
{BACAN,GEOKA,LISMO,STAAR} vs {CLOTE,CLOBA,ENTFA}
-
{BACAN,GEOKA,LISMO}vs {CLOTE,CLOBA,ENTFA,STAAR}
Занятие 2. Реконструкция филогении.
-
Пользуясь таксономическим сервером NCBI https://www.ncbi.nlm.nih.gov/taxonomy/ определим, к каким таксонам относятся выбранные бактерии, а также какие нетривиальные ветви на дереве отобранных бактерий выделяют какие-нибудь из таксонов. Ниже в таблице представлена таксономия бактерий согласно серверу NCBI.
Clostridium tetani
Terrabacteria group; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
Clostridium botulinum
Terrabacteria group; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
Listeria monocytogenes serovar 1/2a
Terrabacteria group; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria; Listeria monocytogenes
Staphylococcus aureus
Terrabacteria group; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
Enterococcus faecalis
Terrabacteria group; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus
Bacillus anthracis
Terrabacteria group; Firmicutes; Bacilli; Bacillales; Bacillaceae;Bacillus
Geobacillus kaustophilus
Terrabacteria group; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus; Geobacillus thermoleovorans group
Соотнесем эти данные с данными о нетривиальных ветвях. Ветви, отделяющие определенный таксон представлены на рисунке.
- Ветвь {CLOTE,CLOBA} vs {ENTFA,BACAN,GEOKA,LISMO,STAAR} отделяет класс Clostridia от класса Bacilli
- Ветвь {BACAN,GEOKA,LISMO,STAAR} vs {CLOTE,CLOBA,ENTFA} отделяет отряд Bacillales от других таксонов

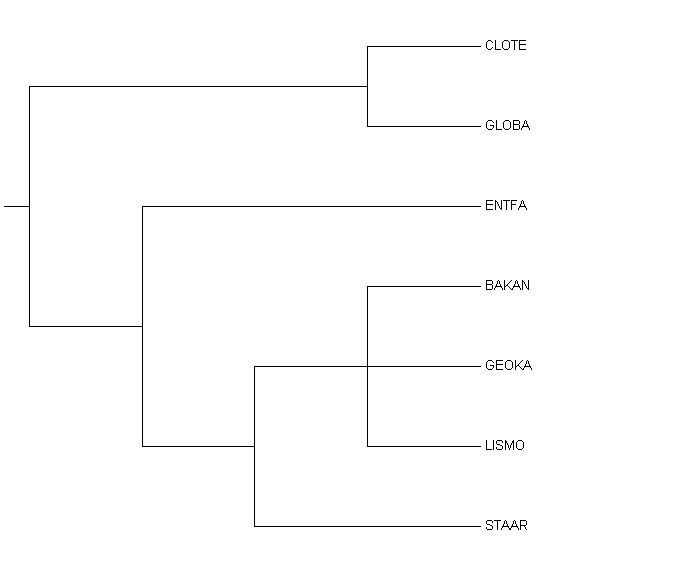
Методом "Neighbor Joining Using % Identity" было реконструировано филогенетическое дерево (ссылка на дерево в Newick-формате).
Ссылка на проект JalView
Полученное дерево открыли в программе Mega (с сохранением длин ветвей и без). Изображение дерева представлено ниже.
В данном случаем имеем четыре нетривиальные ветви:
({CLOBA,CLOTE}vs {LISMO,STAAR,BACAN,GEOKA,ENTFA})
({LISMO,STAAR,GEOKA,BACAN} vs {ENTFA,CLOBA,CLOTE})
({STAAR,GEOKA,BACAN} vs {LISMO,ENTFA,CLOBA,CLOTE})
({GEOKA,BACAN} vs { STAAR,LISMO,ENTFA,CLOBA,CLOTE})При сравнении топологий деревьев замечаем, что за исключением последней ({BACAN,GEOKA,LISMO}vs {CLOTE,CLOBA,ENTFA,STAAR}) нетривиальные ветви одинаковы; появляется новая ветвь ({STAAR,GEOKA,BACAN} vs {LISMO,ENTFA,CLOBA,CLOTE}).
.png)